小漂亮版本的单细胞小提琴图
今天是生信星球陪你的第1057天
公众号里的文章大多数需要编程基础,如果因为代码看不懂,而跟不上正文的节奏,可以来找我学习,相当于给自己一个新手保护期。我的课程都是循环开课,点进去咨询微信,随时可以报名↓ 生信分析直播课程(每月初开一期) 生信新手保护学习小组长期报名中(每月一期) 单细胞陪伴学习小组(每月一期)
0.背景
我的学生看上了一个图,问有木有代码。
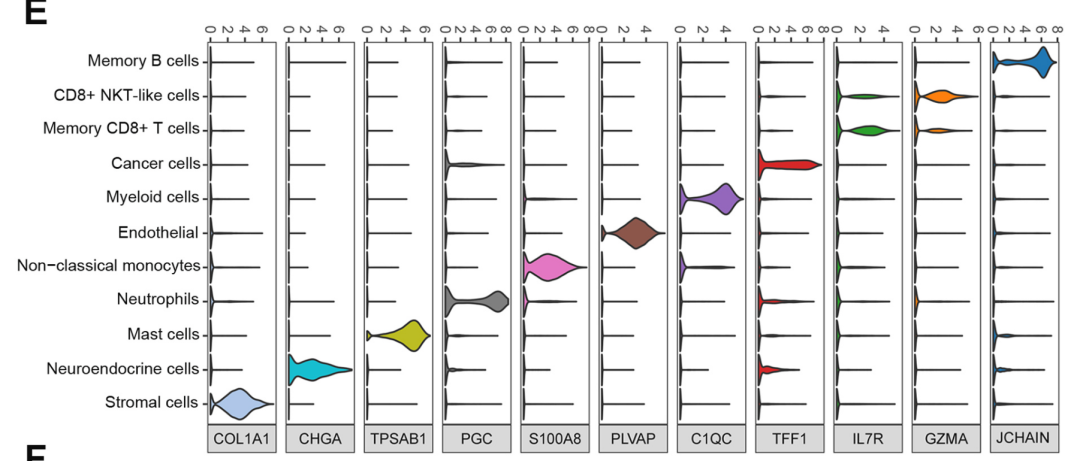
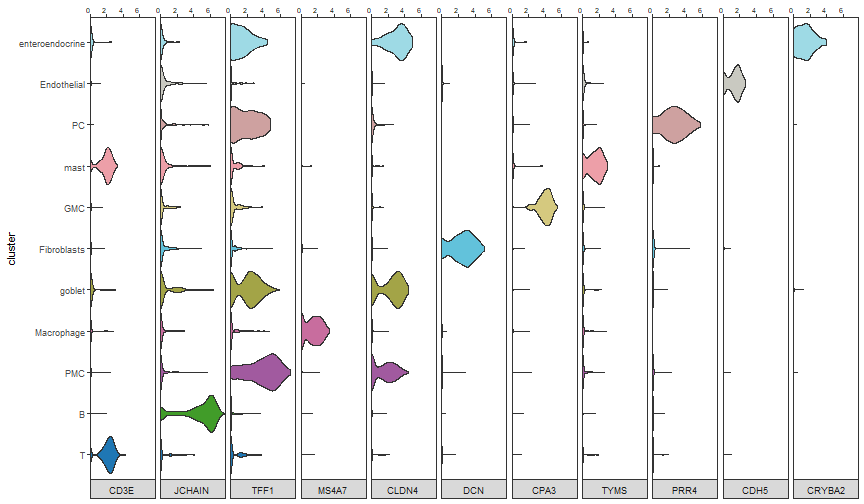
于是我甩给他之前画的图的链接:
图片
but她说她想要个一模一样的!(我怎么觉得原来这个更好看呢!)
好吧,然后我就:
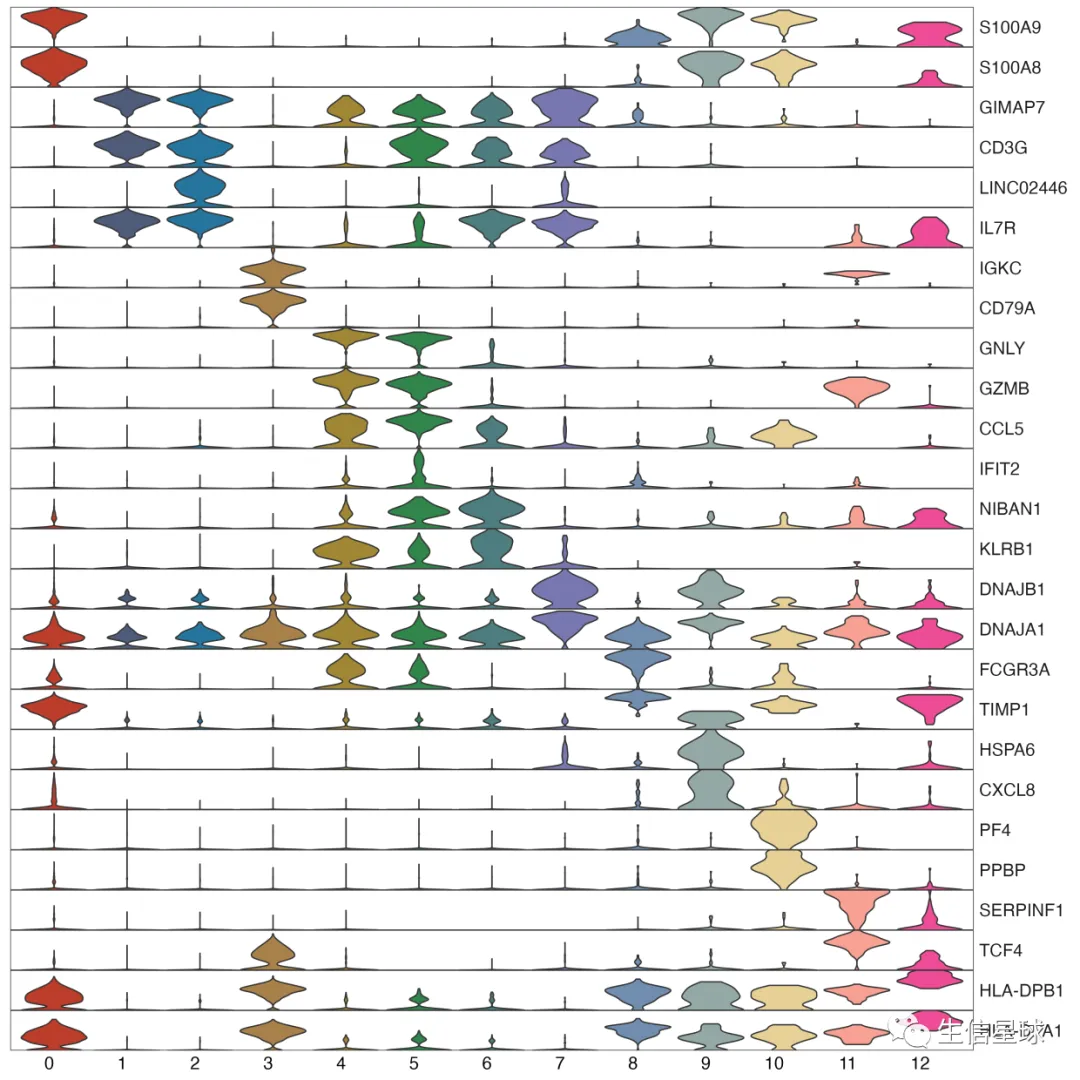
哈哈哈哈,写加排版差不多30分钟搞定。
1.输入数据
提供我的输入数据,在生信星球公众号聊天框回复"我就是命好",获得我的示例数据。其中seu.obj.Rdata是做完注释后的seurat对象,markers.Rdata是marker基因结果表格。
rm(list = ls())
load("seu.obj.Rdata")
dim(seu.obj)
## [1] 26571 4866
load("markers.Rdata")
str(allmarkers)
## 'data.frame': 19133 obs. of 7 variables:
## $ p_val : num 0 0 0 0 0 0 0 0 0 0 ...
## $ avg_log2FC: num 4.27 4.23 3.96 4.14 2.96 ...
## $ pct.1 : num 0.895 0.858 0.733 0.775 0.889 0.742 0.749 0.663 0.668 0.885 ...
## $ pct.2 : num 0.073 0.093 0.065 0.115 0.263 0.117 0.155 0.07 0.084 0.305 ...
## $ p_val_adj : num 0 0 0 0 0 0 0 0 0 0 ...
## $ cluster : Factor w/ 11 levels "T","B","PMC",..: 1 1 1 1 1 1 1 1 1 1 ...
## $ gene : chr "CD3E" "CD7" "CD3D" "CCL5" ...
2.数据整理
library(tidyverse)
#如果想要对top1基因限制一下表达比例,可以筛选一下:
# 这里按pct.1 > 0.7过滤,确保Top1基因在该cluster内具有较高“阳性细胞占比”
k = allmarkers$pct.1>0.7;table(k)
## k
## FALSE TRUE
## 16383 2750
allmarkers = allmarkers[k,]
加和不加这两句代码的区别很明显,你对比看看:

# 对每个cluster按avg_log2FC取Top1基因(marker基因越靠前,群集特异性越强)
g = allmarkers %>% group_by(cluster) %>% top_n(1,wt = avg_log2FC) %>% pull(gene) %>% unique()
# 从Seurat对象中提取Top1基因在RNA assay中的表达矩阵
m = as.matrix(seu.obj@assays$RNA$data)[g,]
# 构建长表数据框以适配ggplot2
# 步骤:转置 -> data.frame -> 添加细胞条形码与cluster -> pivot_longer -> 固定基因显示顺序
vln.df <- m %>%
t() %>%
as.data.frame()%>%
rownames_to_column("CB") %>%
mutate(cluster = seu.obj@active.ident)%>%
pivot_longer(cols = 2:(ncol(.)-1),
names_to = "gene",
values_to = "exp") %>%
mutate(gene = factor(gene,levels = g))
head(vln.df)
## # A tibble: 6 × 4
## CB cluster gene exp
## <chr> <fct> <fct> <dbl>
## 1 AAACCTGAGGAGTAGA_9 B CD3E 0
## 2 AAACCTGAGGAGTAGA_9 B JCHAIN 6.07
## 3 AAACCTGAGGAGTAGA_9 B TFF1 0
## 4 AAACCTGAGGAGTAGA_9 B MS4A7 0
## 5 AAACCTGAGGAGTAGA_9 B CLDN4 0
## 6 AAACCTGAGGAGTAGA_9 B DCN 0
3.画图
配色+大的ggplot框架+一些细节调整就可以了。
library(paletteer)
# 自定义颜色,选个调色板
my_color = paletteer_d("ggsci::category20_d3")
# 有多少个cluster就拆出多少种颜色
my_color = colorRampPalette(my_color)(length(unique(vln.df$cluster)))
p1 <- ggplot(vln.df,aes(exp,cluster))+
geom_violin(aes(fill=cluster),scale = "width")+
scale_fill_manual(values = my_color)+
facet_grid(.~gene,scales = "free_y", switch = "x")+
scale_x_continuous(expand = c(0,0),position = "top")+
theme_bw()+
theme(
panel.grid = element_blank(),
axis.title.x.top = element_blank(),
axis.text.x.top= element_text(hjust = 1,vjust = NULL,color = "black",size = 7),
legend.position = "none",
panel.spacing.y = unit(0, "cm"),
strip.text.y = element_text(angle=0,size = 14,hjust = 0),
strip.background.y = element_blank()
)
p1
本文参与 腾讯云自媒体同步曝光计划,分享自微信公众号。
原始发表:2025-10-15,如有侵权请联系 cloudcommunity@tencent.com 删除
评论
登录后参与评论
推荐阅读
目录


