空转联合单细胞分析(五):10X Visium HD上手就是复杂情况?多样本拼片如何进行拆分?
空转联合单细胞分析(五):10X Visium HD上手就是复杂情况?多样本拼片如何进行拆分?

KS科研分享与服务-TS的美梦
发布于 2025-12-20 16:57:02
发布于 2025-12-20 16:57:02
为什么会有这个问题?
一般常见到的空转都是一个sample在一个捕获区域,但是也许您会遇到或者需要进行这样的操作,在一个空转visium/visium HD捕获区域有多个组织,或者多个sample。这样做不仅能够提高提高效率,也能够有效的节约成本,同时还避免了分次处理造成的批次效应。例如这里的演示数据,就是一张片子上有8个组织,同时来源于不同的分组。文章链接:https://www.nature.com/articles/s41467-025-57926-z#data-availability。这个数据的上游分析演示见:空转上游你可以不做,但不能不会呀!详细演示Visium HD上游公共数据库分析及图像对齐矫正!
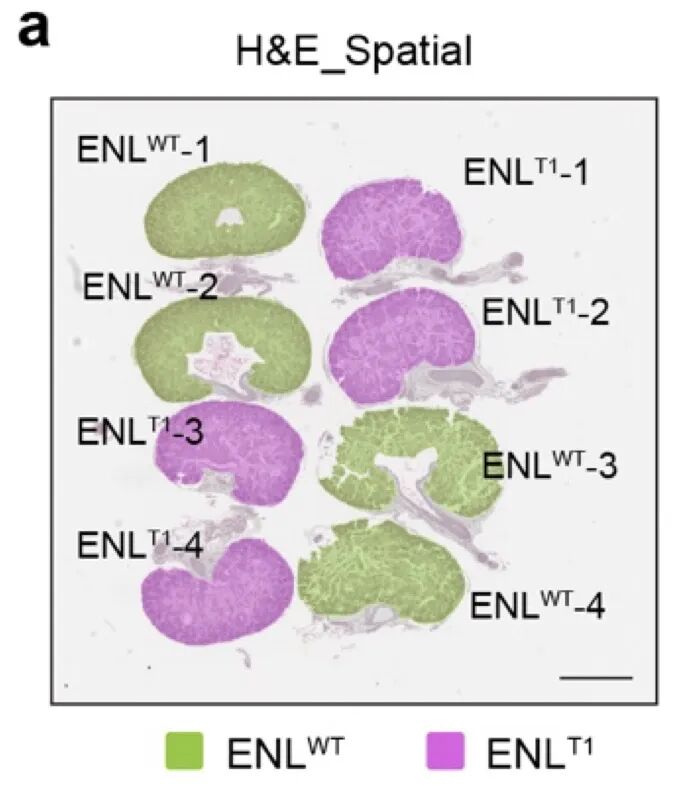
image.png
这就相当于不同的sample提前merge在一起,而一般分析都是单个片子读取,然后进行合并。在分析中,肯定是要对不同的sample进行区分的,多样本的数据如果直接读取全部的处理结果,那么混在一起无法识别具体的sample,同时,从HE染色的图上看,有些组织区域并不是我们需要分析的,是“多余”的组织,这些“碎片”组织也是需要去除的,以便后续分析。那怎么处理这种情况,先看看原文的方法描述:
To distinguish bins derived from different samples processed on the same slide capture area, bins were overlaid on the H&E microscope image for manual annotation based on sample tissue boundaries using Loupe Browser v8.0.0. At the same step, bins corresponding to debris were flagged for removal. Annotations made in the Loupe Browser were exported to comma-separated values (CSV) files, which were imported into R v4.4.0 along with the gene counts.
原文已经给出了答案。虽然在读入seurat或者其他visium处理包中之后,可以根据坐标范围确认,但是如果组织很不规则是比较麻烦的,而且目前的软件在去除‘碎片’组织的效果不是很好。所以采用10x软件Loupe Browser进行处理,不仅可以区分样本,还可以手动框选需要的分析的组织范围(比如这个示例中,我们只需要分析kidney组织,而不需要其他组织)。其实就是获取目标组织样本的barcodes。首先下载软件。
软件下载并安装:https://www.10xgenomics.com/support/software/loupe-browser/downloads
Step1:
打开软件上传文件,上传的是cloupe.cloupe文件,这是Loupe Browser的专用格式,用于可视化和交互式浏览Visium HD的空间转录组结果。spaceranger生成的结果每个分辨率文件夹都有相对应的文件,他们之间是不一样的,我们选择上传的是8um分辨率的,因为后续分析使用这个分辨率。
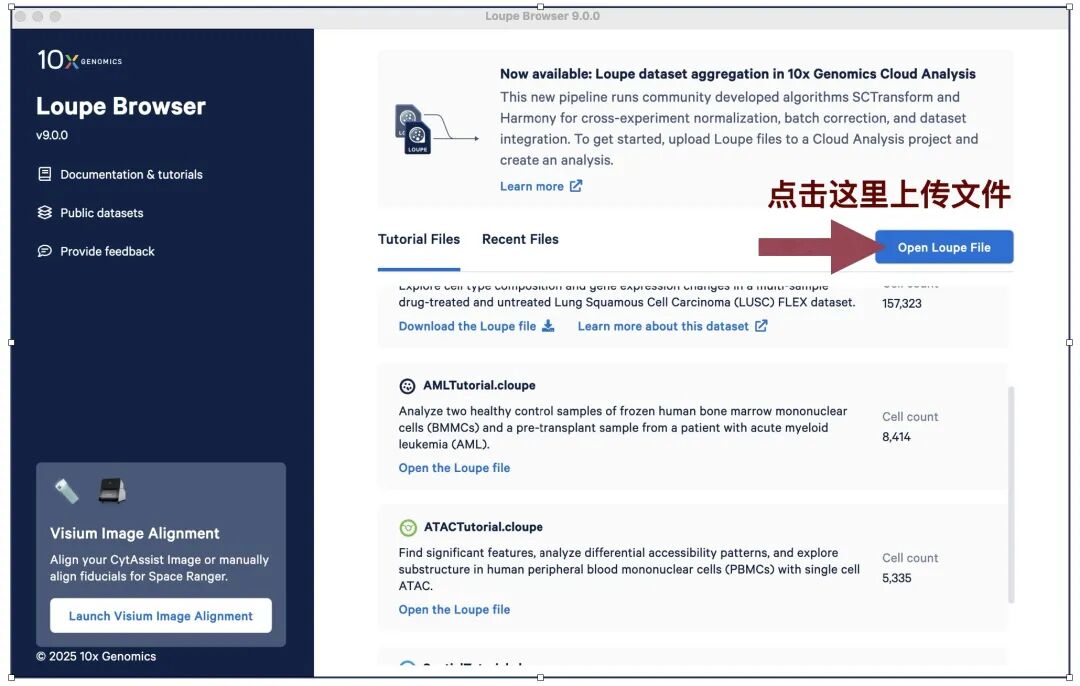
image.png
Step2:
打开之后,试图就是spaceranger扽戏结果视图,这些分析结果我们是不需要的,切换到spatial视图,放大组织区域,使用多边形工具逐个样本框选sample。框选完成,为样本命名。
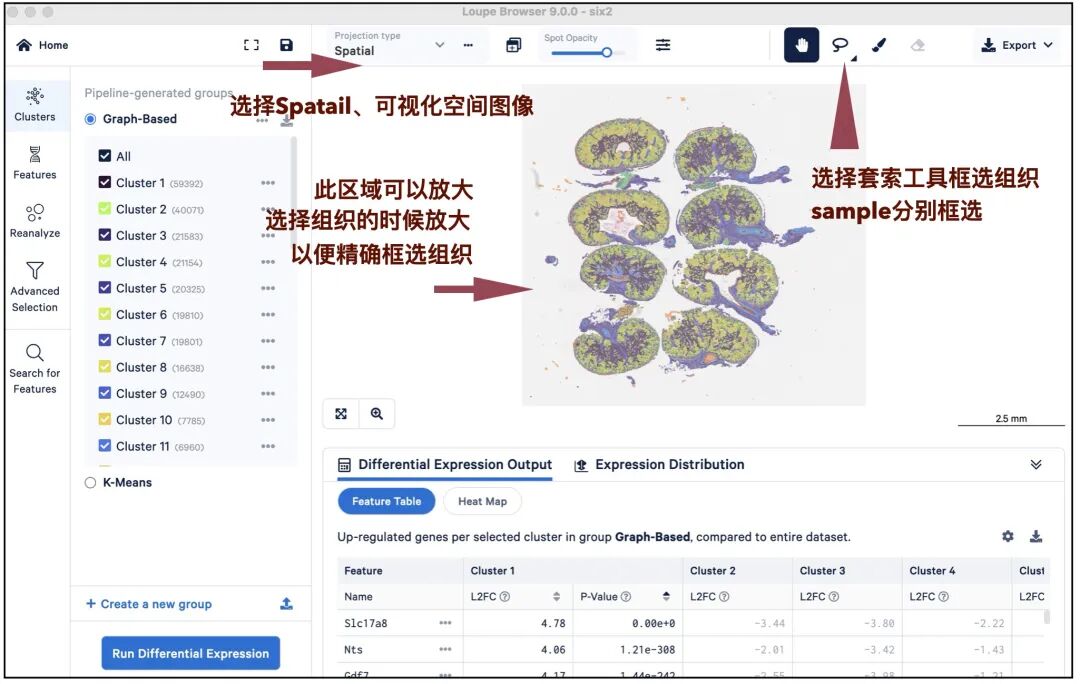
image.png
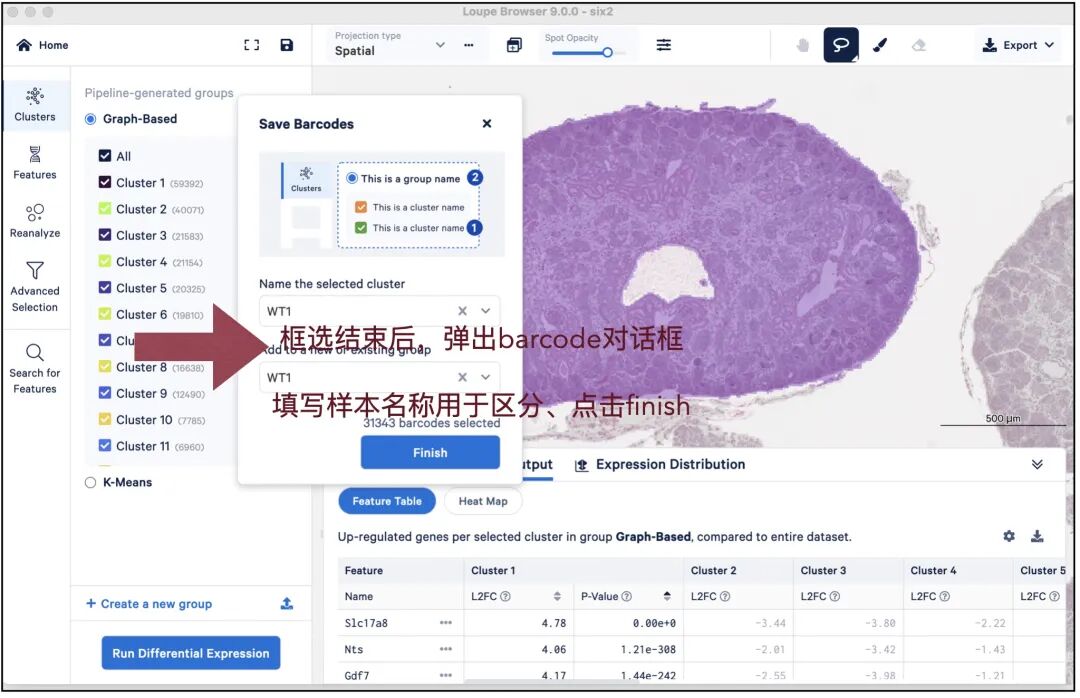
image.png
Step3:
选择完成之后下载每个sample对应的的csv文件即可!
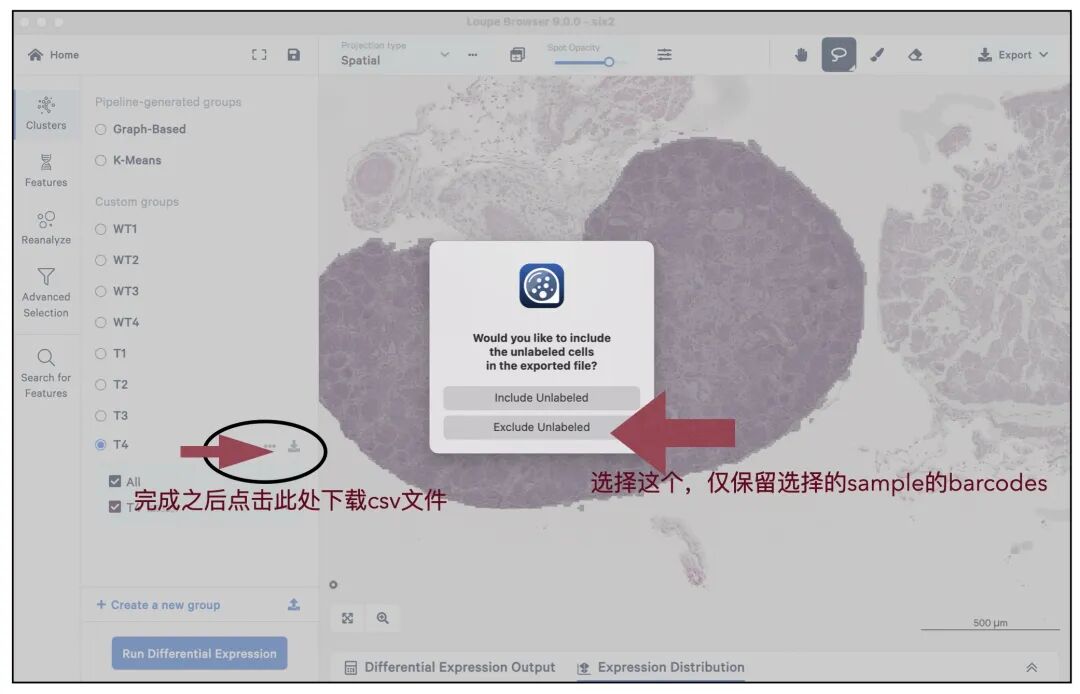
image.png
之后将这些文件读入seurat或者其他空转分析软件,就可以设别具体的样本了!
觉得分享有用的点个赞再走呗!
本文参与 腾讯云自媒体同步曝光计划,分享自微信公众号。
原始发表:2025-12-10,如有侵权请联系 cloudcommunity@tencent.com 删除
评论
登录后参与评论
推荐阅读
目录

