空转上游你可以不做,但不能不会呀!详细演示Visium HD上游公共数据库分析及图像对齐矫正
空转上游你可以不做,但不能不会呀!详细演示Visium HD上游公共数据库分析及图像对齐矫正

KS科研分享与服务-TS的美梦
发布于 2025-12-20 17:27:50
发布于 2025-12-20 17:27:50
Visium HD(High Definition)是10x Genomics 2023年推出的新一代空间转录组平台。相比传统Visium(每个spot直径约55 µm,捕获~10–20个细胞),Visium HD把空间分辨率提升到了亚细胞级别(2µm bin大小)。他们的原理类似,Visium HD相当于是升级版,数据量相应的也超级大。其实10X的上游都差不多,也比较容易运行,自己测序的数据公司已经提供了上游运行的结果,只是需要应对利用公共数据库的情况,没有提供完整的下游分析数据文件,就需要使用上游分析了。10X的上游分析使用SpaceRanger,用法类似于cellranger,只是空间转录组需要提供图片文件。以下为shell脚本,linux终端运行!运行对设备需要有一定的要求,at least 32 cores, 64GB of RAM, and 1TB of disk space。随着visium数据集不断的增加以及visium HD越来越流行,公共数据库使用是必不可少的,所以上游分析可以不做,但是一定要会,以备不时之需!
0-前言
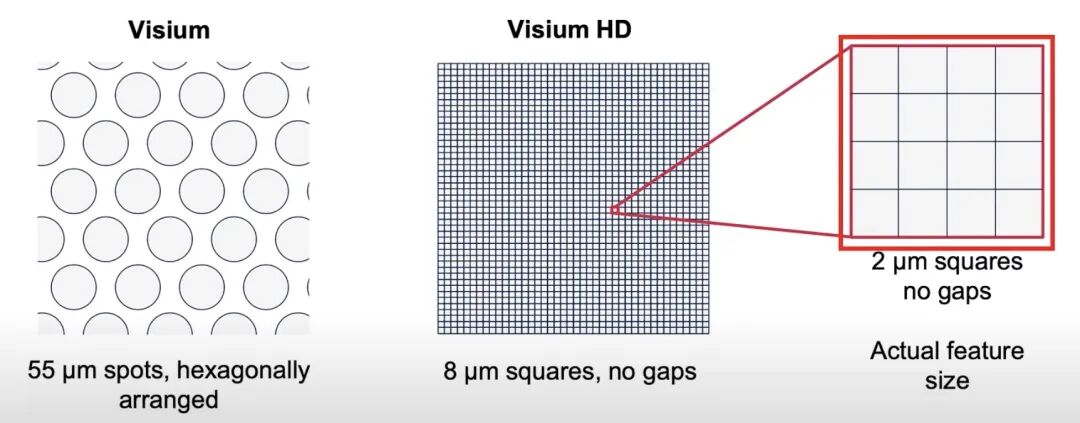
传统的visium每个spot直径约55µm,包含的细胞很多,所以一定意义上,是多个细胞的混合结果。而Visium HD把空间分辨率提升2µm,这每一个格子其实比细胞还小,10X spaceranger在分析的时候,会对2µm,8µm(4X4格子),16µm(8X8格子)进行分析,所以在最终的分析结果中,会分别包含这三种分辨率的结果,一般下游分析会选择8µm或者16µm进行分析。此外,好消息是spaceranger 4.0开始,spaceranger基于基于StarDist算法,会在上游分析中自动进行细胞分割(前提是提供需要的参数),然后在结果的segmented_outputs文件夹中呈现结果。幸运的是,紧追着seurat也推出了visium HD以及spaceranger 4.0细胞分割结果的读取以及分析方式。
3ea1310c1cc096b7131587b0aa0b1f5a.jpg
1-创建新分析环境
conda create -n spaceranger4 python==3.10
conda activate spaceranger42-download spaceranger & installation
#10X官网下载软件---我们这里安装的是最新的4.0.1,spaceranger功能更加全面和强大
#Space Ranger v3.0 及以上版本能够对 Visium HD空转(Spatial Gene Expression)数据集进行分析,
#而在v4.0中进一步新增了对 Visium HD 3′空转的支持
wget -O spaceranger-4.0.1.tar.gz "https://cf.10xgenomics.com/releases/spatial-exp/spaceranger-4.0.1.tar.gz?Expires=1758914290&Key-Pair-Id=APKAI7S6A5RYOXBWRPDA&Signature=Ocz7Nc488B11M9GAlWhvobOZwv~2RJuLYtlPaw~xlbHzkqRFBU3iZSVRs4sniAItp4wHrO9KtwG-kn~tyQmgUZ161a2ejdPPFeYpc2g67LrAGnEF5xpAkNvkBE5iNIJMLYfLsJe3FgrutHejZ6vP8l81Hui0F6UDYmbwlmGfv-NsPWtUuMHZ3Wza-SqEwGREkUJ32iSW-chC6ptghtua4CgGUp1-ZRy1QRjcpt6i4pydtNuLhMat95XXpMfyMldDjt5ZVo0le3V-9uSt-500zLmf1vpccVfXaMx9hLdVTdiK4g2QgJ2KOgoCS428MHAQ1xzJD3QcYo53tv8jb9oJfw__"
tar -zxvf spaceranger-4.0.1.tar.gz #解压
export PATH=/home/biosoft/spaceranger-4.0.1:$PATH
spaceranger --help
# spaceranger spaceranger-4.0.1
#
# Process 10x Genomics Visium data
#
# Usage: spaceranger <COMMAND>
#
# Commands:
# count Count gene expression and protein expression reads from a single capture area
# aggr Aggregate data from multiple 'spaceranger count' runs. Visium HD not supported in this release
# segment Segment nuclei from a microscope image
# mkfastq Run Illumina demultiplexer on sample sheets that contain 10x-specific sample index sets
# testrun Execute the 'count' pipeline on a small test dataset
# mat2csv Convert a feature-barcode matrix to CSV format
# mkref Prepare a reference for use with 10x analysis software. Requires a GTF and FASTA
# mkgtf Filter a GTF file by attribute prior to creating a 10x reference
# upload Upload analysis logs to 10x Genomics support
# sitecheck Collect Linux system configuration information
# telemetry Configure and inspect telemetry settings and data
# help Print this message or the help of the given subcommand(s)
#
# Options:
# -h, --help Print help
# -V, --version Print version#下载参考基因组,这里演示的数据是小鼠的
wget "https://cf.10xgenomics.com/supp/cell-exp/refdata-gex-GRCm39-2024-A.tar.gz"
tar -xf refdata-gex-GRCm39-2024-A.tar.gz
#人的参考基因组
#wget "https://cf.10xgenomics.com/supp/cell-exp/refdata-gex-GRCh38-2024-A.tar.gz"3-NCBI下载原始测序数据
#download and install sar-tool,用于下载NCBI数据
wget https://ftp-trace.ncbi.nlm.nih.gov/sra/sdk/3.2.1/sratoolkit.3.2.1-ubuntu64.tar.gz
tar -zxvf sratoolkit.3.2.1-ubuntu64.tar.gz
export PATH="$PATH:/home/tq_ziv/data_analysis/singleCell_VDJ/sratoolkit.3.2.1-ubuntu64/bin"prefetch SRR31590030 --max-size 50G#转为FSATQ
nohup fastq-dump -O ./ --gzip --split-files SRR31590030.sra &#修改文件名称
mv SRR31590030_1.fastq.gz SRR31590030__S1_L001_R1_001.fastq.gz
mv SRR31590030_2.fastq.gz SRR31590030__S1_L001_R2_001.fastq.gz4. prepare data for spaceranger pipeline
spaceranger需要的input比较多,不像scRNA那样只需要FASTQ就可以了,它还需要图片信息,所以如果原始数据(部分作者并不公开)没有图片信息,我们也无法进行分析!
图像矫正对齐的问题:
在大多数情况下,SpaceRanger会通过自动化的图像分析算法来确定芯片上的定位标记点位置;识别组织边界;将显微镜获取的组织图像(明场图片)与空间转录组表达数据及CytAssist图像进行配准(alignment)。然而,当这些步骤中的某一步出现失败或结果不理想时,就需要进行人工对齐和组织检测。一般测序公司在进行分析时候,这一步应该会完成,但是当我们自己重新分析数据,尤其是从公共数据库重新开始分析,那么这一步就显得很必要,一定要做好检查,否则得到的结果不准确或者是错误的。
对此,10X的Loupe Browser提供 Visium HD Manual Alignment工具,允许用户以交互式方式:将组织图像与载片上的定位标记点进行精确对齐;进行更准确的组织区域选择;并将结果导出为JSON文件,以便在后续的spaceranger count分析中使用。
10X官网流程:https://www.10xgenomics.com/support/software/space-ranger/latest/analysis/inputs/visium-hd-loupe-alignment
软件下载并安装:https://www.10xgenomics.com/support/software/loupe-browser/downloads 接下来是一些我实测的运行步骤:
step1:安装好后打开软件,点击左下角进入Visium手动对齐选项,并且选择合适的选项,比如我们这次演示的数据是visium HD,选择 Visium HD Manual Alignment 选项(其余的工作流程仅适用于Visium v1/v2)。然后可以看到,这个工作流程大概有10个步骤就可以完成。
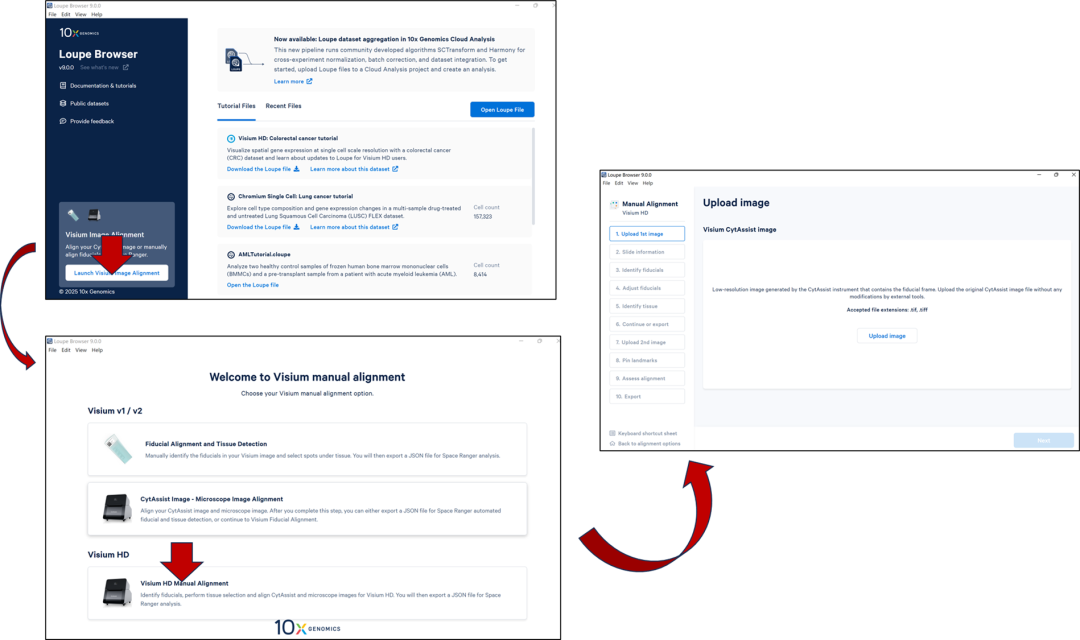
image.png
step2:接下来就是按照要求流程化的上传文件即可。需要注意,第二步需要提供slide information,这些信息可以通过读取CytAssist图像时自动获取(前提是CytAssist图像名称提供了这些信息),如果没有手动输入即可。如果实在没有,可以选择第三个选项,但是也警告了,会出现一些信息丢失。一般来说,即使公共数据库上传的数据,也会有这类信息。接下来,按照屏幕上的说明找到正方形、六边形和三角形的基准,并分别选择点击它们,如果看不清,请将图片放大操作。完成之后进行下一步,选择 Auto-refine。之后就是选择组织区域,可以使用套索工具手动选择,也可以跳过这个步骤,spaceranger会自动识别组织区域。
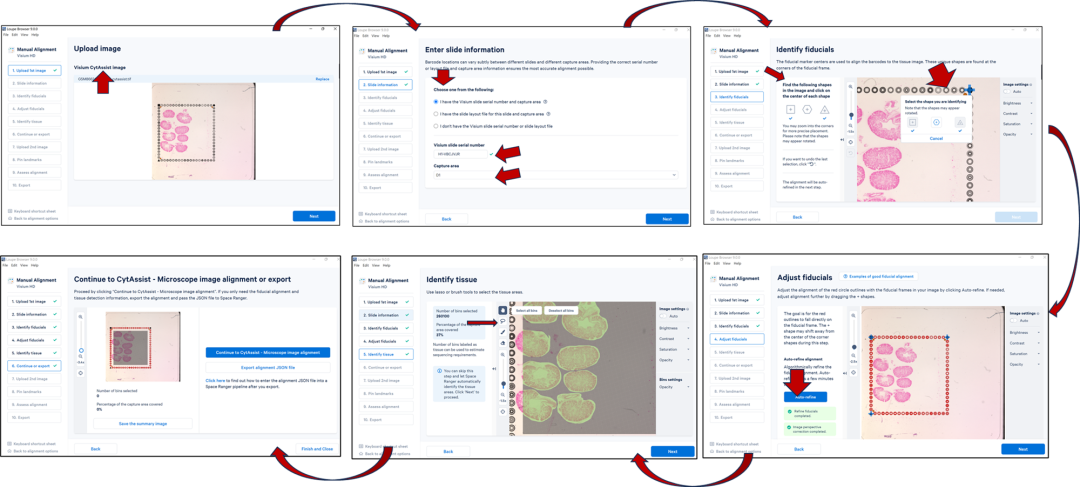
image.png
step3:完成上述步骤之后,如果没有高清显微镜图片,就可以将JSON文件导出,将其传递给SpaceRanger的--loupe-alignment参数,SpaceRanger自动将CytAssist图像与显微镜图像对齐。如果我们有明场图片,那么就直接点击到第7步,上传显微镜图片,图像类型HE选择bright-field,IF选Fluorescent。接着手动选择标记点,放大图片标记,在CytAssist图像和显微镜图片上各选择对应的点(组织边缘以及其他比较清楚的内部结构),用来获取准确的对其,选择好对应点之后,下一步点击"Auto-refine",软件自动优化对齐。完成之后,拖动右上角的滑动条,查看对其结果,你会发现,原先翻转或者镜像的两个图片对齐了。
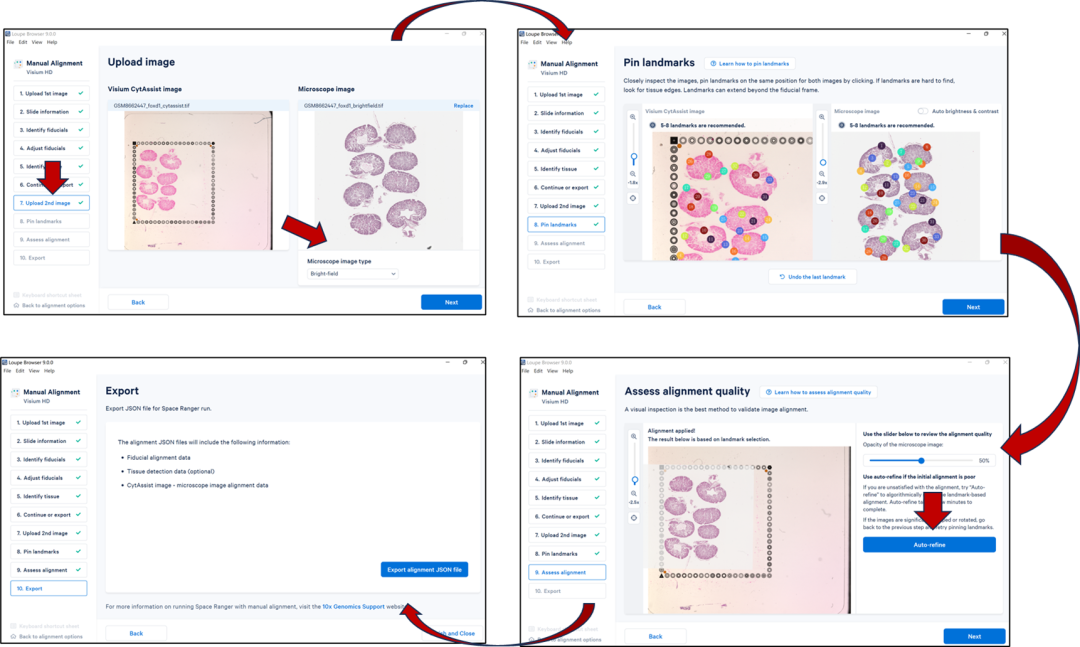
image.png
最后导出json文件:H1-VBCJVJR-D1-fiducials-image-registration.json
5-run spaceranger
注意事项:
1-Space Rangerv4.0或更高版本分析Visium HD 3′空间转录组数据时,不应指定--probe-set参数
2-SpaceRangerv3.0起,运行流程时必须显式设置--create-bam参数为 true或false。True表示创建保留bam文件。False则不需要。一般运行为了节省磁盘空间,可以设置为False,但是如果您后续需要使用BAM文件,那么设置Ture.
3-SpaceRanger v3.0起,如果可由CytAssist图像metadata自动获取,则不再强制要求指定--slide与--area参数。# 意思就是如果CytAssist图像名称类似于PVTFMN010_2023-10-31_sj0309-js-hd1-hbr-t4t7_H1-JJZY42B_D1_sample.tif, 这个名称里面已经包含了slide【H1-JJZY42B】和area【D1】,那么软件就会自动获取载玻片编号(--slide)和区域编号(--area),不再需要手动指定!
4-spaceranger count,提供image和cytaimage,会自动出细胞分割结果,结果在out文件夹segmented_outputs文件夹!
#因为运行时间较长,最好nohup放在后台
nohup\
spaceranger count --id=M_sample1 \ #sample ID,用于区分不同样本,结果储存在M_sample1文件夹
--transcriptome=/home/biosoft/refdata-gex-GRCm39-2024-A \ #参考基因组文件夹
--fastqs=/home/data_analysis/10X空间转录组/spaceranger_data/SRR31590030 \ #FASTQ文件所在文件夹
--probe-set=/home/data_analysis/10X空间转录组/spaceranger_data/Visium_Mouse_Transcriptome_Probe_Set_v2.1.0_GRCm39-2024-A.csv \ #probe-set文件
--cytaimage=/home/data_analysis/10X空间转录组/spaceranger_data/GSM8662447_foxd1_cytassist.tif \ #cytassist图片文件
--slide=H1-VBCJVJR\ #Visium载玻片的序列号。如果未知,请使用参数 --unknown-slide,但是数据质量可能受到影响
--area=D1\#Visium捕获区域标识符,例如 ‘A1’。该参数必须与--slide一同使用,除非使用了 --unknown-slide
--image=/home/data_analysis/10X空间转录组/spaceranger_data/GSM8662447_foxd1_brightfield.tif \ #明场图片文件,如果指定了cytaimage,此项为可选
--create-bam=false\ #不保存BAM文件
--localcores=16\ #线程数
#由Loupe Browser手动对齐步骤生成的对齐文件
--loupe-alignment=/home/data_analysis/10X空间转录组/spaceranger_data/H1-VBCJVJR-D1-fiducials-image-registration.json
&#看看结果文件/out/文件夹
ls -lh
# total 2.4G
# -rw-rw-r-- 1 data data 101M July 27 20:04 barcode_mappings.parquet
# drwxrwxr-x 5 data data 4.0K July 27 20:20 binned_outputs
# lrwxrwxrwx 1 data data 41 July 27 20:20 cloupe_008um.cloupe -> binned_outputs/square_008um/cloupe.cloupe
# lrwxrwxrwx 1 data data 31 July 27 20:20 cloupe_cell.cloupe -> segmented_outputs/cloupe.cloupe
# -rw-rw-r-- 1 data data 678M July 27 20:00 feature_slice.h5
# -rw-rw-r-- 1 data data 2.1K July 27 20:14 metrics_summary.csv
# -rw-rw-r-- 1 data data 1.6G July 27 19:02 molecule_info.h5
# -rw-rw-r-- 1 data data 6.6M July 27 15:29 probe_set.csv
# drwxrwxr-x 6 data data 4.0K July 27 20:20 segmented_outputs
# drwxrwxr-x 2 data data 4.0K July 27 20:20 spatial
# -rw-rw-r-- 1 data data 13M July 27 20:18 web_summary.html6-Understand outputs
barcode_mappings.parquet:此文件高效地存储空间映射信息,本质上相当于一个CSV文件。用来追踪各个barcode(小方格/bin/细胞/nuclei)之间的空间关系。
binned_outputs:这是一个文件夹,默认情况下,此目录包含三个子目录:square_002um、square_008um 和 square_016um。不同分辨率下的表达及空间文件。每个子目录都包含 filtered_feature_bc_matrix、raw_feature_bc_matrix、spatial、filtered_feature_bc_matrix.h5 和 raw_feature_bc_matrix.h5。下游分析 一般推荐选择8um.
feature_slice.h5:Visium HD 特有格式,用于高效地抓取某个基因在 2 µm 分辨率上的图像切片.
segmented_outputs:细胞分割结果文件夹,用于把空间转录组数据映射到单细胞/核层面分析。
web_summary.html:在线数据报告
------END------
本文参与 腾讯云自媒体同步曝光计划,分享自微信公众号。
原始发表:2025-11-30,如有侵权请联系 cloudcommunity@tencent.com 删除
评论
登录后参与评论
推荐阅读
目录

