超级增强子系列9: ggseqlogo进行Motif文件可视化
超级增强子系列9: ggseqlogo进行Motif文件可视化
三兔测序学社
发布于 2025-12-29 12:57:03
发布于 2025-12-29 12:57:03
前言
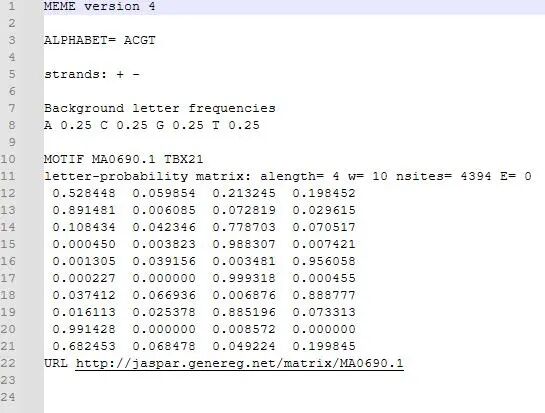
在MEME等在线motif发现工具中,输出的motif通常以比特(bits)形式表示信息含量如图1所示,反映的是每个位置相对于背景的保守性,而非碱基出现的实际频率。然而,在学术论文和序列图谱展示中,研究者普遍采用基于碱基相对频率(即概率)的motif表示方式如图2所示,以直观呈现各位置核苷酸的偏好性。为实现这一目标,我们需将通过MEME鉴定出的关键转录因子motif从不同来源下载其对应的各种格式(如PFM、PWM、MEME、JASPAR、TRANSFAC等)的motif文件。由于这些格式在结构和数值含义上存在差异,必须进行标准化处理,将其统一转换为一致的频率矩阵,从而确保下游分析与可视化结果的准确性与可比性。
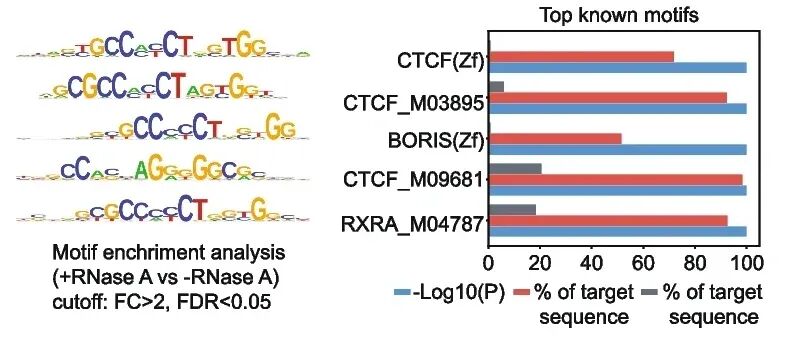
图1:每一个位置碱基按照实际频率来排列。
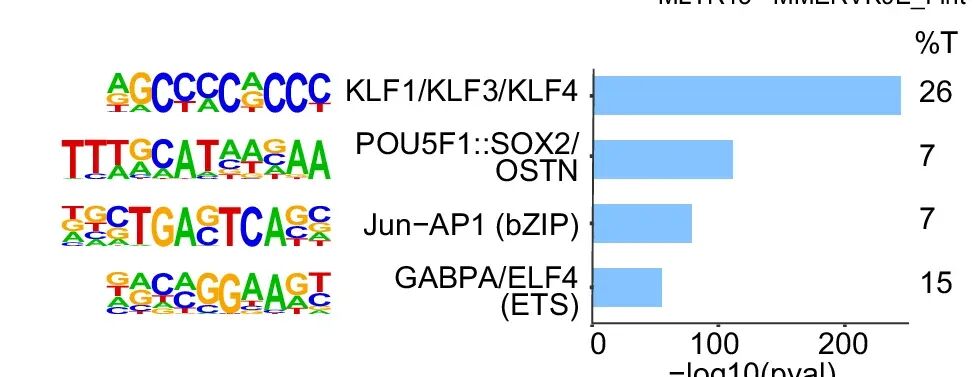
图2:Motif (每一个位置字母按照相对频率大小排列)
不同motif文件格式图
在JASPAR网址,可以看到Motif的频率矩阵存在不同格式。可以点击下载查看motif的不同格式内容。
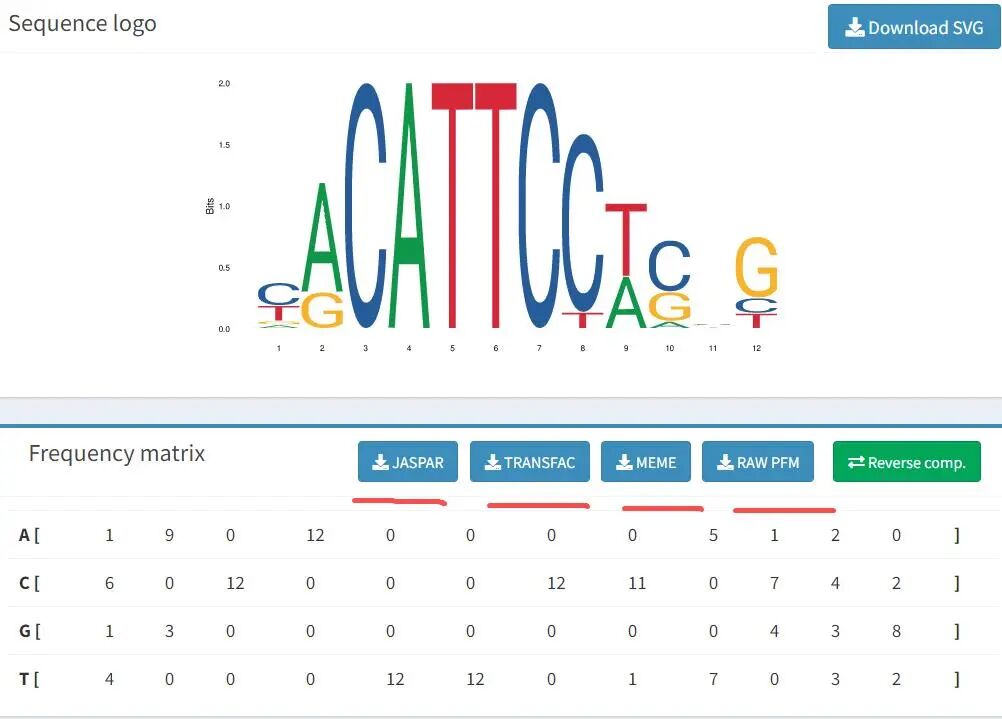
1.PWM格式

pwm格式
2.Jaspar格式
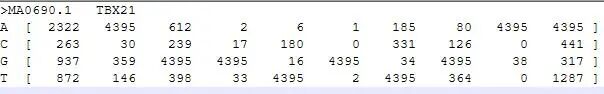
jaspar格式
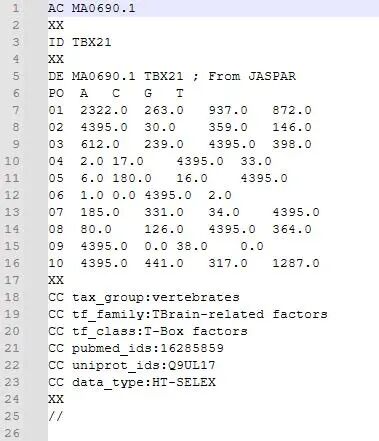
3.MEME格式
meme格式
4.PFM格式

5.TRANSFAC格式
[caption id="attachment_19895" align="aligncenter" width="443"]
比较motif格式的特点
当五种常见 motif 格式(JASPAR、MEME、PFM、PWM、TRANSFAC)的结构与内容特点对比表:
特征 / 格式 | JASPAR | MEME | raw PFM(纯文本) | PWM(带标签文本) | TRANSFAC |
|---|---|---|---|---|---|
数据类型 | 计数矩阵(PFM) | 概率矩阵(PWM) | 计数矩阵(PFM) | 概率矩阵(PWM) | 计数矩阵(PFM) |
数值形式 | 整数 | 小数(0–1,每行和为1) | 浮点数或整数 | 小数(0–1,每列和为1) | 浮点数(如 2322.0) |
碱基顺序 | A, C, G, T(每行一个碱基) | A, C, G, T(每列一个碱基) | A, C, G, T(每行一个碱基) | A:, C:, G:, T:(每行标注) | A, C, G, T(列顺序固定) |
是否含 motif ID/名称 | 是(首行以 > 开头) | 是(MOTIF 行) | 否 | 通常在首行注释 | 是(多行元数据:ID, AC, DE 等) |
是否含元信息 | 否 | 是(版本、背景频率、E值等) | 否 | 有限(可能含蛋白名) | 是(物种、家族、PubMed、实验方法等) |
结构特点 | 每行:A [ ... ] | 数据块紧跟 letter-probability matrix: | 纯数字,4 行 × L 列 | 每行以 A: 等开头 | 以 PO 定义列,位点编号(01,02…) |
典型用途 | 公共数据库存储(如 JASPAR) | MEME 套件输出,用于 logo 绘图 | 脚本中间格式 | TFBS 打分、可视化 | 商业/学术数据库(如 TRANSFAC) |
是否可直接绘图 | 否(需转概率) | 是 | 否 | 是 | 否(需提取并转换) |
说明:
- PFM(Position Frequency Matrix):记录每个位置各碱基出现次数;
- PWM(Position Weight Matrix):通常指概率形式,也可指对数似然权重,此处指概率;
- 所有格式最终可统一转换为 4×L 的概率矩阵 用于序列 logo 可视化或富集分析。
该表格有助于快速判断不同来源 motif 文件的处理方式,为标准化分析流程提供依据。
ggseqlogo批量可视化
我们先下载了TBX21 Motif 不同格式文件,如下图所示。这些文件都可以用Notepad或者Excel打开,查看内容。
我们就以这五个Motif 格式文件,通过ggseqlogo来绘图Motif。
library(ggseqlogo)
library(ggplot2)
library(dplyr)
library(purrr)
library(gridExtra)
read_motif_matrix <- function(file) {
ext <- tolower(tools::file_ext(file))
if (ext == "transfac") {
# ----------------------------
# TRANSFAC 格式
# ----------------------------
lines <- readLines(file)
pos_lines <- lines[grepl("^[[:space:]]*[0-9]+", lines)]
if (length(pos_lines) == 0) stop("No position data in TRANSFAC file: ", file)
df <- read.table(text = pos_lines, header = FALSE, sep = "", stringsAsFactors = FALSE)
if (ncol(df) < 5) stop("Insufficient columns in TRANSFAC file: ", file)
mat <- t(as.matrix(df[, 2:5])) # 转置为 4×L
rownames(mat) <- c("A", "C", "G", "T")
return(mat)
} else if (ext == "meme") {
# ----------------------------
# MEME 格式
# ----------------------------
lines <- readLines(file)
lines <- lines[!grepl("^\\s*$", lines) & !grepl("[A-Za-z]", lines)]
mat_long <- as.matrix(read.table(text = lines, sep = "", header = FALSE))
mat <- t(mat_long) # L×4 → 4×L
rownames(mat) <- c("A", "C", "G", "T")
} else if (ext == "jaspar") {
# ----------------------------
# JASPAR 格式(修正版)
# ----------------------------
lines <- readLines(file)
# 跳过第一行(header)
data_lines <- lines[2:5]
df <- read.table(text = data_lines, sep = "", header = FALSE, stringsAsFactors = FALSE)
# 第一列是碱基(A/C/G/T),其余为计数;跳过任何非数字列(如含 "[" 的列)
# 找出从第2列开始、能转为 numeric 的列
numeric_cols <- sapply(df[-1], function(x) {
!any(is.na(suppressWarnings(as.numeric(as.character(x)))))
})
if (!all(numeric_cols)) {
# 如果有非数字列(如 "["),只保留能转 numeric 的列
count_cols <- which(numeric_cols) + 1 # +1 因为 df[-1]
} else {
count_cols <- 2:ncol(df)
}
mat <- as.matrix(df[, count_cols])
rownames(mat) <- c("A", "C", "G", "T")
} else if (ext == "pfm") {
# ----------------------------
# PFM 格式(原始)
# ----------------------------
mat <- as.matrix(read.table(file, skip = 1, fill = TRUE))
if (nrow(mat) != 4) {
mat <- as.matrix(read.table(file, fill = TRUE))
}
if (nrow(mat) != 4) stop("PFM file must result in 4 rows (A,C,G,T): ", file)
rownames(mat) <- c("A", "C", "G", "T")
} else if (ext == "txt") {
# ----------------------------
# TXT 格式(PWM,带 A:/C: 行名)
# ----------------------------
mat_df <- read.table(
file,
skip = 1,
row.names = 1,
sep = "\t",
check.names = FALSE,
stringsAsFactors = FALSE,
fill = TRUE
)
mat <- as.matrix(mat_df)
cleaned_rownames <- gsub("[^ACGT]", "", rownames(mat))
expected <- c("A", "C", "G", "T")
if (!setequal(cleaned_rownames, expected)) {
stop("File ", file, " missing valid rows. Got: ", paste(cleaned_rownames, collapse = ", "))
}
rownames(mat) <- cleaned_rownames
mat <- mat[expected, , drop = FALSE]
} else {
stop("Unsupported file extension: '", ext, "' for file: ", basename(file))
}
if (any(is.na(mat))) warning("NA values introduced in matrix from: ", file)
return(mat)
}
# 匹配所有支持的格式
all_files <- list.files(
pattern = "\\.(pfm|txt|transfac|meme|jaspar)$",
full.names = TRUE,
ignore.case = TRUE
)
# 批量读取
motif_names <- tools::file_path_sans_ext(basename(all_files))
motif_list <- map(all_files, read_motif_matrix)
# 可视化
plot_list <- map2(motif_list, motif_names, ~{
ggseqlogo(.x, method = "prob") +
theme_classic() +
labs(title = .y, x = "Position", y = "Probability") +
theme(plot.title = element_text(size = 9, face = "bold"),
axis.text = element_text(size = 7))
})
# 5. 创建PDF文件
pdf("all_motifs_col.pdf", width = 8.5, height = 11)
# 每页2列,每列6个图(共12个图)
ncol <- 2
nrow <- 6
plots_per_page <- ncol * nrow
# 分页输出
for (i in seq(1, length(plot_list), by = plots_per_page)) {
end_idx <- min(i + plots_per_page - 1, length(plot_list))
# 获取当前页的图
page_plots <- plot_list[i:end_idx]
# 如果最后一页图不够,用空白填充
if (length(page_plots) < plots_per_page) {
blank_count <- plots_per_page - length(page_plots)
for (j in 1:blank_count) {
page_plots[[length(page_plots) + 1]] <- ggplot() + theme_void()
}
}
# 排列当前页
grid.arrange(
grobs = page_plots,
ncol = ncol,
nrow = nrow,
top = paste("Page", ceiling(i/plots_per_page),
"- Showing motifs", i, "to", end_idx)
)
}
dev.off()
cat("✓ 已创建 all_motifs_col.pdf\n")
cat("✓ 包含", length(plot_list), "个motif\n")
cat("✓ 布局: 每页", ncol, "列 ×", nrow, "行 =", plots_per_page, "个motif\n") 输出图片如下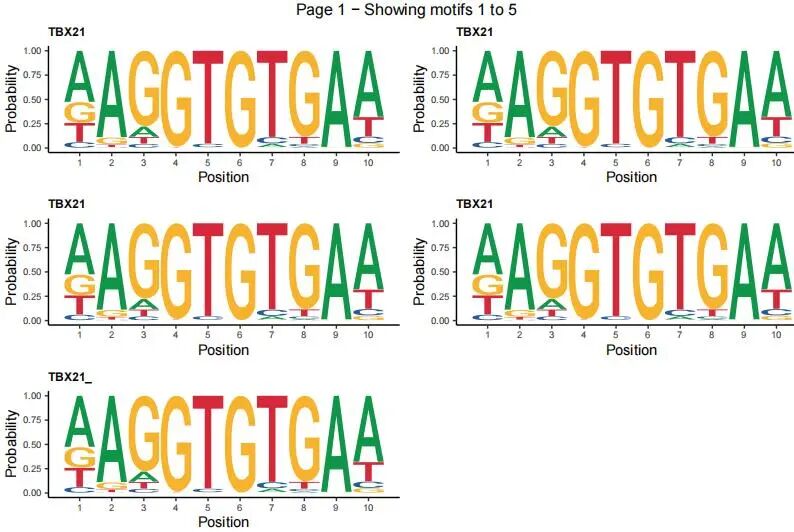
图1来源:Hyle, J., Qi, W., Djekidel, M. N., ..., Li, C. (2025) Deciphering the role of RNA in regulating CTCF’s DNA binding affinity in leukemia cells. Genome Biology 26(1). https://doi.org/10.1186/s13059-025-03582-x
图2来源:Trovato, M., Bunina, D., Yildiz, U., ..., Noh, K. (2024) Histone H3.3 lysine 9 and 27 control repressive chromatin at cryptic enhancers and bivalent promoters. Nature Communications 15(1). https://doi.org/10.1038/s41467-024-51785-w
🔬 科研不止于工具,更在于思路。选择正确的工具,才能让数据说话。
【超级增强子系列文章】
超级增强子系列1:super enhancer鉴定-ROSE软件的安装与使用
超级增强子系列2:ROSE准备gff文件:peak 信息文件转化为9列gff格式文件R代码
超级增强子系列3:R语言批量处理ROSE文件生成SE与TE.bed文件
超级增强子系列5:用ChIPseeker进行超级增强子基因注释
超级增强子系列6:GREAT-基因组调控元件专业注释富集工具
超级增强子系列7: 用MEME进行超级增强子转录因子motif 富集分析实战
超级增强子系列8: motif 富集分析工具XSTREME输出文件解释
如果你觉得这篇博文对你有帮助,请点赞、收藏、转发!支持我们持续输出优质内容!
关注“三兔测序学社”,获取更多实用教程与前沿解读。
本文参与 腾讯云自媒体同步曝光计划,分享自微信公众号。
原始发表:2025-12-17,如有侵权请联系 cloudcommunity@tencent.com 删除
评论
登录后参与评论
推荐阅读
目录

