AutoTax | 基于全长 16S 测序数据创建特定环境的菌群注释数据库
AutoTax | 基于全长 16S 测序数据创建特定环境的菌群注释数据库
16S rRNA 扩增子测序是研究微生物群落多样性和动态变化的重要方法。然而,目前公共 16S rRNA 参考数据库中仍缺乏许多环境微生物的高同一性参考序列,也缺乏针对大多数未培养微生物的系统分类注释。
随着高通量全长 16S rRNA 基因测序方法的发展,我们可以快速生成覆盖真实多样性的高分类水平的 16s 全长数据库。然而,单靠提高序列覆盖率并不能解决许多未培养分类群的分类注释不佳的问题。
本文介绍的 AutoTax 工作流程正是为创建涵盖所有七个分类等级的分类注释提供了一种简单有效的策略,不但可以注释上已知物种,还能为未确定的物种分配一个分类名称。AutoTax 使用 SILVA 分类法作为主干,同时基于序列的从头聚类结果为未分类的分类群提供特定占位符名称。
Dueholm, M. S., Andersen, K. S., McIlroy, S. J., Kristensen, J. M., Yashiro, E., Karst, S. M., ... & Nielsen, P. H. (2020). Generation of comprehensive ecosystem-specific reference databases with species-level resolution by high-throughput full-length 16s rRNA gene sequencing and automated taxonomy assignment (Autotax). MBio, 11(5), e01557-20.
有了为特定环境注释的微生物注释数据库,我们就能直接结合使用一些分类器对包括未知物种在内的数据进行注释。比如 SINTAX 或 q2-feature-classifier。也有研究表明,特定于环境的分类丰度信息可以用作此类分类器的权重,可以进一步提高分类分配的准确性。这也意味着用于生成注释数据库的全长 16S rRNA 频率可用作特定生态系统的系统发育信息权重(参见 qiime2 q2-clawback 插件)。
AutoTax 注释框架
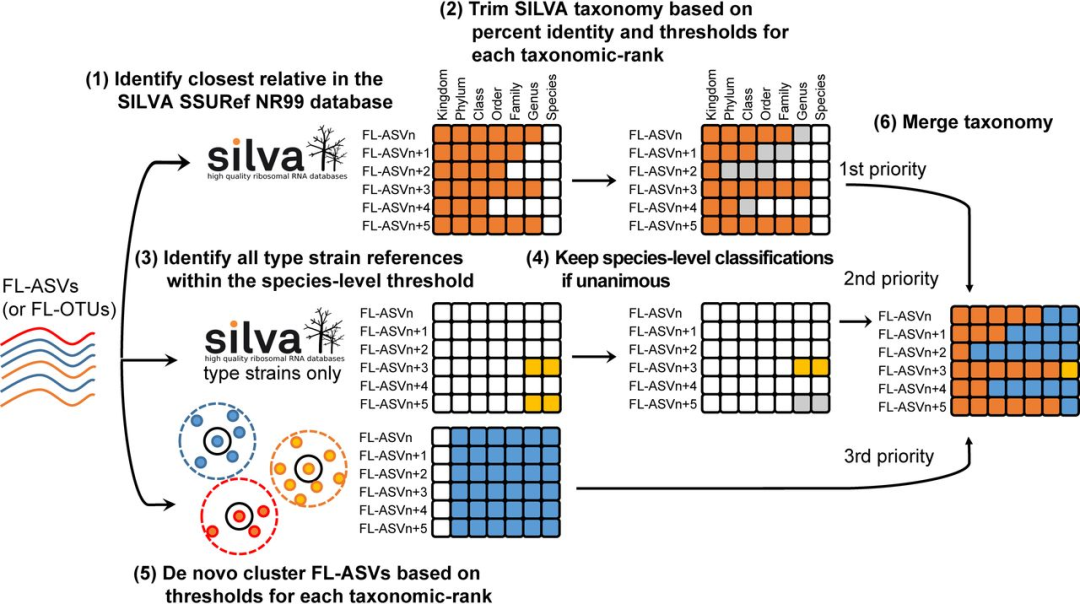
图中彩色方块表示 FL-ASV 的分类注释来源,分别如下:橙色来自SILVA SSURef NR99;黄色来自 SILVA 模式菌株;蓝色为 de novo 命名;灰色为在 AutoTax 流程中被拒绝的命名。
流程步骤:
- FL-ASV (全长 ASV 序列)首先与 SILVA 138 SSURef NR99 数据库进行比对,识别最邻近的物种并计算序列同一性;
- 根据序列同一性以及对应分类阈值,对上一步的比对结果进行过滤;

- 为了获得物种水平的信息,FL-ASVs 也被映射到从 SILVA 数据库中提取的模式菌株的序列;
- 如果序列同一性 >98.7% 并且只有一个物种,则采用该物种名注释;如果 FL-ASV 匹配到不止一个物种,则误分类的风险很高,流程不会在物种级别对其进行分类;
- 同时,FL-ASV 对应不同阈值以不同分类等级进行聚类。用于生成稳定的 de novo 分类注释信息。
- 最后,用 de novo 注释信息填补基于 SILVA 的分类注释中的空白,以获得完整的注释。
由于 SILVA 的分类注释并未对所有序列提供完整的七级分类注释,因此缺失的分类由 de novo 占位符分类注释进行填充。该分类是基于 FL-ASV 在对应于每个分类等级阈值的聚类结果创建的。cluster 以 denovo_x_y 格式进行标记,其中 x 是分类等级(k、p、c、o、f、g 和 s)的一个字母缩写,y 表示核心 FL-ASV 的编号。
基于 SILVA 和 de novo 的分类注释的合并可能会导致一些冲突,例如,来自同一物种的不同 FL-ASV 与多个属相关联。在这种情况下,核心 FL-ASV 的属级分类适用于该物种范围内的所有 FL-ASV。这些类型的冲突仅适用于较低级别的分类群(种)恰好位于接近较高级别的分类群(属)的分类阈值时,这种情况也比较少见(约 1 %)。
AutoTax 程序的详细说明
生成全长 16S rRNA ASV
全长 16S rRNA 基因序列首先根据 SILVA 138 SSURef Nr99 数据库 使用 usearch -orient 命令固定序列方向。然后使用 usearch -fastx_uniques -sizeout -strand plus -threads 1 命令对序列进行去重。其中 -sizeout 参数会在 FASTA 标头中加上 size 注释,即观察到唯一序列的次数。-strand plus 参数确保在识别相同序列时只考虑方向正确的序列。同时,根据 size 注释对去重序列进行排序和编号。-threads 1 参数则确保相同 size 的序列在输出中始终以相同的方式排序和编号。最后使用 usearch -unoise3 -minsize 2 命令对去重的序列进行降噪以生成 FL-ASV。-minsize 参数指定检测 ASV 所需的最小丰度。一般来说,短读长扩增子的默认值为 8,但由于合成的长读长序列是独立扩增的(在 PCR 步骤之前添加了唯一分子标识符 (UMI)),错误率非常低,所以这里可以将阈值降低到 2。
对 FL-OTUs 进行嵌合体过滤
使用 usearch -cluster_smallmem -id 0.99 -maxrejects 0 -centroids -sortedby size 命令,将上面的去重序列以 99% 的序列同一性聚类,创建 FL-OTUs。-maxrejects 0 参数将指定完整的数据库搜索,从而提供更可靠的聚类。使用 usearch -uchime2_ref -strand plus -mode sensitive -chimeras 命令,使用 FL-ASVs 作为参考数据库来识别并提取潜在的嵌合体。这里选择 sensitive 模式是因为它能捕获更多的嵌合体(以更多的误报为代价)。最终使用 usearch -search_exact -strand plus -dbnotmatched命令创建嵌合体过滤后的 FL-OTU,这里使用识别到的嵌合体序列作为查询序列,并使用预过滤得到的 FL-OTU 作为参考数据库。
分类学注释
对于分类学注释,该流程首先创建了两个独立的分类学方法。第一种分类法基于最新版本的 SILVA SSURef Nr99 数据库,反映了微生物分类法的当前状态。第二种则是一种稳健的 de novo 分类法,虽然在进化上不一定正确。后者将用作分类等级的分类占位符,而无需基于 SILVA 的分类法中的信息。
这两种分类方式第一步都是使用 SINA 将每个 FL-ASV 与 ARB 格式的 SILVA 参考数据库进行全局比对。接着使用 Linux 命令 awk 将对齐的序列修剪到全局 SILVA 对齐中的 1048 到 41788 碱基位置。这样的修整主要目的有两个。一是确保当 FL-ASV 比对到使用常用的 27F 和 1391R 引物生成的 SILVA 数据库中的参考序列时,侧翼序列不会导致人为的低同一性。其次,这可以似 de novo 聚类更为稳健。修剪后,使用 usearch -fasta_stripgaps 命令从 FL-ASV 的对齐中删除 gap。最后,在 R 中根据 FL-ASV 编号对 FL-ASV 进行排序。
通过将每个修剪过的 FL-ASV 映射到 FASTA 格式的 SILVA SSURef Nr99 和模式株数据库,从最接近的亲属以及同一性百分比来注释基于 SILVA 的分类信息。使用 usearch -usearch_global -maxrejects 0 -maxaccepts 0 -top_hit_only -strand plus -id 0 -blast6out 命令比对到完整的 SILVA 数据库,使用 -maxrejects 0 -maxaccepts 0 -strand plus -id 0.987 -blast6out参数比对到模式株数据库。-maxrejects 0 -maxaccepts 0 参数确保执行全面检索。-top_hits_only -id 0 参数可提供完整 SILVA 数据库中的最佳匹配,而没有 -top_hit_only 参数的 -id 0.987 则会提供物种级别阈值内的所有匹配。接下来将 SILVA 比对的输出文件加载到 R 中,并创建一个数据框,其中包含 FL-ASV 编号、同一性百分比和最近亲属的 SILVA 分类法的列。分类学注释会进一步被拆分为从界到种的七个主要分类学等级。然后进行分类学注释的过滤,包含以下任何单词的字段将被清除:“uncultured”、“unknown”、“unidentified”、“incertae sedis”、“metagenome”、“bacterium”和“possible”。此外,还会把所有“candidatus”替换为“Ca”,把所有空格替换为下划线。最后,所有字符除字母、数字和句号、破折号和下划线之外将被删除。
当然,从 SILVA 数据库中最接近的亲属获得的分类法不一定与 FL-ASV 的分类法相匹配。因此,我们还需根据 FL-ASV 与其最亲缘关系之间的同一性百分比来过滤分类注释。这里使用了 Yarza 等人提出的分类阈值。(属级 94.5%,科级 86.5%,目级 82.0%,纲级 78.5%,门级 75.0%)。物种级别的分类则根据与模式菌株的比对结果获得,如果多个物种的参考序列都在物种级别阈值内,则不提供分类注释。
为了生成全面的 de novo 分类注释,FL-ASV 使用 usearch -cluster_smallmem -id x -maxrejects 0 -uc -sortedby other 命令,根据 Yarza 等人提出的分类阈值进行聚类。这里 x 代表给定分类等级的阈值。-sortedby other 参数将根据 FL-ASV 在输入 FASTA 文件中出现的时间对它们进行聚类,所以即使将来有额外的 FL-ASV 附加到 FL-ASV 数据库,也会形成相同的聚类结果。输出文件是一个 UCLUST 格式的文本。
六个 UCLUST 输出文件(种到门级)被加载到 R 中,每个文件都被转换成一个包含两列的数据框。带有聚类信息的第一列根据分类聚类等级命名,带有输入序列的第二列将命名为下面的分类等级。随后,数据框从种到门级进行合并。以此产生一个更全面的分类注释,其中聚类的质心进一步根据上述分类等级的从属关系进行确定。
最后,用 denovo 分类信息替换 SILVA 注释中的空字段,以得到最终的分类注释。两个分类注释的合并也可能会导致一个分类单元有多个父分类的情况(例如,来自同一物种的序列可能附属于多个属)。在这些情况下,分类群中具有最低 ASV 编号的 FL-ASV 的分类将被分配给所有成员。日志文件中会写入所有此类情况的详细信息。
格式化的参考数据库,可直接用于使用 SINTAX 或 QIIME 2 框架中的分类器进行分类注释。
使用 AutoTax 对 FL-ASV 进行分类注释
Github:https://github.com/KasperSkytte/AutoTax
软件依赖
- usearch (version 10 or later)
- SINA (version 1.6 or later)
- R (version 3.5 or later) with the following packages installed (the script will attempt to install if missing):
- Biostrings (from Bioconductor through
BiocManager::install()) - doParallel
- stringr (and stringi)
- data.table
- tidyr
- dplyr
- Biostrings (from Bioconductor through
推荐使用 docker 安装
直接从 docker hub 下载:
docker pull kasperskytte/autotax:latest
从 dockerfile 生成:
git clone https://github.com/KasperSkytte/AutoTax.git
cd AutoTax
docker build -t kasperskytte/autotax:latest .
镜像中已经包含了最新的 github 仓库内容,位于 /opt/autotax/。运行AutoTax 容器,并将当前工作目录挂载在容器内部 /AutoTax 路径下:
docker run -it --rm --name autotax -v ${PWD}:/autotax kasperskytte/autotax:latest -h
同样也可以用 singularity:
singularity run --bind ${PWD}:/autotax docker://kasperskytte/autotax:latest -h
使用 docker 的 -e 参数或者 singularity 的 --env 参数可以轻松调整 AutoTax.Bash 脚本开始时的各种设置参数,比如 -e denovo_prefix="midas" -e denoise_minsize=4。
准备数据库文件
除了这些软件工具之外,还需要 UDB 和 ARB 格式的 SILVA 和 SILVA 模式株数据库文件。所有 4 个文件都可以在 figshare 上下载(SILVA 版本 132 和 138)。运行脚本前需在 autotax.bash 脚本中正确设置了些文件的路径。当然也可以使用其他数据库。
对于 SILVA 版本 138,这可以通过以下命令下载:
wget https://ndownloader.figshare.com/files/22790396 -O SILVA138_NR99.zip
unzip SILVA138_NR99.zip -d refdatabases/
也能直接通过 getsilvadb.sh 脚本下载。
$ singularity exec --bind ${PWD}:/autotax docker://kasperskytte/autotax:latest getsilvadb.sh -h
INFO: Using cached SIF image
This script downloads a desired release version of the SILVA database and makes it ready for AutoTax.
Version: 1.0
Options:
-h Display this help text and exit.
-r (required) The desired SILVA release version, fx "138.1".
-o Output folder. (Default: refdatabases/)
-t Max number of threads to use. (Default: all available except 2)
-v Print version and exit.
使用方法
$ bash autotax.bash -h
Pipeline for extracting Full-length 16S rRNA Amplicon Sequence Variants (FL-ASVs) from full length 16S rRNA gene DNA sequences and generating de novo taxonomy
Version: 1.6.1
Options:
-h Display this help text and exit.
-i Input FASTA file with full length DNA sequences to process (required).
-c Cluster the resulting FL-ASVs at 99% (before generating de novo taxonomy),
do chimera filtering on the clusters, and then add them on top in the same way as when using -d.
-d FASTA file with previously processed FL-ASV sequences.
FL-ASVs generated from the input sequences will then be appended to this and de novo taxonomy is rerun.
-t Maximum number of threads to use. Default is all available cores except 2.
-b Run all BATS unit tests to assure everything is working as intended (requires git).
-v Print version and exit.
可使用 Github 仓库中包含的示例数据进行测试 /test/example_data/:bash autotax.bash -i test/example_data/10k_fSSUs.fa -t 20。可在 output/ 文件夹中看到所有输出结果,中间文件位于 temp/。
一些注意点
流程依赖的 usearch 并非免费,所以不包含在 docker 镜像中。需要先购买或使用免费的 32 位版本,并将可执行文件放在安装在容器内的同一文件夹中,并将其命名为 usearch11。
AutoTax 也提供了包含免费的 vsearch 镜像,可直接在 docker hub 上下载 tag 带 _vsearch 后缀的镜像。
docker pull kasperskytte/autotax:1.5.4_vsearch
通过容器运行时,所有路径都必须为相对工作目录。
通过 docker 运行时需调整数据库文件的路径,需在启动容器时调整变量 silva_db、silva_udb 和 typestrains_udb(例如 --env silva_db="refdatabases/SILVA_138_SSURef_NR99_tax_silva.udb")。denovo_prefix 变量也是如此。

