BIB | “DSN-DDI”实现药物间相互作用预测性能的突破!
编译 | 杨慧丹 审稿 | 李梓盟
近日,由微软研究院科学智能中心研究团队与湖南大学DrugAI团队共同研究的一项最新成果“DSN-DDI:an accurate and generalized framework for drug-drug interaction prediction by dual-view representation learning”在Briefings in Bioinformatics期刊(中科院1区top期刊)发表。

论文要点
- 如何充分利用药物表示和药物对表示是子结构提取和DDI预测的关键。
- 本文提出DSN-DDI算法,该算法采用局部和全局表示学习模块,同时从单一药物(“intra-view”)和药物对(“inter-view”)这两个视图中学习药物的子结构。
- 综合评估表明,DSN-DDI在transductive setting和inductive setting方面均达到了最先进的性能,并显示了其在实际DDI应用中的实用性。
- DSN-DDI具有良好的可移植性,可作为药物发现领域的一个通用框架。
研究背景
临床治疗中,联合用药具有显著的治疗效果,但会增加由药物组合的理化不相容引起不良副作用的风险。药物-药物相互作用(DDI)的识别仍然是一项具有挑战性的任务,因为大量的药物对数据导致药物研究和临床试验的成本非常高昂且效率较低。目前已开发了许多关于DDI预测的计算方法,这些方法大多遵循一种假设,即具有相似特征的药物可能具有相似的相互作用。为了充分利用药物的原始特征,最近的工作主要集中在利用深度神经网络强大的特征提取能力上。
考虑到一种药物可以分成几个官能团或化学子结构,共同产生整体药理特性。一些研究尝试将药物细分为亚结构用于DDI预测。然而,这些方法使用的子结构仅从单一药物的隐藏表征中学习,忽略了两种药物之间的交互可以为子结构提供更多有价值的信息。此外,在大多数DDI预测模型中,药物表示学习是GNN信息传递模块中一个单视图过程,只对药物本身的信息进行编码。也有一些工作尝试将双视图表示学习引入,但主要关注的还是整个分子的全局表征学习,其实可以进一步用于药物分子中原子的局部表征学习。具体来说,可以采用双视图学习来更新GNN block中的每个节点表示,并在原子水平上集成双视图信息。
研究方法
DSN-DDI是一个图神经网络(GNN),它迭代使用局部与全局表示学习模块,同时从intra-view和inter-view二个视角学习药物子结构,并使用所有层次的全局表示进行DDI预测,而不依赖于额外的领域知识。这使得模型可以同样适用于只有药物化学结构可获得的inductive settings场景。
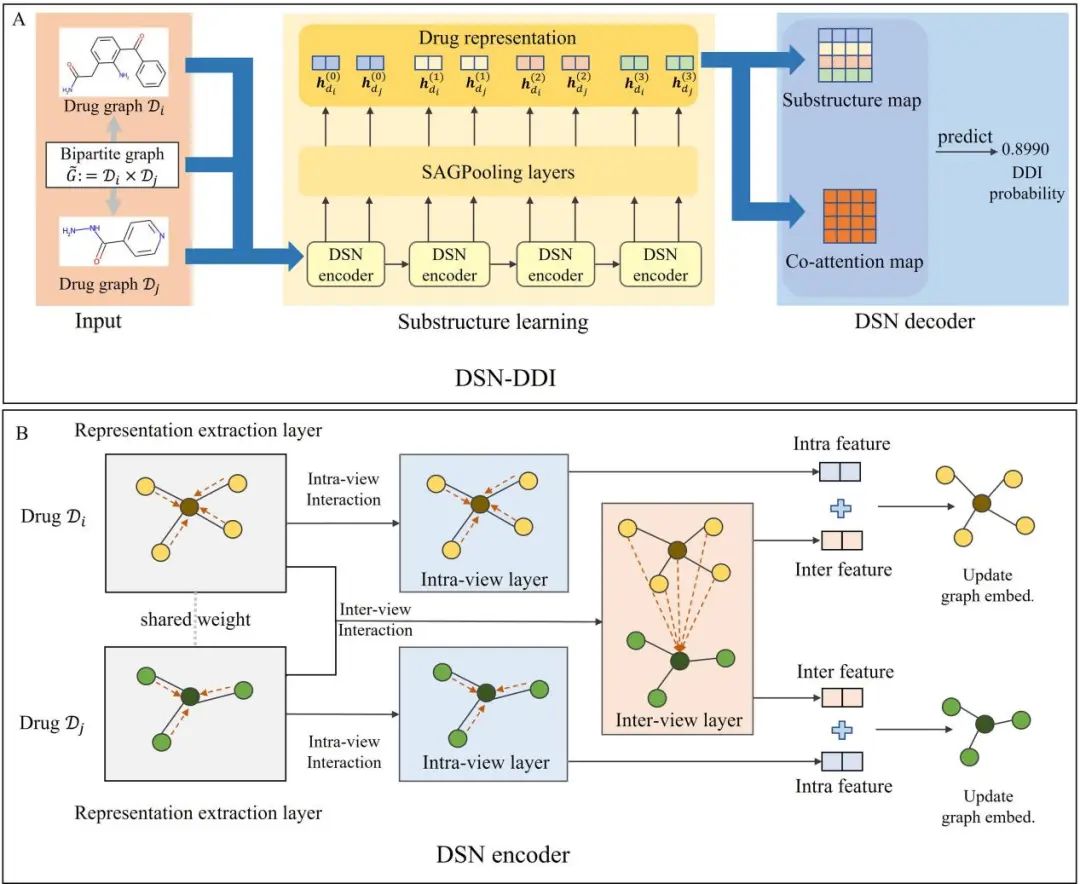
图1.DSN-DDI总体概览
模型输入包含两个用于intra-view学习的单一药物图(Di和Dj)以及一个用于inter-view学习的二部图。DSN-DDI可分为两个部分:用于从双视图学习药物对子结构的子结构学习模块,和用于子结构整合和最终预测的DSN解码器模块。对于子结构学习模块,通过一系列重复的DSN编码器学习每个原子的局部原子表示,然后通过SAGPooling层学习全局药物表示并生成药物子结构嵌入,最后所有子结构嵌入都被馈送到DSN解码器。
对于DSN编码器,一对药物由具有共享权重的表示提取层编码,然后同时被馈送到用于单个药物的intra-view层和药物对的inter-view层,最后聚合双视图信息来更新用于下一DSN编码器的节点表示。而对于DSN解码器,其被定义为给定三元组的协同注意力评分函数(),用于DDI预测。

图2.子结构提取和双视图表示学习的草图
最初的药物用分子图表示,节点代表原子,边代表化学键,图中的每个节点具有55维的初始化学特征(例如:原子符号)。在每个GNN块中,通过聚合来自相邻节点的信息来更新节点,然后进行子结构提取。一系列GNN块具有不同尺度的感受野,所以能够提取到不同的子结构。在inter view中,将一种药物的每个单一节点都与另一种药物中的所有节点相连,形成inter edges,然后通过聚合另一种药物中所有节点的信息来更新这个药物的节点。而在intra view中,一种药物中的节点则通过聚合其自身的相邻节点来更新。
研究结果
文中将DDI预测问题描述为一个二元分类问题,利用二元交叉熵(BCE)损失对模型进行端到端训练,并在两个广泛使用的数据集(DrugBank和Twosides)以及两种设置场景(transductive和inductive)上进行评估。
现有药物在transductive setting上的性能
transductive setting场景:测试集内的药物也存在于训练集中。两个数据集上正负样本的数据拆分方案都为训练:验证:测试=6:2:2。
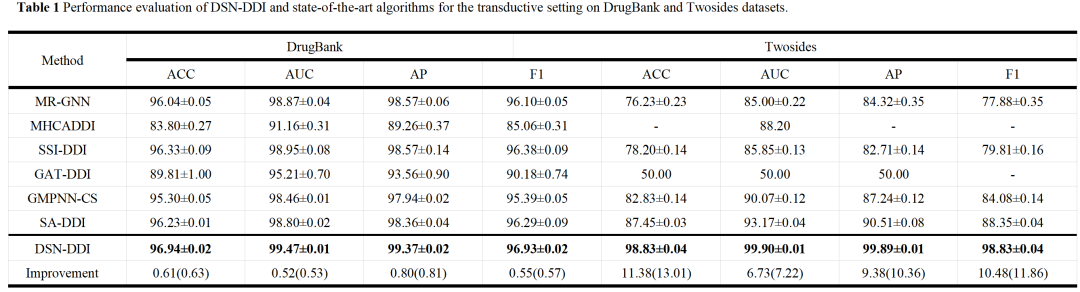
未知药物在inductive setting上的性能
Inductive setting场景:测试集中包含训练集中不存在的药物。数据拆分比例同上。随机抽取20%的药物作为未知药物,其余作为已知药物。训练集中所有正负样本都是已知药物,而测试集有两种方案:S1——测试集的正负样本都是一对未知药物,旨在预测一对新药的DDI;S2——测试集正负样本的药物对中一个是未知药物一个是已知药物,旨在预测一种新药与一种现有药物的DDI。
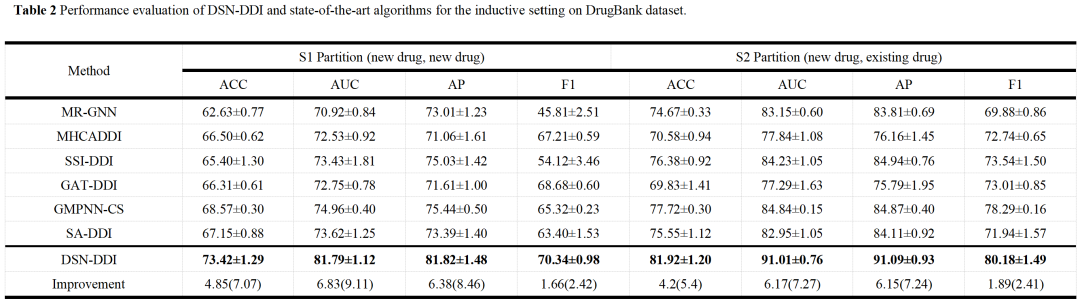
此外,为了证明DSN-DDI在实际DDI应用中的有用性,作者用已有的旧药信息训练的模型评估了对FDA新批准药物的DDI预测性能。其训练和评估设置与在inductive setting中使用的超参数相同。实验选取了三种最先进的DDI预测算法SSI-DDI、GMPNNN-CS和SA-DDI作为对比,结果如下图3。
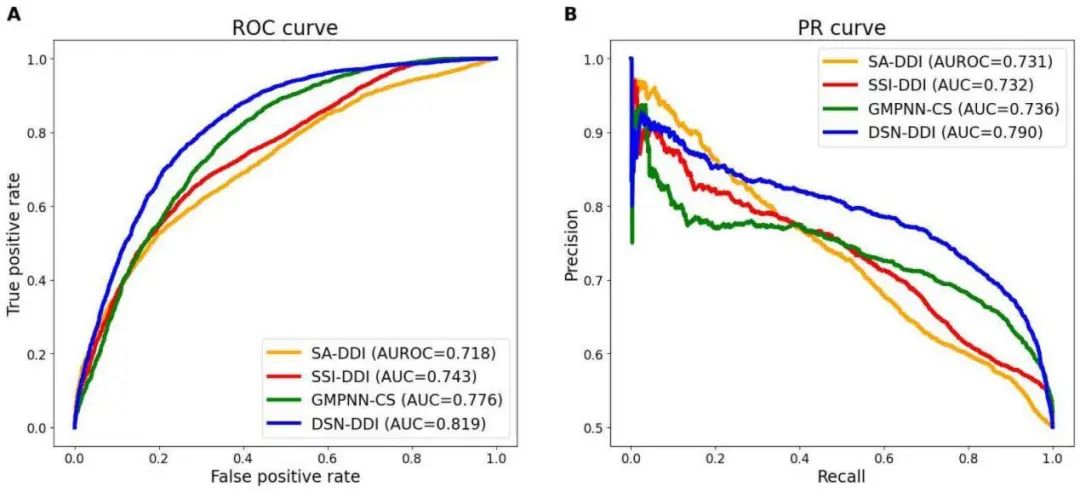
图3.SSI-DDI、GMPNN-CS、SA-DDI和DSN-DDI对FDA批准新药的性能评估
综上,在DrugBank和Twosides数据集上的综合评估表明,DSN-DDI在transductive setting上的准确率相对提高了13.01%,AUC在DrugBank和Twosides上分别达到了99.47%和99.9%。在inductive settings上对未知药物的准确率也相对提高了7.07%。此外,DSN-DDI还对新批准药物的DDI预测具有一定的实用性,对联合用药预测具有良好的可移植性。这些结果表明,DSN-DDI不仅可以作为DDI预测的有力工具,还可以作为一个通用框架,极大促进药物设计和药物发现领域的研究。
总结
这项工作提出了一个双视图子结构学习框架来预测药物对可能的多药副作用。综合实验证实了DSN-DDI在transductive和inductive两种环境下的DDI预测性能。未来的研究可以进一步提高模型在归纳学习环境下对新药的推广能力,因为这近似于一个现实世界的场景:有一个新药没有任何先前的相关药物相互作用信息。
参考资料
Zimeng Li, Shichao Zhu, Bin Shao, Xiangxiang Zeng, Tong Wang, Tie-Yan Liu, DSN-DDI: an accurate and generalized framework for drug–drug interaction prediction by dual-view representation learning, Briefings in Bioinformatics, 2023;, bbac597,
https://doi.org/10.1093/bib/bbac597
代码
https://github.com/microsoft/Drug-Interaction-Research/tree/DSN-DDI-for-DDI-Prediction

