🤫 Seurat | 强烈建议收藏的单细胞分析标准流程(差异分析与细胞注释)(五)
🤫 Seurat | 强烈建议收藏的单细胞分析标准流程(差异分析与细胞注释)(五)
生信漫卷
发布于 2023-02-24 13:46:30
发布于 2023-02-24 13:46:30
文章被收录于专栏:R语言及实用科研软件
1写在前面
本期我们介绍一下如何使用Seurat包进行差异分析,以及如何使用SingleR进行细胞注释。😘
2用到的包
rm(list = ls())
library(Seurat)
library(tidyverse)
library(SingleR)
library(celldex)
library(RColorBrewer)
library(SingleCellExperiment)
3示例数据
这里我们还是使用之前建好的srat文件,我之前保存成了.Rdata,这里就直接加载了。🧐
load("./srat2.Rdata")
srat
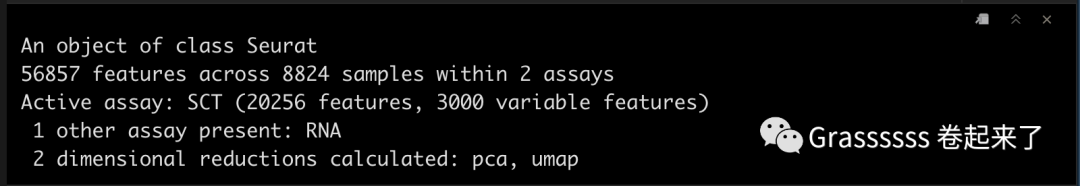
4差异分析
首先要和大家说的是,尽量使用counts进行差异分析,而不是你SCTransform等操作后的数据。😉
我们先将assy还原回原始矩阵吧,进行一下过滤。🤜
4.1 初步处理
DefaultAssay(srat) <- "RNA"
srat <- NormalizeData(srat)
srat <- FindVariableFeatures(srat, selection.method = "vst", nfeatures = 2000)
all.genes <- rownames(srat)
srat <- ScaleData(srat, features = all.genes)
4.2 计算所有marker
all.markers <- FindAllMarkers(srat, only.pos = T, min.pct = 0.5, logfc.threshold = 0.5)
4.3 Top3
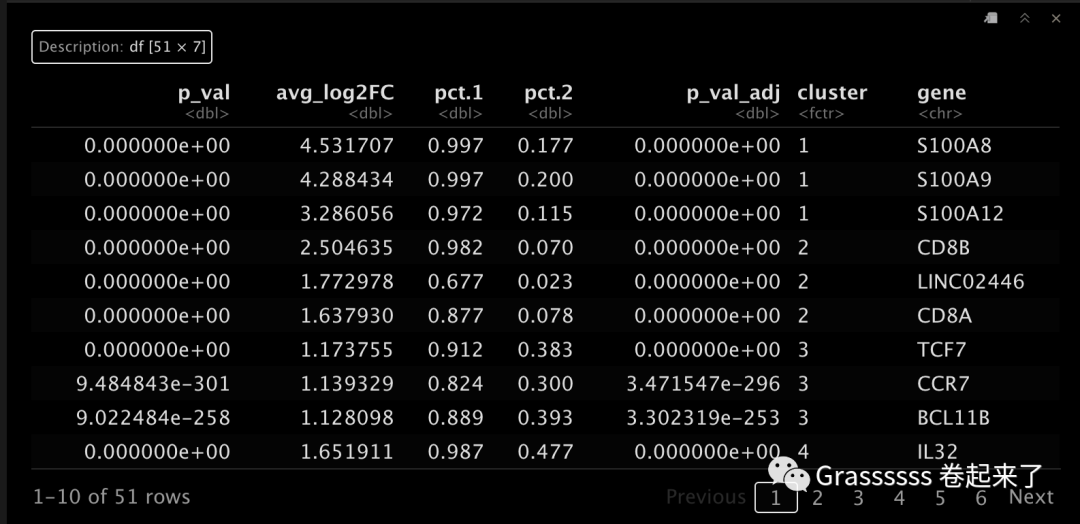
接着我们看看各个Cluster的top3基因是什么。
top3_markers <- as.data.frame(all.markers %>% group_by(cluster) %>%
top_n(n = 3, wt = avg_log2FC))
top3_markers
5细胞注释
5.1 手动注释
通过上面找到的基因,结合我们通过文献、数据库等可以成功注释细胞的类型。🤗 有的小伙伴就会说了:那不就是手动注释了吗!?🧐
是的,其实目前最准确的注释方法就是手动注释,提供一个我个人常用的数据库(CellMarker): 👇
http://xteam.xbio.top/CellMarker/
5.2 自动注释
这里我们先将Seurat转成SingleCellExperiment,方便后续操作。😘
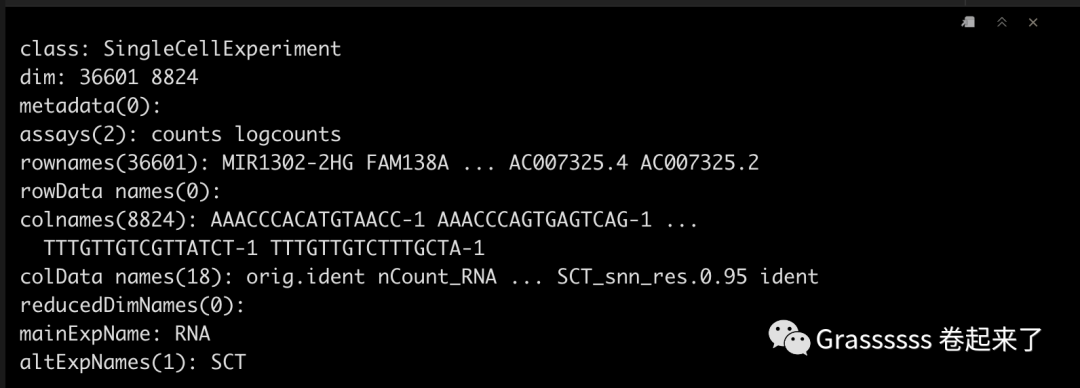
sce <- as.SingleCellExperiment(DietSeurat(srat))
sce
5.3 背景文件
背景文件我们使用celldex包的数据,大家根据自己的需要选择就行了。😘
Note! 这个文件里会有两种类型,label.main和label.fine,我们后面都使用一下吧。
monaco.ref <- celldex::MonacoImmuneData()
# hpca.ref <- celldex::HumanPrimaryCellAtlasData()
# dice.ref <- celldex::DatabaseImmuneCellExpressionData()
5.4 SingleR
常用的自动注释包,包括signleR, Cellassign等。🥳
这里我们用SingleR试一下吧。👀
monaco.main <- SingleR(test = sce,assay.type.test = 1,ref = monaco.ref,labels = monaco.ref$label.main)
monaco.fine <- SingleR(test = sce,assay.type.test = 1,ref = monaco.ref,labels = monaco.ref$label.fine)
# hpca.main <- SingleR(test = sce,assay.type.test = 1,ref = hpca.ref,labels = hpca.ref$label.main)
# hpca.fine <- SingleR(test = sce,assay.type.test = 1,ref = hpca.ref,labels = hpca.ref$label.fine)
# dice.main <- SingleR(test = sce,assay.type.test = 1,ref = dice.ref,labels = dice.ref$label.main)
# dice.fine <- SingleR(test = sce,assay.type.test = 1,ref = dice.ref,labels = dice.ref$label.fine)
5.5 查看结果
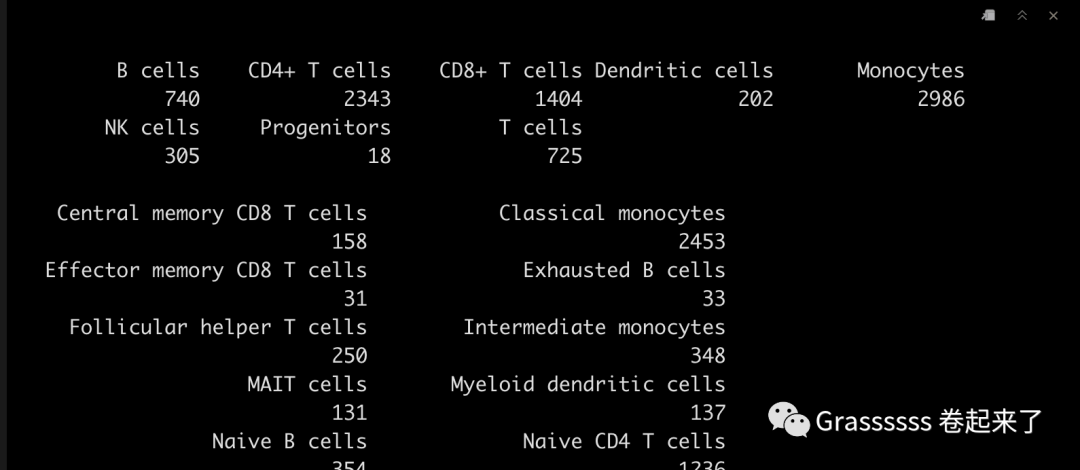
table(monaco.main$pruned.labels)
table(monaco.fine$pruned.labels)
#table(hpca.main$pruned.labels)
# table(hpca.fine$pruned.labels)
#table(dice.main$pruned.labels)
# table(dice.fine$pruned.labels)
5.6 添加信息
srat@meta.data$monaco.main <- monaco.main$pruned.labels
srat@meta.data$monaco.fine <- monaco.fine$pruned.labels
# srat@meta.data$hpca.main <- hpca.main$pruned.labels
# srat@meta.data$dice.main <- dice.main$pruned.labels
# srat@meta.data$hpca.fine <- hpca.fine$pruned.labels
# srat@meta.data$dice.fine <- dice.fine$pruned.labels
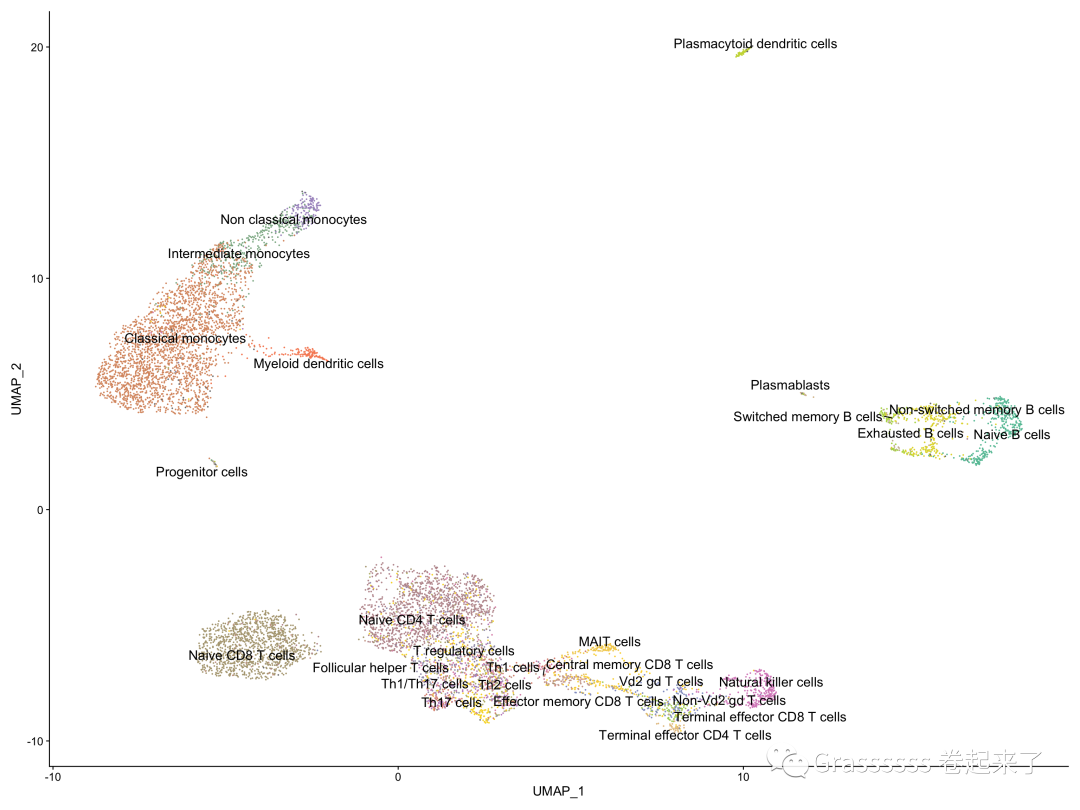
5.7 可视化
srat <- SetIdent(srat, value = "monaco.fine")
ncluster <- length(unique(srat[[]]$monaco.fine))
mycol <- colorRampPalette(brewer.pal(8, "Set2"))(ncluster)
DimPlot(srat,
label = T , repel = T,
label.size = 5,
cols = mycol) +
NoLegend()
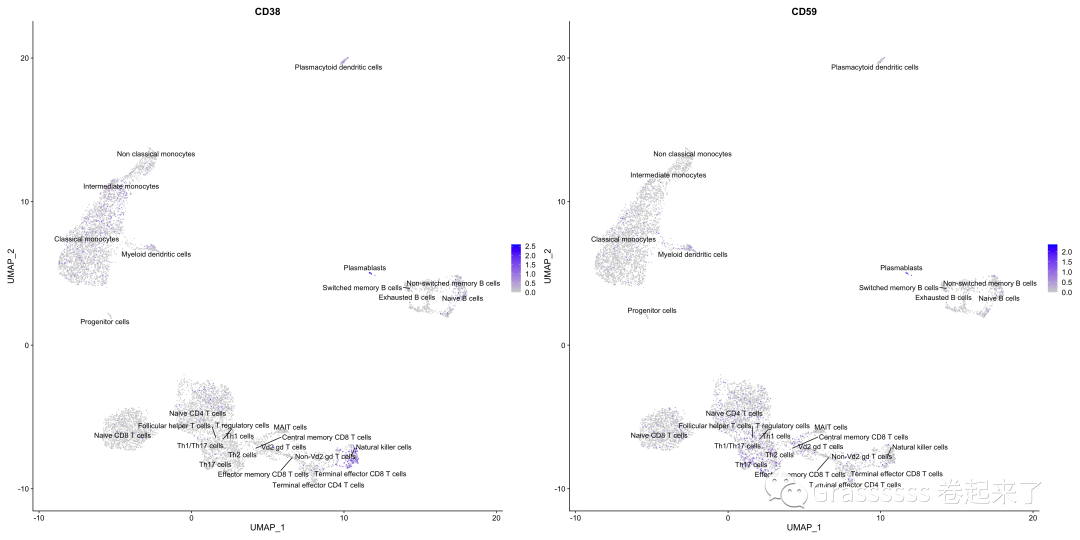
6简单验证
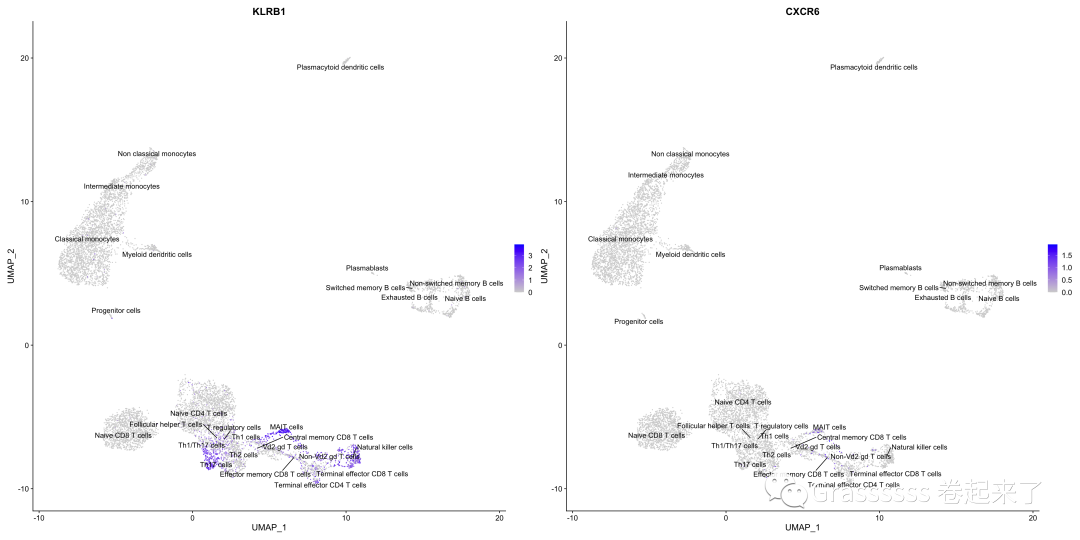
我们简单验证一下注释效果,用几个已知marker看一下吧,已知:👇
plasma B,CD38和CD59;MAIT cells,CD161 (KLRB1)和CXCR6。
6.1 plasma B
FeaturePlot(srat,c("CD38","CD59"),
label = T, repel = T)
6.2 MAIT cells
FeaturePlot(srat,c("KLRB1", "CXCR6"),
label = T, repel = T)
最后祝大家早日不卷!~
本文参与 腾讯云自媒体同步曝光计划,分享自微信公众号。
原始发表:2022-10-22,如有侵权请联系 cloudcommunity@tencent.com 删除
评论
登录后参与评论
推荐阅读
目录
相关产品与服务
数据库
云数据库为企业提供了完善的关系型数据库、非关系型数据库、分析型数据库和数据库生态工具。您可以通过产品选择和组合搭建,轻松实现高可靠、高可用性、高性能等数据库需求。云数据库服务也可大幅减少您的运维工作量,更专注于业务发展,让企业一站式享受数据上云及分布式架构的技术红利!
