拯救你多年前质量差的单细胞数据
拯救你多年前质量差的单细胞数据
不知道有多少课题组仍然是有一些自己的束之高阁的单细胞转录组测序数据呢,也许是年少轻狂也许是轻信了销售的嘴。
看到了前两天(2023年1月31日)的一篇肺癌单细胞文章:《Single-cell analysis reveals prognostic fibroblast subpopulations linked to molecular and immunological subtypes of lung cancer》,下意识的看了看它是否公开测序数据方便我们做教学,发现居然是;https://www.ncbi.nlm.nih.gov/geo/query/acc.cgi?acc=GSE153935
熟悉GEO规则的小伙伴呢都知道,它这个ID是顺序增加的,现在一般来说都是2万以上的数值了,所以我留意了一下它这个数据集确实是三年前就公开了,而且公开的时候对这个数据集下结论是非小细胞肺癌病人的成纤维单细胞亚群有异质性,可以分成7群:
- Inflammatory fibroblasts
- myofibroblasts (representing terminal differentiation states),
- quiescent fibroblasts,
- proto-myofibroblasts (x2)
- proto-inflammatory fibroblasts (x2).
其中前面的两个是非常经典是 myofibroblastic-CAFs (myoCAFs) and inflammatory-CAFs (iCAFs) ,后面的那些亚群大概率是具有数据集异质性,仅仅是单细胞降维聚类分群后,算法区分的数学意义的单细胞亚群而且有生物学功能数据库注释,然后认为挑选合适的功能作为它的名字。
这个GSE153935数据集虽然有18个单细胞样品,如下所示:
GSM4658758 Donor 1 tumour
GSM4658759 Donor 2 non-involved lung
GSM4658760 Donor 2 tumour
GSM4658761 Donor 3 non-involved lung
GSM4658762 Donor 3 tumour
GSM4658763 Donor 4 tumour
GSM4658764 Donor 5 tumour
GSM4658765 Donor 6 tumour
GSM4658766 Donor 7 non-involved lung
GSM4658767 Donor 7 tumour
GSM4658768 Donor 8 tumour
GSM4658769 Donor 9 non-involved lung
GSM4658770 Donor 9 tumour
GSM4658771 Donor 10 non-involved lung
GSM4658772 Donor 10 tumour
GSM4658773 Donor 11 non-involved lung
GSM4658774 Donor 11 tumour
GSM4658775 Donor 12 tumour
但并不是通过实验技术手段富集了成纤维细胞,诡异的是表达量矩阵文件如此小:
GSE153935_Merged_StromalCells.txt.gz 4.3 Mb
GSE153935_TLDS_AllCells.txt.gz 10.2 Mb
文章里面的第一层次降维聚类分群也表明确实是常规肿瘤单细胞:
- This identified multiple distinct mesenchymal, immune and epithelial cell populations
- The mesenchymal cells included two separate clusters:
- endothelial cells (marked by VWF among other canonical markers)
- stromal cells (marked by DCN and DPT)
如下所示:
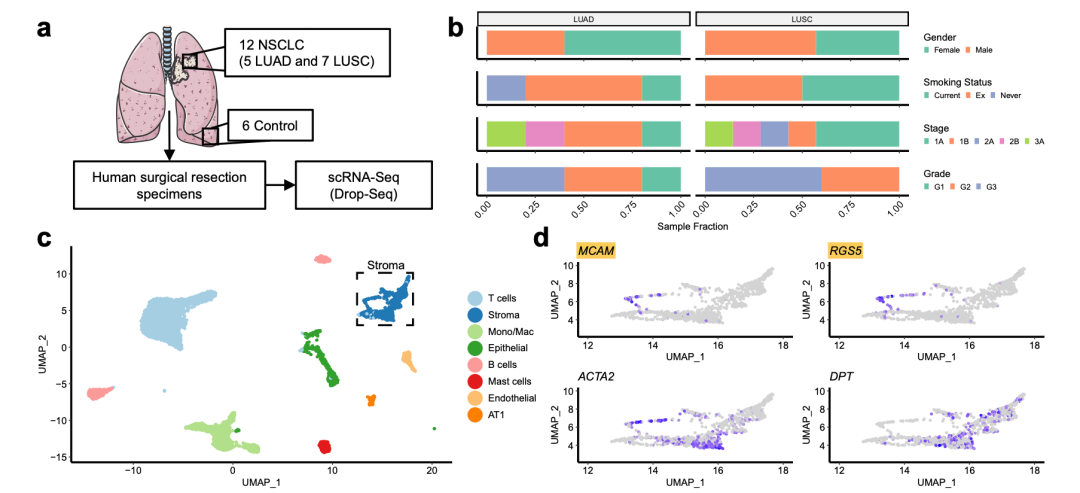
就是我们这五年一直分享的,针对肿瘤单细胞的第一层次降维聚类分群 , 是:
- immune (CD45+,PTPRC),
- epithelial/cancer (EpCAM+,EPCAM),
- stromal (CD10+,MME,fibo or CD31+,PECAM1,endo)
参考我前面介绍过 CNS图表复现08—肿瘤单细胞数据第一次分群通用规则,这3大单细胞亚群构成了肿瘤免疫微环境的复杂。绝大部分文章都是抓住免疫细胞亚群进行细分,包括淋巴系(T,B,NK细胞)和髓系(单核,树突,巨噬,粒细胞)的两大类作为第二次细分亚群。但是也有不少文章是抓住stromal 里面的fibo 和endo进行细分,并且编造生物学故事的。
成纤维细胞里面的混入的周细胞(壁细胞)( mural cells)
一般来说,肿瘤单细胞里面排除了免疫细胞和上皮细胞后的,都是基质细胞(间质细胞),这里面继续排除内皮细胞后,就是成纤维混合体。但是这个排除法没办法排除周细胞和平滑肌细胞,所以很多时候成纤维混合体其实就包含它们。这个不能怪数据分析人员,因为就是成纤维混合体的定义不清晰导致。
感兴趣成纤维细胞的非常多,可能主要是因为肿瘤领域的火爆,肿瘤微环境里面的免疫相关,不管是淋巴系还是髓系,都有了大量的 成果。反而是肿瘤微环境里面的基质细胞,比如内皮细胞,成纤维和周细胞目前还大有可为。
前面提到了:肿瘤样品的单细胞需要提取上皮细胞继续细分,就是感兴趣的fibroblasts等细胞亚群占比非常少,所以研究者们做了第二次单细胞转录组数据,见:什么,你想要的单细胞亚群比例太少了?,其实这样的策略屡见不鲜。在小鼠模型里面也有:4T1这个TNBC小鼠肿瘤模型的CAFs异质性,其实大家并不能很好的确定自己研究的对象就是成纤维细胞。比如这个 肿瘤相关成纤维细胞异质性的文章里面 就是把成纤维分成了6群:
- Subcluster 0 被作者定义为 vascular CAFs ,高表达 GJA4, and RGS5 等
- Subcluster 1 被作者定义为 matrix CAFs ,高表达 LUM, DCN, and VCAN等
- Subcluster 2 被作者定义为 inflammatory CAFs ,高表达 complement genes (C3 and C7). 等
- Subcluster 3 被作者定义为 antigen-presenting CAFs ,高表达CD74, HLA- DRA, and HLA-DRB1 等
- Subcluster 4 被作者定义为 EMT-like CAFs ,高表达 KRT19, KRT8 等
- Subcluster 5 被作者定义为 lip- ofibroblast–c5–FABP1 ,高表达APOA2, FABP1, FABP4, and FRZB等
如果按照其它理论,会认为所谓的 vascular CAFs ,高表达 GJA4, and RGS5 等,其实是周细胞,而 EMT-like CAFs ,高表达 KRT19, KRT8 ,可能都不一定是一个真正的单细胞亚群。
这个时候多看不同的文献,进行归纳汇总,应该是一个比较好的策略。
本文也是使用了大量笔墨描述如何区分 mural cells (MCAM and RGS5), mural cells and myofibroblasts (ACTA2) and fibroblasts (DPT), 还是蛮容易区分的 mural cells (n = 69) and fibroblasts (n = 885) :
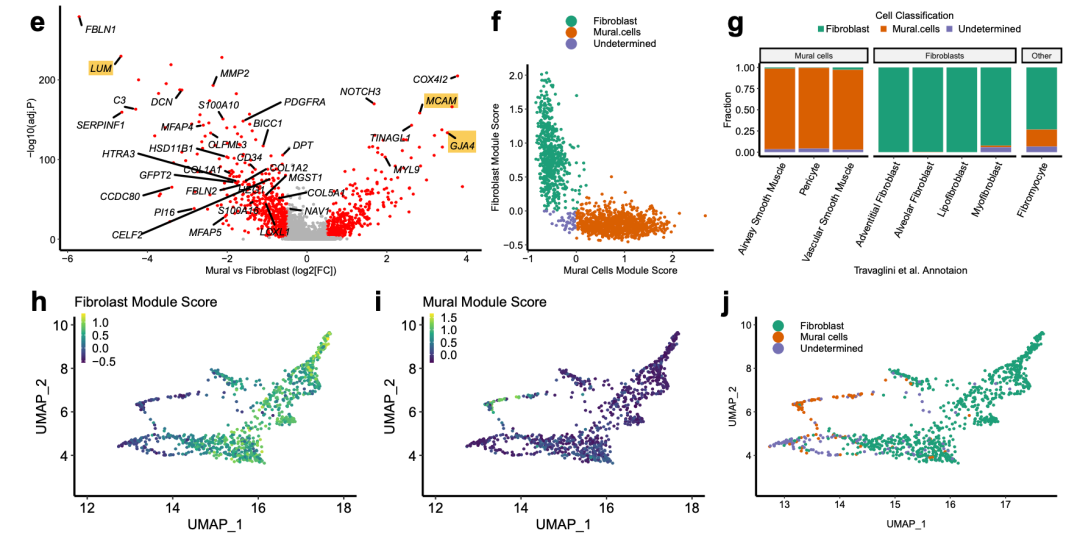
混入的周细胞(壁细胞)( mural cells)
很明显作者不认为混入的周细胞(壁细胞)( mural cells)是成纤维,是需要排除的。但是 myofibroblasts (ACTA2) 也有可能是平滑肌细胞,但是作者这个时候不排除了,这个时候就是生命科学领域的乱象丛生了,我们没办法苛责。
如果到这里,无论我们前面的GSE153935数据集是三年前还是五年前,都是十几分钟就分析完毕了,不可能是支撑一个Nature Communications | (2023)发表,接下来就一起看看作者是如何神来之笔,拯救这个尘封已久的单细胞转录组数据。
结合公共数据集
首先作者自己的肺癌单细胞转录组数据集里面的成纤维细胞数量就很少,测完序后终于等到了很多肺癌数据集发表,一口气结合了6个数据集,挑选到了 近万个成纤维细胞:
- 5183 from 39 control tissues;
- 3440 from 46 LUAD samples
- 654 from 16 LUSC samples).
整合后可以比较清晰的看到3个有独立特性的成纤维单细胞亚群:
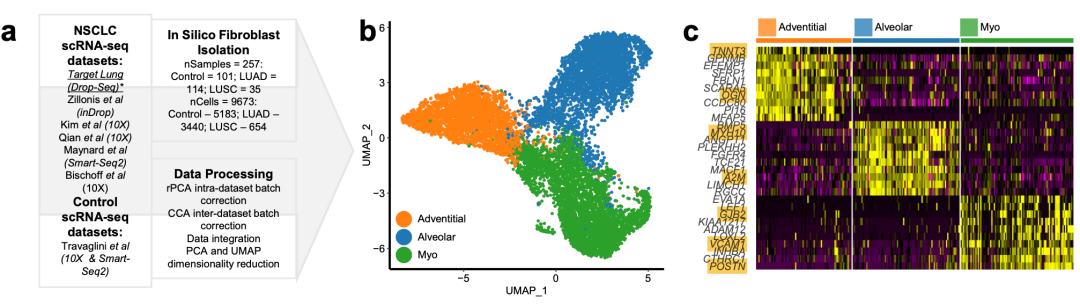
3个有独立特性的成纤维单细胞亚群
每个单细胞亚群都可以跟其它两个进行 差异分析,来确立特征:
- 622 genes upregulated by myofibroblasts,比如:MMP11, POSTN, CTHRC1, COL1A1, ACTA2 and COL3A1.
- 481 genes upregulated in adventitial fibroblasts ,比如:PI16, IGFBP6, MFAP5, APOD, PLA2G2A and GSN
- 672 upregulated genes, 78 of which remained significant in sample-level analysis -including MACF1, RGCC, INMT, LIMCH1, A2M and GPC3
值得注意的是肺癌里面的成纤维单细胞亚群分成了3个:adventitial, alveolar and myofibroblasts ,但是来源于多个数据集多个病人,所以这里作者做了一个sample-level的数据处理:
但是 To comprehensively characterise these fibroblast subpopulations at a population level, we first calculated sample-level gene expression profiles for each subpopulation (averaged over single cells) and then performed differential gene expression analysis.
所以要善于结合公共数据集。

