umap的单细胞可视化效果比tSNE好
昨晚有幸听了上海长海医院任善成教授的关于前列腺癌的精准诊疗的演讲,任教授提到了他们2021年1月在在 Nature Cell Biology 杂志发表了题为《Single-cell analysis reveals transcriptomic remodellings in distinct cell types that contribute to human prostate cancer progression》论文.
我就顺手下载了打开读了一下,发现他们的单细胞转录组数据降维聚类分群后的可视化选择的是tSNE,感觉看起来呢各个单细胞亚群之间的边界线有点不清晰。所以想着下载作者提供的单细胞表达量矩阵自己走一遍流程使用umap可视化看看。作者的tSNE图如下所示:
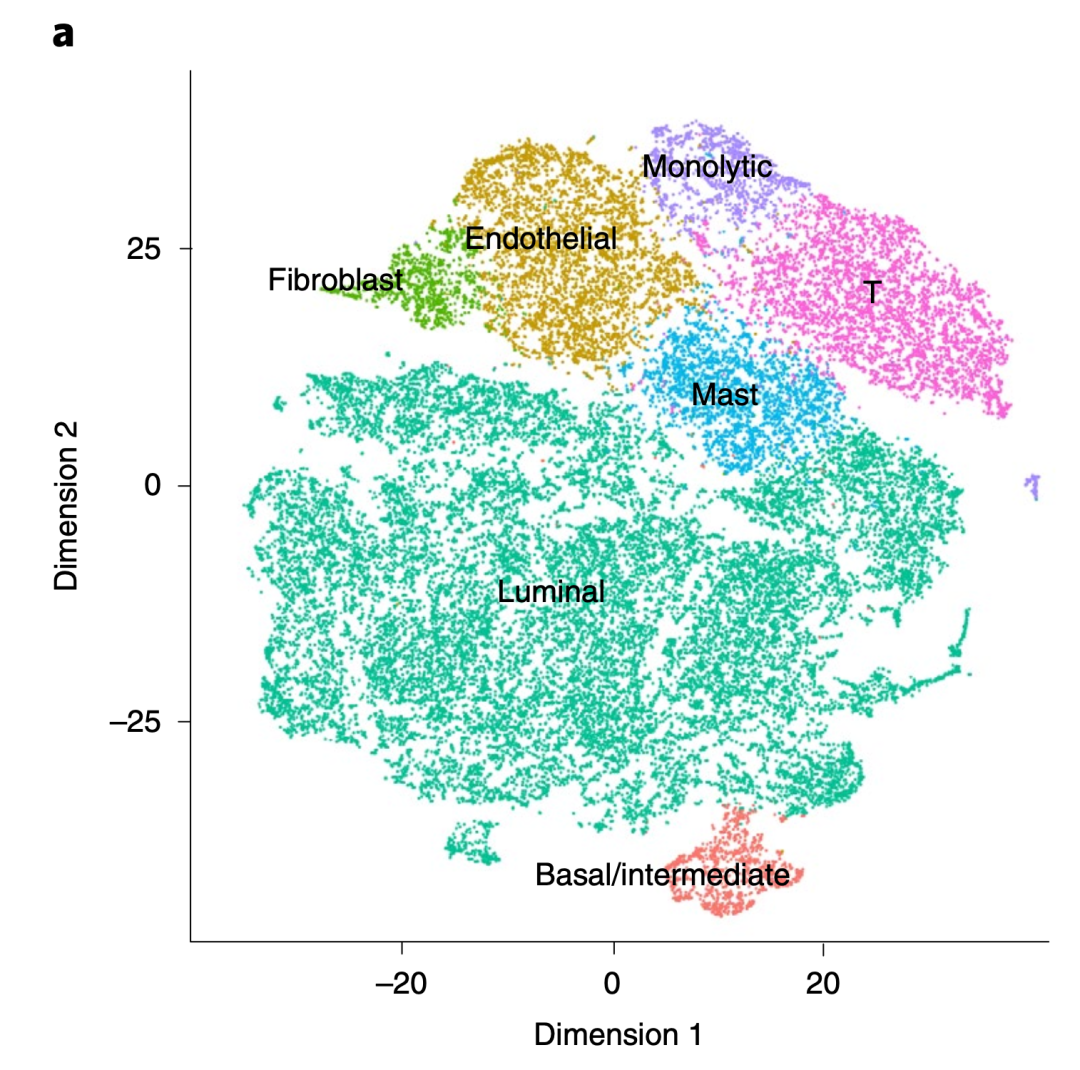
tSNE图
表达量矩阵在:https://www.ncbi.nlm.nih.gov/geo/query/acc.cgi?acc=GSE141445
是 36,424 single-cells from 13 prostate tumors ,作者的实验技术很棒,取样的病人肿瘤组织样品而且里面绝大部分都是恶性肿瘤上皮细胞,在其它癌症很少看到这样的完美的的结果。作者提供的表达量矩阵是:
GSM4203181_data.matrix.txt.gz 675.4 Mb
GSM4203181_data.raw.matrix.txt.gz 81.6 Mb
大家很容易自行下载,然后读取就可以成为Seurat对象啦 :
library(data.table)
Sys.time()
raw.data <- fread( 'GSM4203181_data.raw.matrix.txt.gz',
data.table = F)
Sys.time()
dim(raw.data) #
raw.data[1:4,1:4]
rownames(raw.data)=raw.data[,1]
raw.data=raw.data[,-1]
head(colnames(raw.data))
library(stringr)
metadata <- as.data.frame(str_split(colnames(raw.data),'[-]',
simplify = T))
head(metadata)
table(metadata[,2])
metadata=metadata[,c(2,1)]
colnames(metadata)=c('orig.ident' ,'barcode')
metadata$orig.ident=paste0('p',metadata$orig.ident)
table(metadata$orig.ident)
rownames(metadata) = colnames(raw.data)
identical(rownames(metadata),colnames(raw.data))
sce.all <- CreateSeuratObject(counts = raw.data)
sce.all <- AddMetaData(object = sce.all, metadata = metadata)
as.data.frame(sce.all@assays$RNA@counts[1:10, 1:2])
head(sce.all@meta.data, 10)
table(sce.all@meta.data$orig.ident)
并且对这个Seurat对象进行降维聚类分群,参考前面的例子:人人都能学会的单细胞聚类分群注释 ,而且每个亚群找高表达量基因,如果你对单细胞数据分析还没有基础认知,可以看基础10讲:
- 01. 上游分析流程
- 02.课题多少个样品,测序数据量如何
- 03. 过滤不合格细胞和基因(数据质控很重要)
- 04. 过滤线粒体核糖体基因
- 05. 去除细胞效应和基因效应
- 06.单细胞转录组数据的降维聚类分群
- 07.单细胞转录组数据处理之细胞亚群注释
- 08.把拿到的亚群进行更细致的分群
- 09.单细胞转录组数据处理之细胞亚群比例比较
因为前面的例子:人人都能学会的单细胞聚类分群注释 ,是很早期的笔记,那个时候还没有采用harmony这样的节省时间的多个单细胞样品整合的算法。第一次默认分群和简单的可视化基因如下所示:
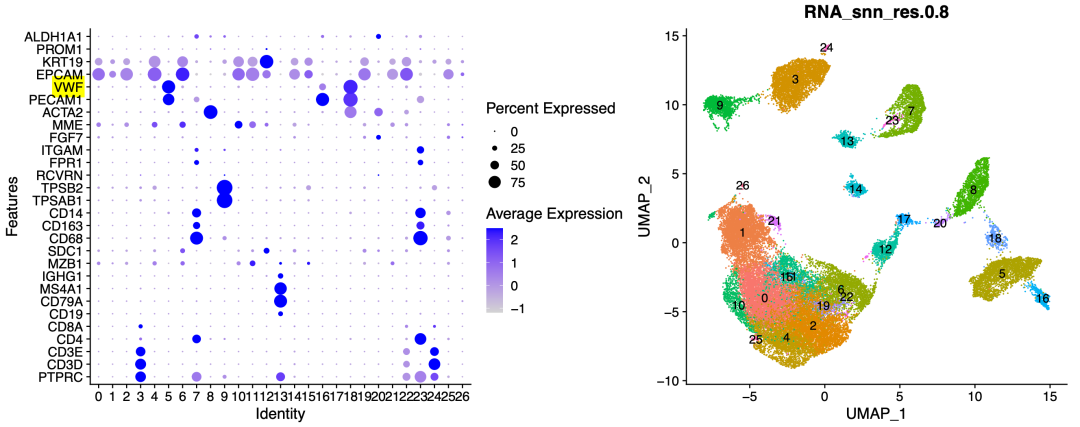
简单的可视化基因
因为我这个是标准流程,所以主要是第一层次降维聚类分群,区分上皮细胞,免疫细胞,间质细胞即可。就是:
- immune (CD45+,PTPRC),
- epithelial/cancer (EpCAM+,EPCAM),
- stromal (CD10+,MME,fibo or CD31+,PECAM1,endo)
参考我前面介绍过 CNS图表复现08—肿瘤单细胞数据第一次分群通用规则,这3大单细胞亚群构成了肿瘤免疫微环境的复杂。绝大部分文章都是抓住免疫细胞亚群进行细分,包括淋巴系(T,B,NK细胞)和髓系(单核,树突,巨噬,粒细胞)的两大类作为第二次细分亚群。但是也有不少文章是抓住stromal 里面的fibo 和endo进行细分,并且编造生物学故事的。
大家可以自己去读文章, 复制粘贴里面的单细胞分群规则以及对应的基因。我们直接看看默认命名后的结果:
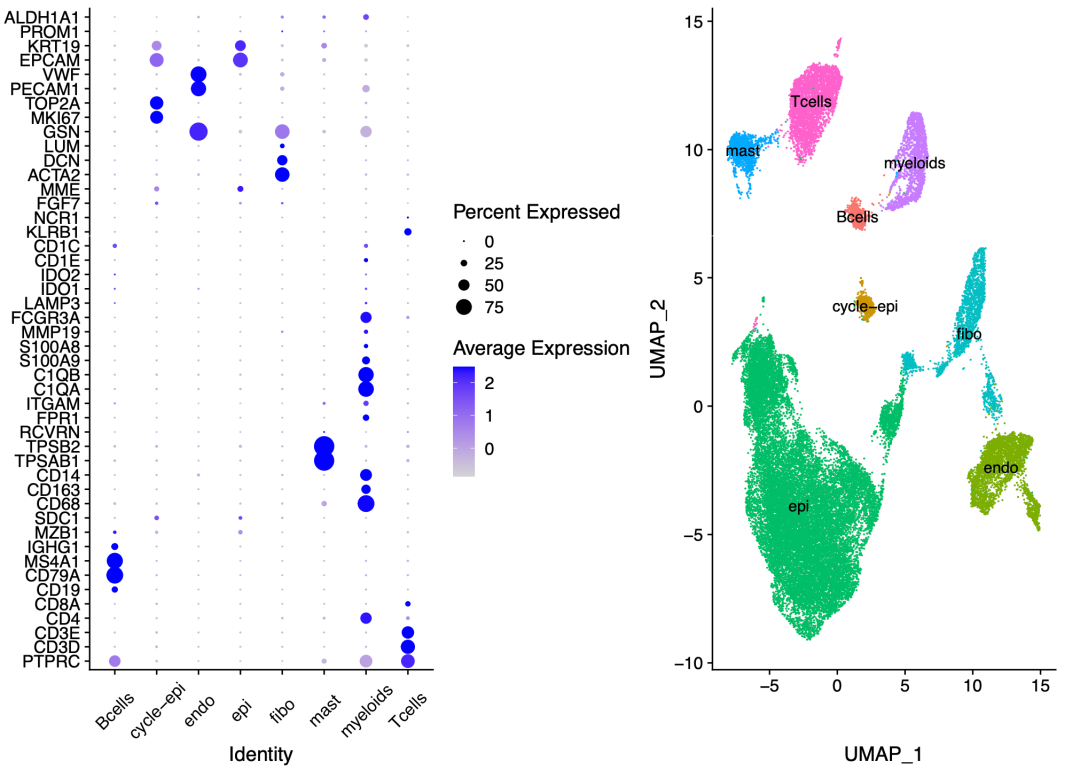
我自己觉得,我们复现后的降维聚类分群结果,理论上比文章的好看一点,这就是我先表达的结论:umap的单细胞可视化效果比tSNE好
不知道你是否认同呢?
学徒作业
从我们上面的简单的降维聚类分群来块,我们所命名的成纤维和内皮其实仍然是异质性比较大的群,比如上面的成纤维里面肯定是有周细胞和平滑肌细胞,需要细分。另外内皮细胞也是有很多,要是区分成为了淋巴内皮和血管内皮, 其中血管可以细分为动脉静脉和毛细血管:
- lymphatic ECs (LECs; CCL21, PROX1).
- arteries (HEY1, IGFBP3), capillaries (CD36, CA4), veins (ACKR1)
而且这个内皮细胞里面的还混入了周细胞,特别麻烦,有可能是双细胞也有可能是全新的有功能的真实单细胞亚群。
作业就是,提取上面的成纤维和内皮单细胞亚群后进行亚群细分,继续划分子集,并且尝试命名或者给出生物学意义。
