甲基化探针相对于基因来说太多了怎么办
如果是表达量芯片,探针数量很明显是比标准的2万多个蛋白质编码基因多不少, 很容易理解嘛,因为每个基因长度那么给力,在上面设计多个探针很正常。
针对表达量芯片,我们会有一个很常规的操作,就是相当于基因来说的去冗余操作,如果一个基因对应多个探针我们会仅仅是保留表达量最大的探针作为那个基因的唯一表达量,这样之前的五六万个探针的表达量矩阵去冗余操作后就是两三万个基因的表达量矩阵啦。但是这样的操作并不是万无一失,仅仅是一个优先选择而已。之前就学员提出来了一个蛮古老的表达量芯片数据集的讨论,因为 它是做了这个PPARα的基因敲除,但是学员在分析表达量矩阵做差异的时候发现PPARα本身其实并没有统计学显著的差异表达。数据集是:https://www.ncbi.nlm.nih.gov/geo/query/acc.cgi?acc=GSE8292
学员选取的样品是control条件下,而不是 a single dose of WY14643 (400 μl of 10 mg/ml WY14643) 药物处理的 :
GSM205769 liver_wildtype_WY14643_6h_rep1
GSM205770 liver_wildtype_WY14643_6h_rep2
GSM205771 liver_wildtype_WY14643_6h_rep3
GSM205772 liver_wildtype_WY14643_6h_rep4
GSM205778 liver_PPARa-knockout_WY14643_6h_rep1
GSM205779 liver_PPARa-knockout_WY14643_6h_rep2
GSM205780 liver_PPARa-knockout_WY14643_6h_rep3
GSM205781 liver_PPARa-knockout_WY14643_6h_rep4
GSM205782 liver_PPARa-knockout_WY14643_6h_rep5
同样的control条件下,我们比较 野生型和PPARα的基因敲除两个分组, 理论上肯定是PPARα的基因表达量应该是有统计学显著的差异。但是如果一个基因对应多个探针我们会仅仅是保留表达量最大的探针作为那个基因的唯一表达量,我们就会发现PPARα的基因表达量没有统计学显著的差异,因为这个基因有多个探针。详见:一个基因上面有多个探针最后只能选一个吗
如果是甲基化芯片,那么探针数量会更多,从27K到450K再到850K的探针数量,但是基因仍然是标准的2万多个蛋白质编码基因,基因不仅仅是很长而且基因这个时候有结构,启动子区域,外显子,内含子,这些不同元件的生物学意义不一样,所以每个基因设计十几个甚至几十个探针都有可能。这个时候就国家的不可能简简单单去冗余了,因为同一个基因的不同位置的甲基化探针的信号值的生物学意义是不一样的。
查看champ包的450k芯片的注释信息
champ包是目前甲基化芯片数据处理的集大成者:
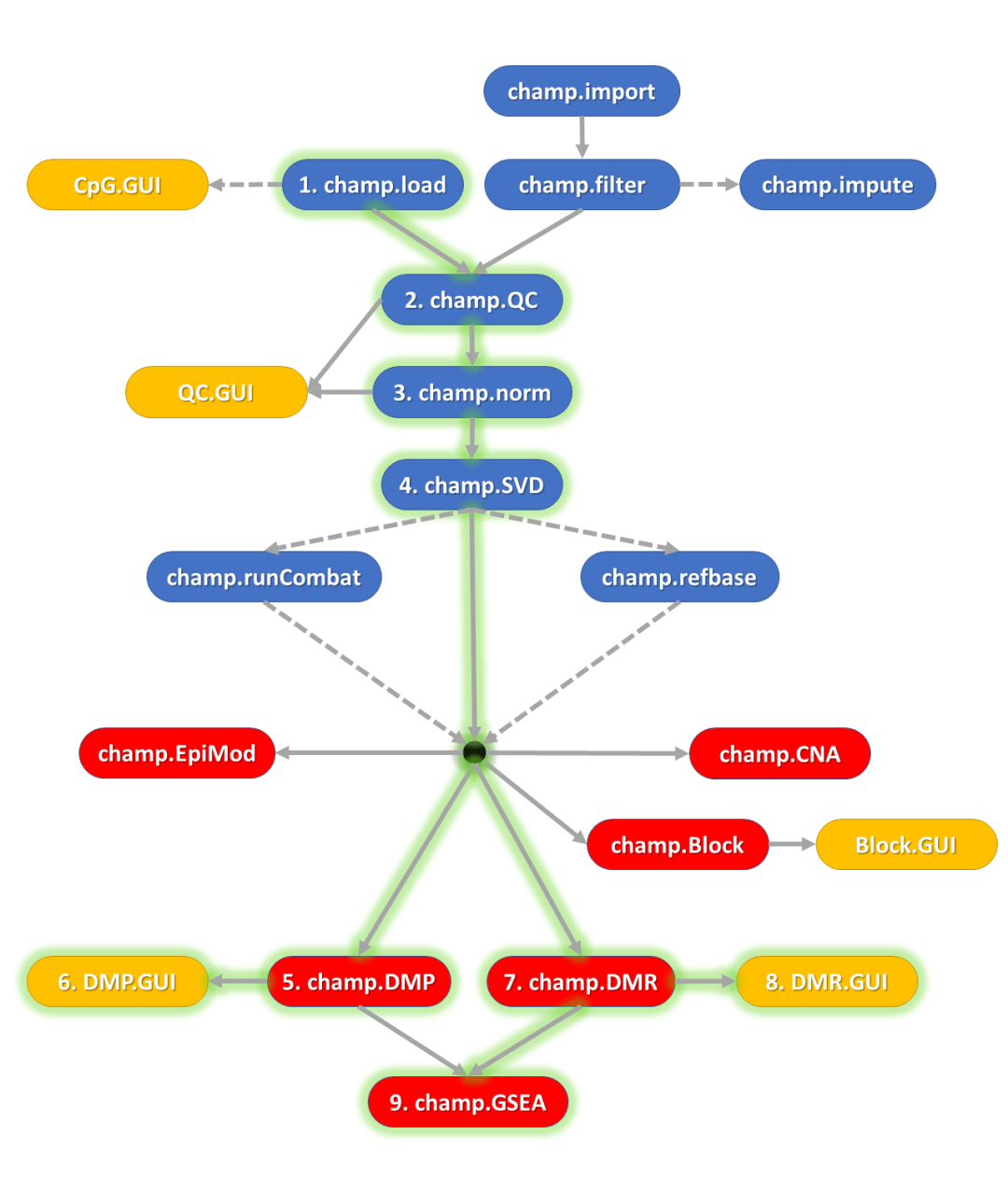
甲基化芯片数据处理的集大成者
也是我们推荐给大家的首选流程:
rm(list = ls())
options(stringsAsFactors = F)
library("ChAMP")
library("minfi")
require(GEOquery)
require(Biobase)
data(hm450.manifest.hg19)
data(probe.features)
head(hm450.manifest.hg19)
head(probe.features)
sort(table(probe.features$feat.cgi))
sort(table(probe.features$feature))
可以看到甲基化探针确实是有非常多的属性 :
> sort(table(probe.features$feat.cgi))
1stExon-shelf TSS200-shelf TSS1500-shelf 3'UTR-shelf 3'UTR-island
368 857 1685 1802 1992
1stExon-shore 3'UTR-shore 5'UTR-shelf 1stExon-opensea TSS200-opensea
2506 3426 3789 4282 9058
TSS200-shore 5'UTR-shore 3'UTR-opensea 5'UTR-opensea TSS1500-opensea
9372 9460 10274 11855 14667
1stExon-island 5'UTR-island IGR-shelf Body-shelf IGR-shore
15581 17581 18390 20253 21121
TSS1500-island IGR-island TSS1500-shore TSS200-island Body-shore
21610 22392 31022 32996 35160
Body-island IGR-opensea Body-opensea
38102 57814 68162
> sort(table(probe.features$feature))
3'UTR 1stExon 5'UTR TSS200 TSS1500 IGR Body
17494 22737 42685 52283 68984 119717 161677
而且450K的探针里面有接近120K的探针甚至是没有注释到基因本身:
> table(probe.features$gene!='')
FALSE TRUE
119717 365860
可以看到这119717个没有注释到基因的就是IGR的属性啦,也是可以细分成为:
IGR-shore IGR-island IGR-opensea
21121 22392 57814
IGR-shelf
18390
需要对表观背景知识有一点了解才能理解上面的450K芯片的探针的分布情况。
既然一个基因会设计十几个探针甚至几十个探针,如何量化这个基因的甲基化信号值就需要考虑生物学背景啦。
单个基因的启动子区域全部甲基化探针的平均值
比如2018的文章:《Deep Learning–Based Multi-Omics Integration Robustly Predicts Survival in Liver Cancer》就是对单个基因的启动子区域全部甲基化探针的平均值的处理:
For the DNA methylation, we mapped CpG islands within 1,500 base pairs (bp) ahead of transcription start sites (TSS) of genes and averaged their methylation values.
也就是说仅仅是考虑了 TSS1500-island 的21610个探针,可以看到, 这两万多个探针并不意味有两万多个基因,因为仍然是一个基因有好多个探针:
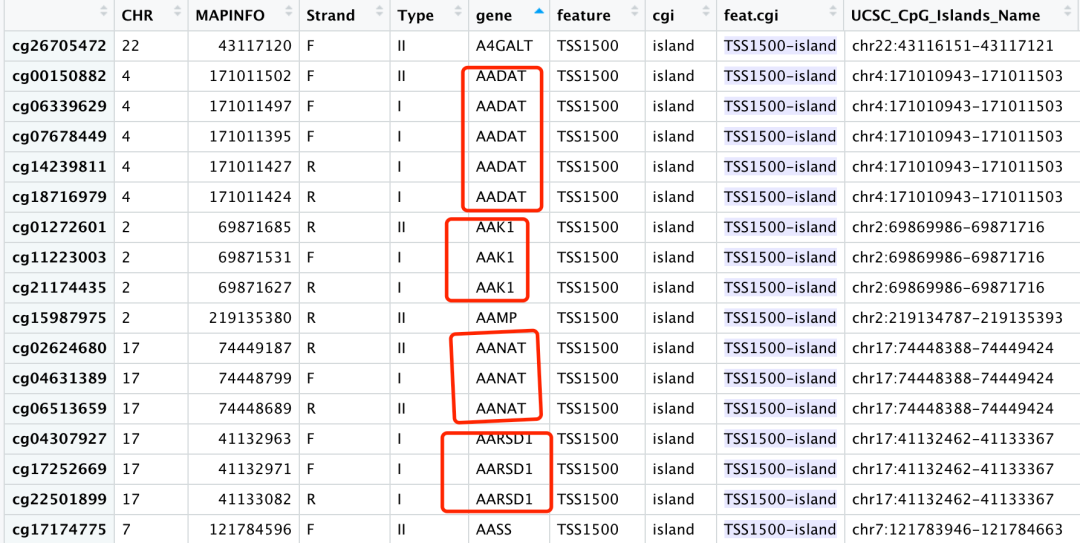
一个基因有好多个探针
ChAMP包中包括了两个测试集。
library("ChAMP")
# HumanMethylation450 data (.idat)
testDir=system.file("extdata",package="ChAMPdata")
myLoad <- champ.load(testDir,arraytype="450K")
# simulated EPIC data
data(EPICSimData)
我们可以针对这个数据集来做单个基因的启动子区域全部甲基化探针的平均值:
library("ChAMP")
data(EPICSimData)
head(myLoad$pd)
myLoad$beta[1:4,1:4]
data(probe.features)
sort(table(probe.features$cgi))
sort(table(probe.features$feature))
sort(table(probe.features$feat.cgi))
cg = probe.features[probe.features$feat.cgi=='TSS1500-island',]
length(unique(cg$gene))
cg=cg[rownames(cg) %in% rownames(myLoad$beta), ]
lt= split(rownames(cg),as.character(cg$gene))
TSS1500_beta <- do.call(rbind,
lapply( lt , function(x){
colMeans(myLoad$beta[x,,drop=F])
}))
居然就六千多个基因了,唉,代码很漂亮,就是运行起来好慢啊。我号召大家帮忙改写最后一句代码:
第一个方案是并行:

并行并不是真正的加速
之前是耗时3分钟,哪怕是并行1000000个线程也不可能很快。但是真正的改写代码可以造成百倍的加速:

百倍的加速
原来的方案需要3min,现在只需要1.26秒,真正的百倍加速!!!
学徒作业
大家可以尝试自己的方式来改写这个代码!
