Top_Cow 2023——用于CTA和MRA的Willis环的拓扑解剖结构分割docker推理部署
Top_Cow 2023——用于CTA和MRA的Willis环的拓扑解剖结构分割docker推理部署
今天将分享CTA和MRA的Willis环的拓扑解剖结构分割docker推理部署完整实现版本,为了方便大家学习理解整个流程,将整个流程步骤进行了整理,并给出详细的步骤结果。感兴趣的朋友赶紧动手试一试吧。
一、Top_Cow 2023介绍
威利斯环 (CoW) 是连接大脑前循环和后循环以及左右大脑半球的重要动脉吻合网络。由于其中心地位,CoW 通常涉及动脉瘤和中风等疾病。临床上,CoW的血管结构被认为会影响中风的发生和严重程度。因此,对 CoW 的准确表征具有重要的临床意义。然而临床医生明确表达了对分析和比较CoW血管结构的高效软件工具的未满足需求。从医学血管造影图像评估CoW的解剖结构和血管成分仍然是一项专家任务且耗时。此外,CoW自然有许多解剖学变体。据估计,我们人口中只有大约一半拥有完整的CoW。CoW的解剖结构因人而异也不例外。自动化和个性化的CoW血管特征将引起临床和研究界的极大兴趣。
在这里,提出了第一个关于 CoW 血管结构提取和脑血管片段注释的公开挑战,两种常见的血管造影成像方式,即磁共振血管造影(MRA)和计算机断层扫描血管造影(CTA)。确定了高质量解剖注释、更新的成像数据集和多种模式的数据集之间的差距。通过发布一个涵盖 CTA 和 MRA 的公共数据集,并对脉管系统进行仔细注释,希望促进对 CoW 血管分割结果的研究和更好的基准比较。
二、Top_Cow 2023任务
目的是从3D血管造影成像中提取 CoW 血管结构。有两个子任务:CoW 血管的二元分割和多类 CoW 解剖分割。提取的血管应保留基础解剖结构的拓扑结构,将评估基于拓扑的指标的分割性能。该挑战旨在获得血管特征,以捕捉CoW 的基本拓扑结构和几何变异性。
一共有两个模态任务可以选择,一个是CTA模态,一个是MRA模态。
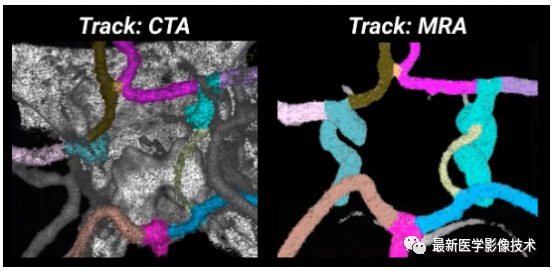
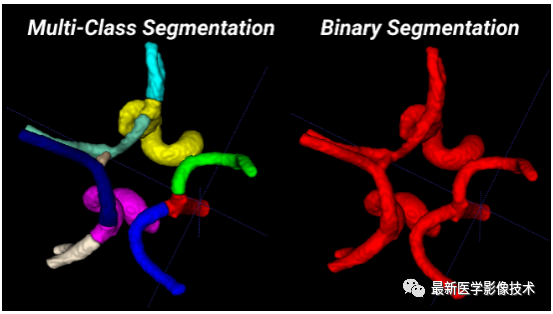
分割任务分为两个(子)任务:CoW 血管的二元分割和多类 CoW 血管解剖分割。
三、Top_Cow 2023数据集
挑战数据队列由2018年和2019年苏黎世大学医院 (USZ) 中风中心收治的患者组成。挑战数据的纳入标准是:1) MRA 和 CTA 扫描均适用于该患者;2) 至少 MRA 或 CTA 允许评估Cow的解剖结构和几何形状。挑战队列的患者是从中风相关的神经系统疾病中恢复,包括缺血性中风、短暂性脑缺血发作、拟中风、视网膜梗塞或一过性黑蒙、脑出血和脑窦静脉血栓形成。
使用的两种成像方式是:计算机断层扫描血管造影 (CTA) 和飞行时间磁共振血管造影 (TOF-MRA) 或 MRA。这些数据是在苏黎世大学医院 (USZ) 按照 MRA 和 CTA 标准程序进行例行检查期间获得的。MRA 扫描通常由 SIEMENS Skyra 模型或 Avanto Fit 模型获取,磁场强度为 3 特斯拉或 1.5 特斯拉,并使用 TOF-3D 模式或 TOF-3D 多平板模式。CTA 通常由 SIEMENS SOMATOM Definition Flash 使用 Dual Energy 获取。对于 CTA,体素大小在 XY 维度上的范围从大约 0.34 到 0.53 毫米,在 Z 维度上的范围从大约 0.62 到 0.75 毫米。对于 MRA,体素大小在 XY 维度上的范围从大约 0.29 到 0.35 毫米,在 Z 维度上的范围从大约 0.5 到 0.6 毫米。数据是匿名的(相关 DICOM 患者信息的删除和匿名化)。执行额外的去表面处理和裁剪程序以确保图像数据中的患者隐私。具体来说,我们屏蔽或剪切面部区域,然后裁剪图像数据以仅包含脑壳区域。
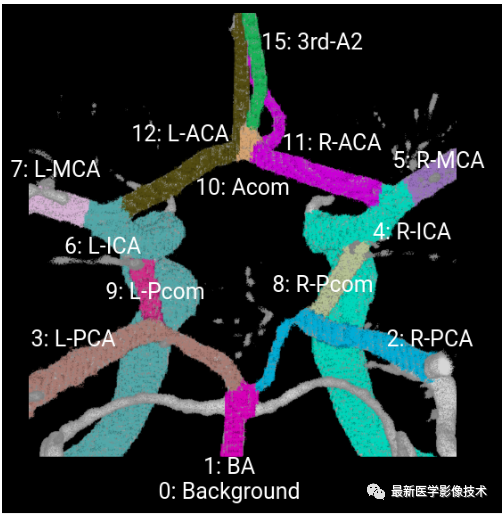
标注的CoW血管成分为左右颈内动脉(ICA)、左右大脑前动脉(ACA)、左右大脑中动脉(MCA)、前交通动脉(Acom)、左右后交通动脉动脉 (Pcom)、左右大脑后动脉 (PCA) 和基底动脉 (BA)。仅对诊断 CoW 血管结构和变体所需的血管成分和区域进行了注释。
最初的CoW注释由接受过临床专家的CoW解剖学教育的研究人员手动标记体素。注释者不确定的案例被标记后再发送给临床专家进一步验证和批准。临床专家团队由神经放射科医生、神经科医生和神经外科医生组成。关于如何在ACA-ICA-MCA、ACA-Acom、PCA-Pcom、Pcom-ICA等分叉点处分割血管成分的注释协议,由临床专家讨论并达成一致。
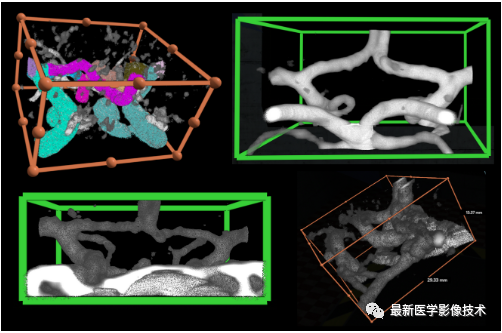
还提供了CoW感兴趣区域 (ROI) 注释了 3D 边界框。CoW ROI 定义为捕获 CoW 解剖变体的 3D 边界框。为所有训练案例发布带注释的 CoW ROI。请注意,分割结果的评估仅限于感兴趣的 CoW 区域。
训练、验证和测试用例都有 MRA 和 CTA 联合模态对,每个模态都有一次扫描。任务是在 MRA 或 CTA 中分割 CoW 血管(任务 1)和 CoW 区域(任务 2)上的解剖结构。
训练数据集:90 名患者(已发布图像和注释)
验证集:10 名患者(图像公开但没有注释)
测试集:30 名患者(未公开)
除了上述 TopCoW 挑战数据外,我们还发布了来自 CROWN 挑战的 20 个 MRA 用于训练,并有 10 个案例进行测试(总共 30 个 MRA)。
诊断 CoW 血管结构所需的所有 CoW 血管组件均按体素进行注释。对于不同的 CoW 血管段,多类标签包含不同的体素值——0:背景,1:BA,2:R-PCA,3:L-PCA,4:R-ICA,5:R-MCA,6:L-ICA,7:L-MCA,8:R-Pcom, 9:L-Pcom,10:Acom,11:R-ACA,12:L-ACA,15:3rd-A2
对于二进制分割任务,通过组合多类标签提供单独的二进制容器标签——0:背景,1:CoW血管
评价指标:体积指标:Dice系数和CoW血管的中心线-Dice。基于拓扑的度量:Betti 数值的误差,例如连通分量和圆孔。基于图形的指标:基于路口/地标的 F1 分数。
四、Docker部署CTA和MRA的Willis环的拓扑解剖结构分割模型
CTA和MRA的Willis环的拓扑解剖结构分割实现参考这篇文章Top_Cow 2023——用于CTA和MRA的Willis环的拓扑解剖结构分割。
1、在window下先安装docker安装程序,如果出现安装错误可以网上找一下解决方法,一般是安装补丁即可解决。
2、以CTA_Binary分割任务为例,docker build的配置文件
2.1、编写Dockerfile
ARG PYTORCH="1.12.1"
ARG CUDA="11.3"
ARG CUDNN="8"
FROM python:3.8.3
# Pull the docker image | 拉取镜像
FROM pytorch/pytorch:${PYTORCH}-cuda${CUDA}-cudnn${CUDNN}-devel
# only work on linux system
#FROM nvidia/cuda:11.4.3-cudnn8-devel-ubuntu18.04
# 安装所需的软件包
RUN apt-get update && apt-get install -y \
build-essential \
cmake \
git \
wget \
unzip \
yasm \
pkg-config \
libswscale-dev \
libtbb2 \
libjpeg-dev \
libpng-dev \
libtiff-dev \
libavformat-dev \
libpq-dev \
python3-pip \
libgl1-mesa-dev
# Create the user
RUN groupadd -r user && useradd -m --no-log-init -r -g user user
RUN mkdir -p /opt/app /input /output \
&& chown user:user /opt/app /input /output
USER user
WORKDIR /opt/app
ENV PATH="/home/user/.local/bin:${PATH}"
## Copy requirements
COPY --chown=user:user ./requirements.txt /opt/app/
## Install Python packages in Docker image
RUN pip3 install -r requirements.txt -i https://pypi.tuna.tsinghua.edu.cn/simple
## Copy all files
COPY --chown=user:user ./ /opt/app/
## Execute the inference command
ENTRYPOINT [ "python", "-m", "process" ]2.2、编写requirements.txt
# ===================================================================
# Do not change these packages !!!
# Since you are using a Docker image that already has the PyTorch library installed, there is no need to reinstall Torch.
# ===================================================================
arrow==1.2.3
binaryornot==0.4.4
build==0.10.0
certifi==2022.12.7
chardet==5.1.0
charset-normalizer==3.1.0
click==8.1.3
cookiecutter==2.1.1
idna==3.4
imageio[tifffile]==2.27.0
jinja2==3.1.2
jinja2-time==0.2.0
joblib==1.2.0
markupsafe==2.1.2
numpy==1.21.6
packaging==23.1
pandas==1.3.5
pillow==9.5.0
pip-tools==6.13.0
pyproject-hooks==1.0.0
python-dateutil==2.8.2
python-slugify==8.0.1
pytz==2023.3
pyyaml==6.0
requests==2.28.2
scikit-learn==1.0.2
scipy==1.7.3
simpleitk==2.2.1
six==1.16.0
text-unidecode==1.3
threadpoolctl==3.1.0
tifffile==2021.11.2
tomli==2.0.1
tzdata==2023.3
urllib3==1.26.15
wheel==0.40.0
scikit-image==0.19.3
evalutils==0.3.1
# ===================================================================
# If you have other additional dependencies, please list them below.
# ===================================================================
opencv-python==4.7.0.683、将运行脚本process.py与requirements.txt和Dockerfile放在同一目录下。
#!/usr/bin/env python3
# -*- coding: utf-8 -*-
import SimpleITK as sitk
import os
from inferencemodel import getbodyroi, GetLargestConnectedCompont, GetLargestConnectedCompontBoundingbox, \
MorphologicalOperation, getRangImageRange
from inferencemodel import BinaryVNet3dModel, MutilVNet3dModel
import numpy as np
from enum import Enum
class TRACK(Enum):
MR = "mr"
CT = "ct"
class TASK(Enum):
BINARY_SEGMENTATION = "bin_seg"
MULTICLASS_SEGMENTATION = "mul_seg"
def inferenceCTA(src):
newSize1 = (256, 256, 240)
newSize2 = (256, 256, 160)
# step1.get brain roi
binary_body_src = getbodyroi(src)
binary_body_src = GetLargestConnectedCompont(binary_body_src)
boundingbox = GetLargestConnectedCompontBoundingbox(binary_body_src)
x1, y1, z1, x2, y2, z2 = boundingbox[0], boundingbox[1], boundingbox[2], \
boundingbox[0] + boundingbox[3], boundingbox[1] + boundingbox[4], \
boundingbox[2] + boundingbox[5]
src_array = sitk.GetArrayFromImage(src)
roi_src_array = src_array[z1:z2, y1:y2, x1:x2]
roi_src = sitk.GetImageFromArray(roi_src_array)
roi_src.SetSpacing(src.GetSpacing())
roi_src.SetDirection(src.GetDirection())
roi_src.SetOrigin(src.GetOrigin())
# step2.binary segmentation
vnet3d = BinaryVNet3dModel(image_depth=240, image_height=256, image_width=256, image_channel=1, numclass=1,
batch_size=1, loss_name='BinaryCrossEntropyDiceLoss',
model_path=r"CTABinaryVNet3d.pth")
sitk_mask1 = vnet3d.inferenceCTA(roi_src, newSize1)
vnet3d.clear_GPU_cache()
roi_binary_array = sitk.GetArrayFromImage(sitk_mask1)
binary_array_vessels = np.zeros_like(src_array)
binary_array_vessels[z1:z2, y1:y2, x1:x2] = roi_binary_array[:, :, :]
binary_sitk_vessels = sitk.GetImageFromArray(binary_array_vessels)
binary_sitk_vessels = MorphologicalOperation(binary_sitk_vessels, 5, 'dilate')
binary_array_vessels = sitk.GetArrayFromImage(binary_sitk_vessels)
# step3.get binary vessel roi
z1, z2 = getRangImageRange(binary_array_vessels, 0)
y1, y2 = getRangImageRange(binary_array_vessels, 1)
x1, x2 = getRangImageRange(binary_array_vessels, 2)
roi_src_array = src_array[z1:z2, y1:y2, x1:x2]
roi_src = sitk.GetImageFromArray(roi_src_array)
roi_src.SetSpacing(src.GetSpacing())
roi_src.SetDirection(src.GetDirection())
roi_src.SetOrigin(src.GetOrigin())
# step4.segmentatio subregion
mutilVnet3d = MutilVNet3dModel(image_depth=160, image_height=256, image_width=256, image_channel=1, numclass=14,
batch_size=1, loss_name='MutilCrossEntropyDiceLoss',
model_path=r'CTAMutilVNet3dModel.pth')
sitk_mask2 = mutilVnet3d.inferenceCTA(roi_src, newSize2)
mutilVnet3d.clear_GPU_cache()
roi_mutil_array = sitk.GetArrayFromImage(sitk_mask2)
mutil_array_vessels = np.zeros_like(src_array)
mutil_array_vessels[z1:z2, y1:y2, x1:x2] = roi_mutil_array[:, :, :]
mutil_array_vessels[mutil_array_vessels == 13] = 15
mutil_mask_sitk = sitk.GetImageFromArray(mutil_array_vessels.astype('uint8'))
mutil_mask_sitk.SetSpacing(src.GetSpacing())
mutil_mask_sitk.SetDirection(src.GetDirection())
mutil_mask_sitk.SetOrigin(src.GetOrigin())
binary_array_vessels = mutil_array_vessels.copy()
binary_array_vessels[mutil_array_vessels == 0] = 0
binary_array_vessels[mutil_array_vessels != 0] = 1
binary_mask_sitk = sitk.GetImageFromArray(binary_array_vessels.astype('uint8'))
binary_mask_sitk.SetSpacing(src.GetSpacing())
binary_mask_sitk.SetDirection(src.GetDirection())
binary_mask_sitk.SetOrigin(src.GetOrigin())
return binary_mask_sitk, mutil_mask_sitk
def inferenceMRA(src):
newSize1 = (320, 320, 160)
newSize2 = (256, 256, 160)
# step1.get brain roi
binary_body_src = getbodyroi(src)
binary_body_src = GetLargestConnectedCompont(binary_body_src)
boundingbox = GetLargestConnectedCompontBoundingbox(binary_body_src)
x1, y1, z1, x2, y2, z2 = boundingbox[0], boundingbox[1], boundingbox[2], \
boundingbox[0] + boundingbox[3], boundingbox[1] + boundingbox[4], \
boundingbox[2] + boundingbox[5]
src_array = sitk.GetArrayFromImage(src)
roi_src_array = src_array[z1:z2, y1:y2, x1:x2]
roi_src = sitk.GetImageFromArray(roi_src_array)
roi_src.SetSpacing(src.GetSpacing())
roi_src.SetDirection(src.GetDirection())
roi_src.SetOrigin(src.GetOrigin())
# step2.binary segmentation
vnet3d = BinaryVNet3dModel(image_depth=160, image_height=320, image_width=320, image_channel=1, numclass=1,
batch_size=1, loss_name='BinaryCrossEntropyDiceLoss', model_path=r"MRABinaryVNet3d.pth")
sitk_mask1 = vnet3d.inferenceMRA(roi_src, newSize1)
vnet3d.clear_GPU_cache()
roi_binary_array = sitk.GetArrayFromImage(sitk_mask1)
binary_array_vessels = np.zeros_like(src_array)
binary_array_vessels[z1:z2, y1:y2, x1:x2] = roi_binary_array[:, :, :]
binary_sitk_vessels = sitk.GetImageFromArray(binary_array_vessels)
binary_sitk_vessels = MorphologicalOperation(binary_sitk_vessels, 5, 'dilate')
binary_array_vessels = sitk.GetArrayFromImage(binary_sitk_vessels)
# step3.get binary vessel roi
z1, z2 = getRangImageRange(binary_array_vessels, 0)
y1, y2 = getRangImageRange(binary_array_vessels, 1)
x1, x2 = getRangImageRange(binary_array_vessels, 2)
roi_src_array = src_array[z1:z2, y1:y2, x1:x2]
roi_src = sitk.GetImageFromArray(roi_src_array)
roi_src.SetSpacing(src.GetSpacing())
roi_src.SetDirection(src.GetDirection())
roi_src.SetOrigin(src.GetOrigin())
# step4.segmentatio subregion
mutilVnet3d = MutilVNet3dModel(image_depth=160, image_height=256, image_width=256, image_channel=1, numclass=14,
batch_size=1, loss_name='MutilCrossEntropyDiceLoss',
model_path=r'MRAMutilVNet3dModel.pth')
sitk_mask2 = mutilVnet3d.inferenceMRA(roi_src, newSize2)
mutilVnet3d.clear_GPU_cache()
roi_mutil_array = sitk.GetArrayFromImage(sitk_mask2)
mutil_array_vessels = np.zeros_like(src_array)
mutil_array_vessels[z1:z2, y1:y2, x1:x2] = roi_mutil_array[:, :, :]
mutil_array_vessels[mutil_array_vessels == 13] = 15
mutil_mask_sitk = sitk.GetImageFromArray(mutil_array_vessels.astype('uint8'))
mutil_mask_sitk.SetSpacing(src.GetSpacing())
mutil_mask_sitk.SetDirection(src.GetDirection())
mutil_mask_sitk.SetOrigin(src.GetOrigin())
binary_array_vessels = mutil_array_vessels.copy()
binary_array_vessels[mutil_array_vessels == 0] = 0
binary_array_vessels[mutil_array_vessels != 0] = 1
binary_mask_sitk = sitk.GetImageFromArray(binary_array_vessels.astype('uint8'))
binary_mask_sitk.SetSpacing(src.GetSpacing())
binary_mask_sitk.SetDirection(src.GetDirection())
binary_mask_sitk.SetOrigin(src.GetOrigin())
return binary_mask_sitk, mutil_mask_sitk
def inferenceDummy(main_input):
"""
dummy threshold segmentation
"""
stats = sitk.StatisticsImageFilter()
stats.Execute(main_input)
print("Main input max val: ", stats.GetMaximum())
segmentation = sitk.BinaryThreshold(
main_input,
lowerThreshold=stats.GetMaximum() // 3, # some arbitrary threshold
upperThreshold=stats.GetMaximum(),
)
return (segmentation, segmentation)
track = TRACK.CT
task = TASK.BINARY_SEGMENTATION
print("Track = ", track.value)
print("Task = ", task.value)
if task == TASK.MULTICLASS_SEGMENTATION:
task_output = "multiclass"
elif task == TASK.BINARY_SEGMENTATION:
task_output = "binary"
# NOTE: init the input and output dir with track and task
input_dir = f"/input/images/head-{track.value}-angio/"
output_dir = f"/output/images/cow-{task_output}-segmentation/"
# If output_dir doesn't exist, create it
if not os.path.exists(output_dir):
print(f"{output_dir} does not exist, will create it.")
os.makedirs(output_dir)
print("Path at terminal when executing this file")
print(os.getcwd() + "\n")
path_img = os.path.join(input_dir, '{}.mha')
path_pred = os.path.join(output_dir, '{}.mha')
list_case = [k.split('.')[0] for k in os.listdir(input_dir)]
for case in list_case:
src = sitk.ReadImage(path_img.format(case))
if track.value == "ct":
binary_mask_sitk, mutil_mask_sitk = inferenceCTA(src)
# save the segmentation mask
if task.value == "bin_seg":
sitk.WriteImage(binary_mask_sitk, path_pred.format(case), useCompression=True)
if task.value == "mul_seg":
sitk.WriteImage(mutil_mask_sitk, path_pred.format(case), useCompression=True)
if track.value == "mr":
binary_mask_sitk, mutil_mask_sitk = inferenceMRA(src)
# save the segmentation mask
if task.value == "bin_seg":
sitk.WriteImage(binary_mask_sitk, path_pred.format(case), useCompression=True)
if task.value == "mul_seg":
sitk.WriteImage(mutil_mask_sitk, path_pred.format(case), useCompression=True)
4、在命令行里运行如下命令编译模型并推理测试
#!/usr/bin/env bash
# If you intended to pass a host directory, use absolute path.
SCRIPTPATH="$( cd "$(dirname "$0")" ; pwd -P )"
echo "SCRIPTPATH = ${SCRIPTPATH}"
docker build -t junqiangmler_ctabinary "$SCRIPTPATH"
OUTPUT_VOL="topcow-test-docker-output"
echo "OUTPUT_VOL = ${OUTPUT_VOL}"
docker volume create ${OUTPUT_VOL}
# Do not change any of the parameters to docker run, these are fixed
# This is to mimic a restricted Grand-Challenge running environment
# ie no internet and no new privileges etc.
docker run --rm \
-v G:/MedicalData/2023Top_Cow/download/test/input/images/head-ct-angio:/input/images/head-ct-angio \
-v G:/MedicalData/2023Top_Cow/download/test/input/images/head-mr-angio:/input/images/head-mr-angio \
-v ${OUTPUT_VOL}:/output/ \
--gpus=all \
junqiangmler_ctabinary
5、在命令行里运行如下命令编译模型并保存模型
#!/usr/bin/env bash
SCRIPTPATH="$( cd "$(dirname "$0")" ; pwd -P )"
echo "SCRIPTPATH = ${SCRIPTPATH}"
docker build -t junqiangmler_ctabinary "$SCRIPTPATH"
docker save junqiangmler_ctabinary | xz -T0 -c > junqiangmler_ctabinary.tar.xz6、其他三个任务:CTA_Mutil分割部署,MRA_Binary分割部署,MRA_Mutil分割部署,只需要简单修改一下process.py中的track和task变量即可,例如CTA_Mutil任务修改如下,其他任务类似修改即可。
track = TRACK.CT
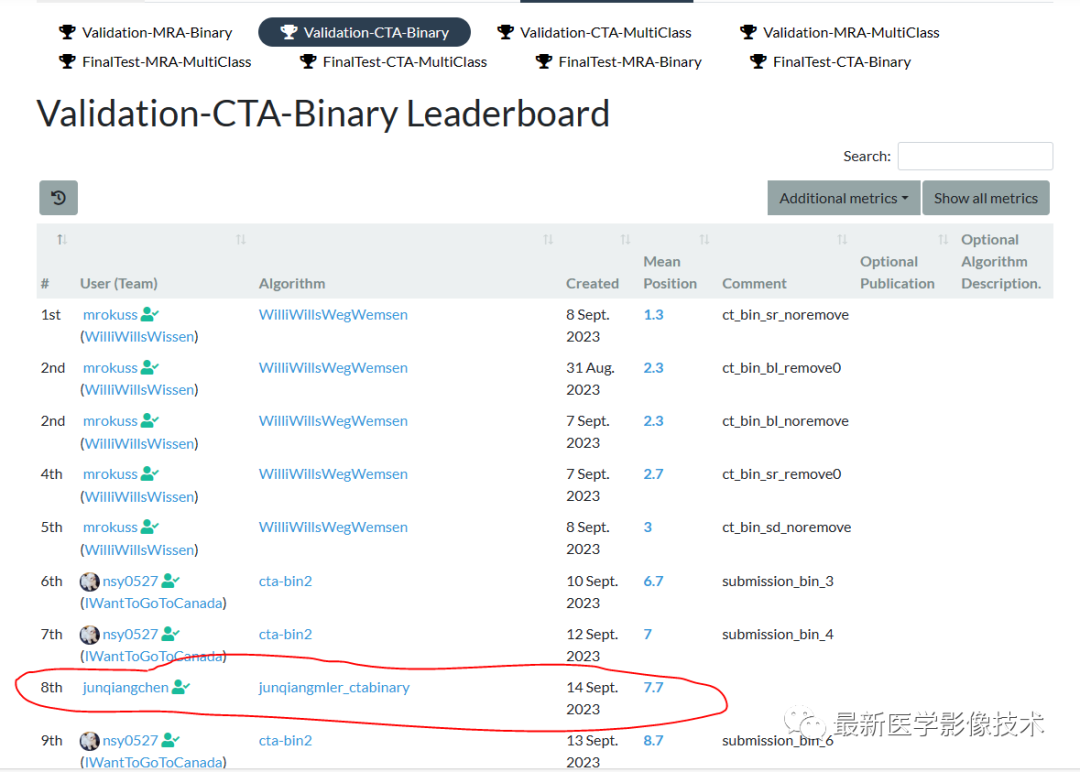
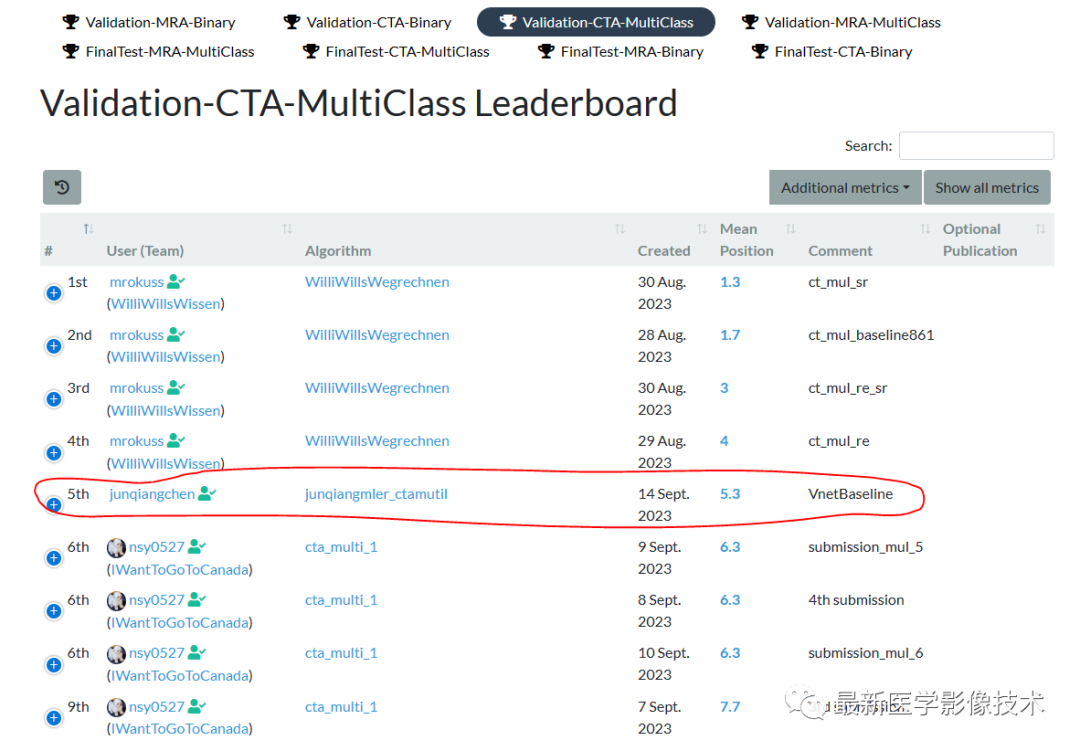
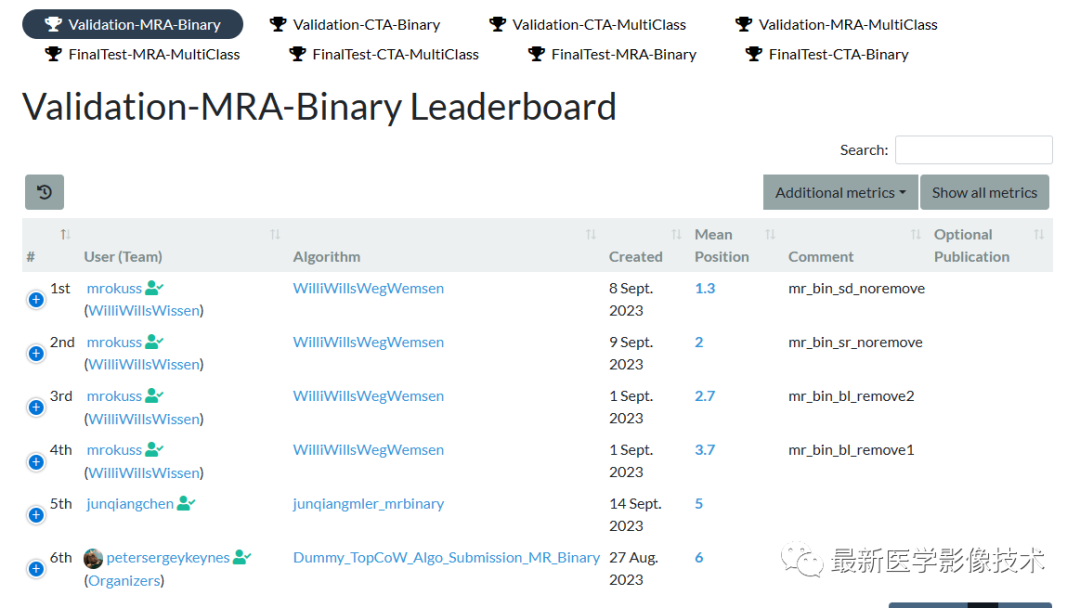
task = TASK.MULTICLASS_SEGMENTATION7、验证集结果
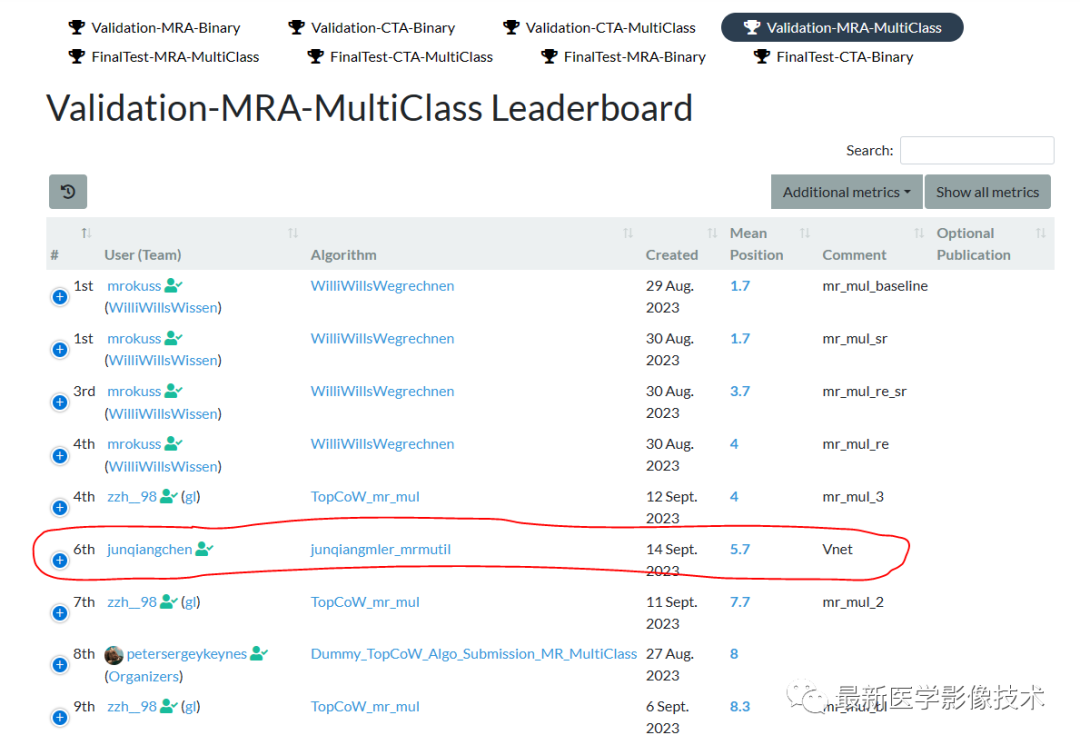
如果大家觉得这个项目还不错,希望大家给个Star并Fork,可以让更多的人学习。如果有任何问题,随时给我留言我会及时回复的。
