空间转录组数据分析之空间基因梯度(STG)
原创空间转录组数据分析之空间基因梯度(STG)
原创
追风少年i
发布于 2024-03-24 11:36:09
发布于 2024-03-24 11:36:09
作者,Evil Genius
太原的天气还是有点冷~~~~
今天我们要实现的目标
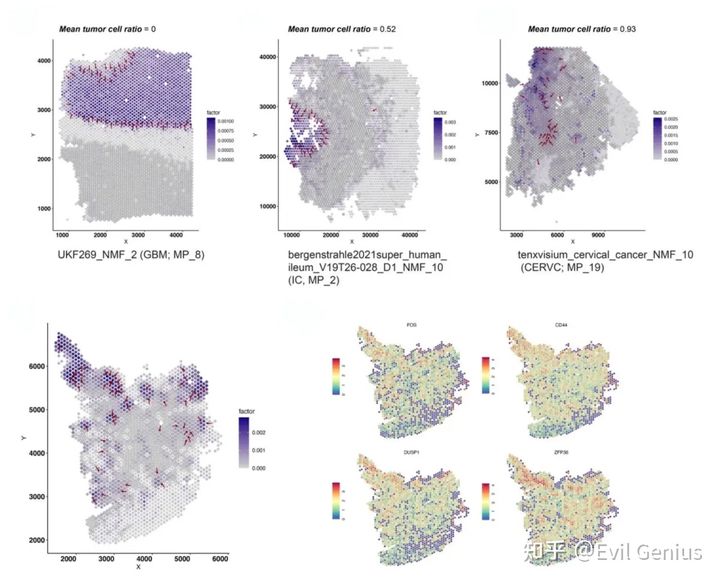
Spatial relationship between gradients and tumor boundary
实现的效果
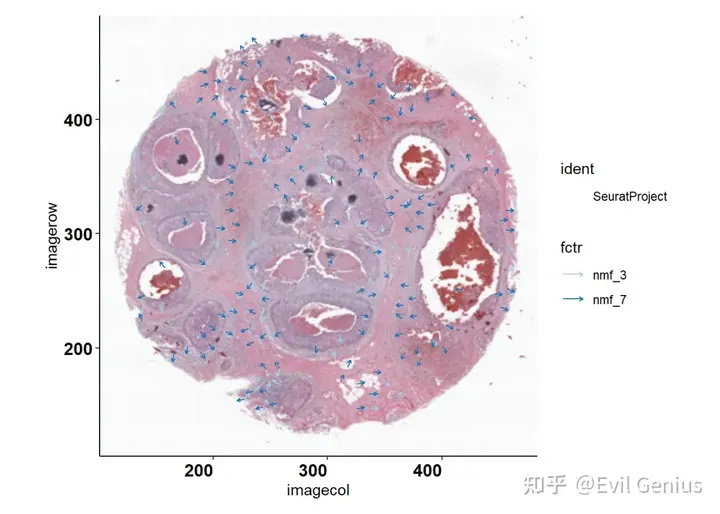
方法可以魔改,我们可以看基因、细胞、通路的空间等级
看起来和之前分享的空间向量场差不多,但是还是有很大的区别。
关于NMF也分享了很多,
借助NMF的力量对单细胞RNA和单细胞ATAC进行联合分析
10X单细胞(10X空间转录组)数据分析之Consensus Non-negative Matrix factorization (cNMF)
10X单细胞(10X空间转录组)分析回顾之harmony的各种运用(联合NMF和python的harmonypy)
10X单细胞(10X空间转录组)分析之寻找目标bases基因集(factors)(PNMF)
10X单细胞(10X空间转录组)数据分析之NMF寻找转录programs
10X单细胞(10X空间转录组)数据分析之主成分分析(PCA)与因子分析(NMF)
10X单细胞(10X空间转录组)数据分析总结之各种NMF
10X单细胞(10X空间转录组)之NMF的实际运用示例(探索肿瘤特征)
10X单细胞(10X空间转录组)数据分析之NMF(非负矩阵分解)
先来学学基础知识
细胞组成和信号传导在不同的生态位中有所不同,这可以诱导细胞亚群中基因表达的梯度。这种空间转录组梯度(STG)是肿瘤内异质性的重要来源,可以影响肿瘤的侵袭、进展和对治疗的反应。
肿瘤组织包含异质性细胞群,在复杂的细胞微环境中具有不同的转录、遗传和表观遗传特征。解剖这种多因素的肿瘤内异质性(ITH)是了解肿瘤发生、转移和治疗耐药性的基础。细胞中转录变异的一个来源是它们的微环境,微环境通过不同的方式塑造基因表达,如细胞间通讯(如配体受体信号)或局部信号提示(如pH值、氧、代谢物)。因此,一些细胞会随着它们的空间定位而表现出渐变的转录变异,这被称为“空间转录组梯度”(STG)。
实现的目标:同时检测到STGs的存在和方向
方法原理:应用NMF从ST数据的基因表达矩阵中获得定量的、可解释的细胞表型,同时检测每个生态位中线性空间梯度的存在和方向。
分析框架
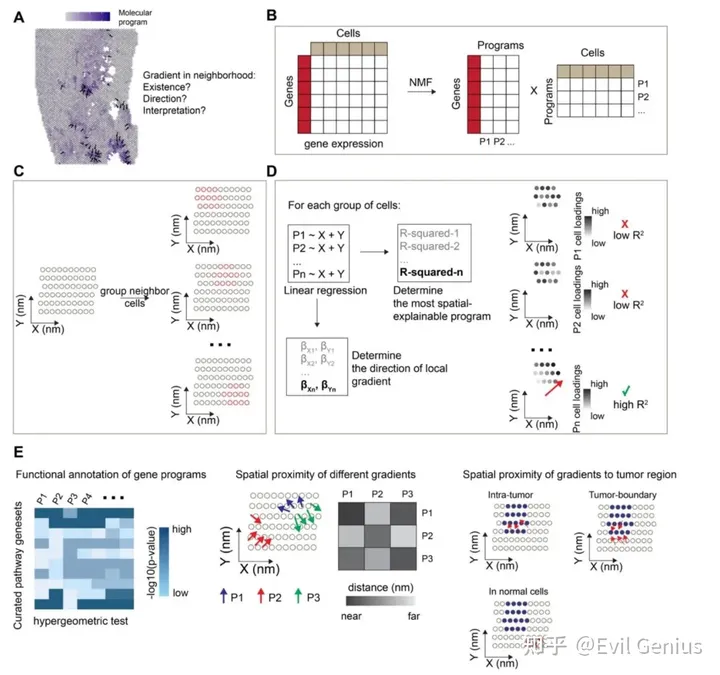
The LSGI framework and downstream analysis
三个需要回答的生物学问题
- 1、空间基因梯度的位置
- 2、空间基因梯度的方向性
- 3、空间基因梯度的生物学功能
为了实现目标,利用NMF将ST数据中所有细胞或SPOT的基因表达谱分解成多个因子,包括描述细胞组成和调节细胞表型。通过这一步,计算 cell loadings and gene loadings ,分别表明program在细胞/spot水平上的活性和program在基因水平上的属性。
关于空间的数据分析采用slide-window strategy ,在此基础上,cells/spots在overlapping windows中按空间定位分组,然后,使用空间坐标作为预测因子,并将细胞NMF loadings作为目标,对每个NMF program和每组细胞拟合线性模型。使用r平方来评价拟合优度,较大的值表示存在STG。梯度的方向由相应的回归系数决定。这些步骤创建了一个map,其中包含STG的定位和方向,以及它们在一个或多个NMF program中的分配。然后,利用精选的功能基因集,通过统计方法(例如,超几何测试)对program进行功能性注释。并研究在肿瘤ST数据集中,分配给不同程序的梯度的空间关系,或梯度到肿瘤-TME边界的空间关系。
来看看示例
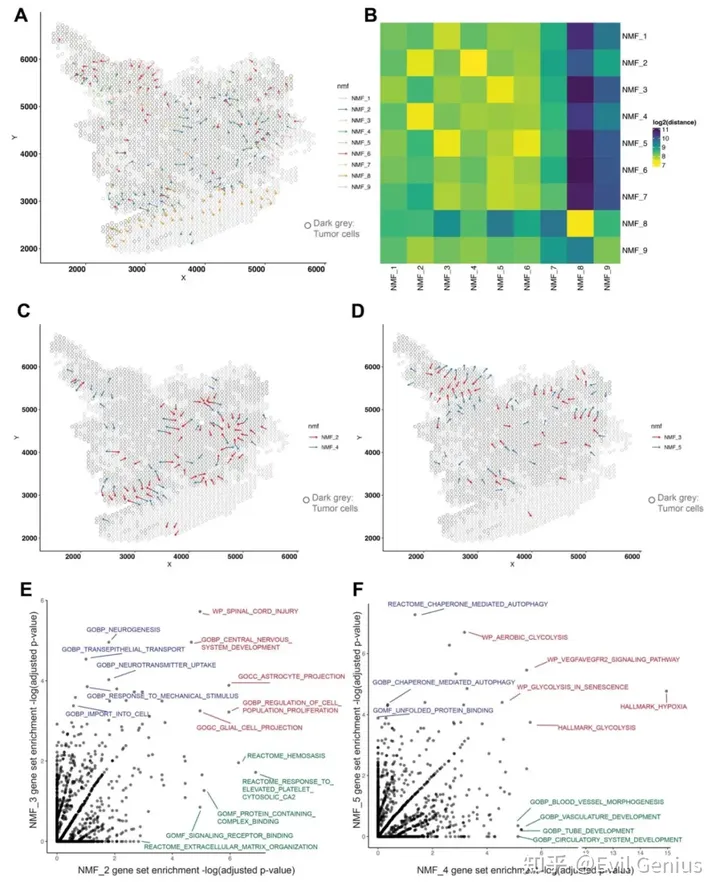
Application of LSGI on single ST dataset
最后看看实现方法,R语言的代码
library(Seurat)
library(Matrix)
library(RcppML)
library(ggplot2)
library(dplyr)
library(LSGI)10X的数据,当然其他类型的空间数据均可以用
img <- Read10X_Image(image.dir = "C:/Users/liang/work/42_LSGI/LSGI.test/Visium_FFPE_Human_Breast_Cancer_spatial/",
image.name = "tissue_lowres_image.png", filter.matrix = TRUE)
data <- Load10X_Spatial(data.dir = "C:/Users/liang/work/42_LSGI/LSGI.test/",
filename = "Visium_FFPE_Human_Breast_Cancer_filtered_feature_bc_matrix.h5",
assay = "RNA", slice = "slice1", filter.matrix = TRUE, to.upper = FALSE,
image = img)
data <- NormalizeData(data)使用NMF将ST数据中所有细胞或SPOT的基因表达谱汇总到多个program中。
Run NMF(类似PCA)
# define functions as below some code adapted from this
# preprint:
# https://www.biorxiv.org/content/10.1101/2021.09.01.458620v1.full
scan.nmf.mse <- function(obj, ranks = seq(1, 30, 2), tol = 1e-04) {
# users can customize the scan by changing 'ranks'
dat <- obj@assays$RNA@data
errors <- c()
ranks <- seq(1, 30, 2)
for (i in ranks) {
# cat('rank: ', i, '\n')
mod <- RcppML::nmf(dat, i, tol = 1e-04, verbose = F)
mse_i <- mse(dat, mod$w, mod$d, mod$h)
errors <- c(errors, mse_i)
}
results <- data.frame(rank = ranks, MSE = errors)
return(results)
}
sr.nmf <- function(obj, k = 10, tol = 1e-06, assay = "RNA") {
dat <- obj@assays$RNA@data
nmf_model <- RcppML::nmf(dat, k = k, tol = tol, verbose = F)
embeddings <- t(nmf_model$h)
rownames(embeddings) <- colnames(obj)
colnames(embeddings) <- paste0("nmf_", 1:k)
loadings <- nmf_model$w
rownames(loadings) <- rownames(obj)
obj@reductions$nmf <- CreateDimReducObject(embeddings = embeddings,
loadings = loadings, key = "nmf_", assay = assay)
return(obj)
}
scan.nmf.res <- scan.nmf.mse(obj = data)
ggplot(scan.nmf.res, aes(x = rank, y = MSE)) + geom_point(size = 0.7) +
geom_smooth(method = "loess", span = 0.2, color = "black",
linewidth = 1, se = F) + labs(x = "NMF rank", y = "MSE") +
theme_classic() + scale_y_continuous(expand = c(0.01, 0)) +
theme(aspect.ratio = 1)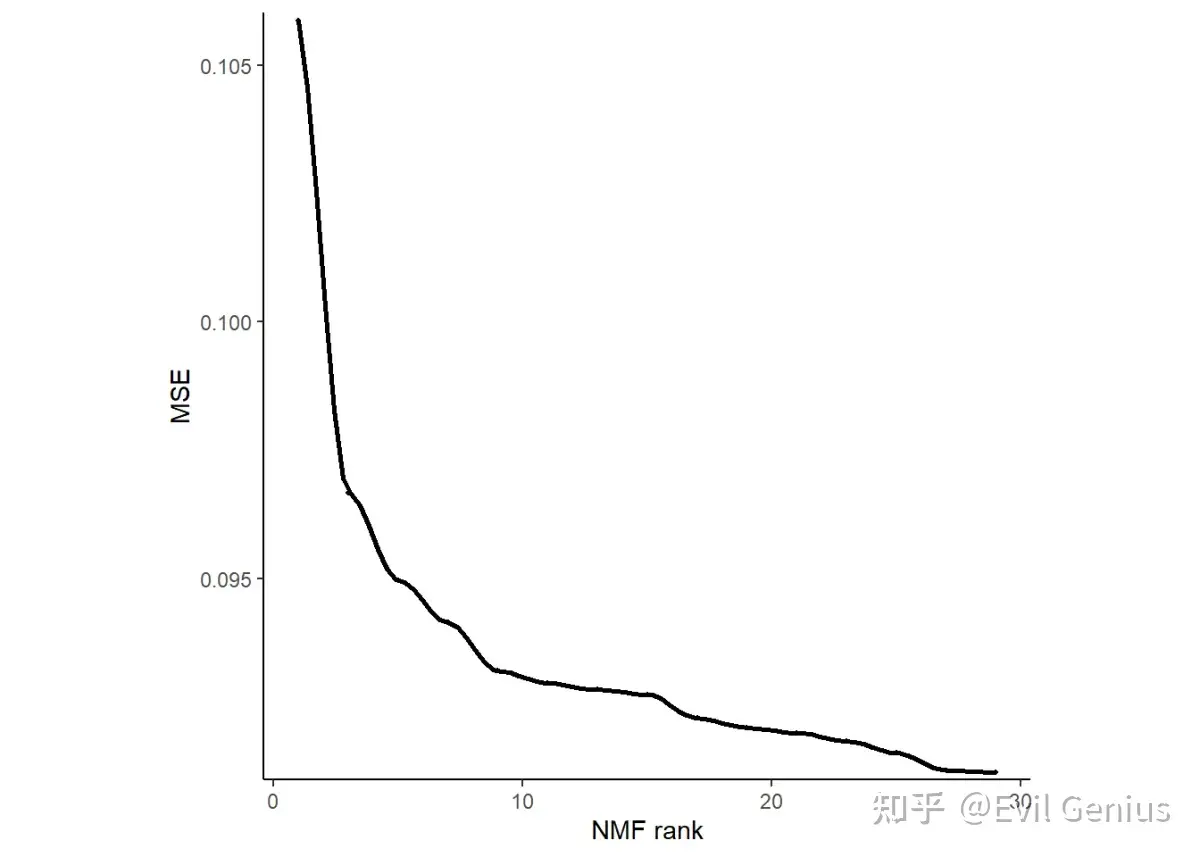
Prepare input data for LSGI
# LSGI requires two inputs: spatial_coords and embeddings
# In the current version, we require the spatial_coords
# have colnames as 'X', and 'Y'.
spatial_coords <- data@images$slice1@coordinates[, c(4, 5)]
colnames(spatial_coords) <- c("X", "Y")
print(head(spatial_coords))
#> X Y
#> AAACAAGTATCTCCCA-1 16265 19934
#> AAACACCAATAACTGC-1 18526 7893
#> AAACAGAGCGACTCCT-1 7178 18782
#> AAACAGCTTTCAGAAG-1 14487 6446
#> AAACAGGGTCTATATT-1 15497 7025
#> AAACCGGGTAGGTACC-1 14237 9202
# row names of embeddings are cell/spot names the row names
# of embeddings and spatial_coords should be the same (in
# the same order as well) here
embeddings <- data@reductions$nmf@cell.embeddings
print(embeddings[1:5, 1:5])
#> nmf_1 nmf_2 nmf_3 nmf_4
#> AAACAAGTATCTCCCA-1 1.796119e-03 7.531603e-05 5.608306e-05 4.609495e-04
#> AAACACCAATAACTGC-1 2.799021e-05 2.456455e-04 8.588333e-04 7.773138e-04
#> AAACAGAGCGACTCCT-1 4.259168e-04 4.443754e-04 1.912114e-04 0.000000e+00
#> AAACAGCTTTCAGAAG-1 0.000000e+00 1.527858e-04 2.645159e-04 1.147089e-03
#> AAACAGGGTCTATATT-1 6.634297e-05 0.000000e+00 9.323769e-04 3.925687e-05
#> nmf_5
#> AAACAAGTATCTCCCA-1 1.404469e-04
#> AAACACCAATAACTGC-1 0.000000e+00
#> AAACAGAGCGACTCCT-1 5.804265e-04
#> AAACAGCTTTCAGAAG-1 0.000000e+00
#> AAACAGGGTCTATATT-1 3.575715e-05计算空间基因等级
# n.grids.scale: LSGI calculate spatial gradients in
# multiple small neighborhoods (centered in the 'grid
# point'), and this n.grid.scale decide the number of this
# type of neighborhood. The number of neighborhoods equals
# to (total number of cells)/n.grids.scales
# n.cells.per.meta: number of cells/spots for each
# neighborhood
lsgi.res <- local.traj.preprocessing(spatial_coords = spatial_coords,
n.grids.scale = 5, embeddings = embeddings, n.cells.per.meta = 25)可视化
# plot multiple factors
plt.factors.gradient.ind(info = lsgi.res, r_squared_thresh = 0.6,
minimum.fctr = 10) # plot gradient (had to appear in at least 10 grids)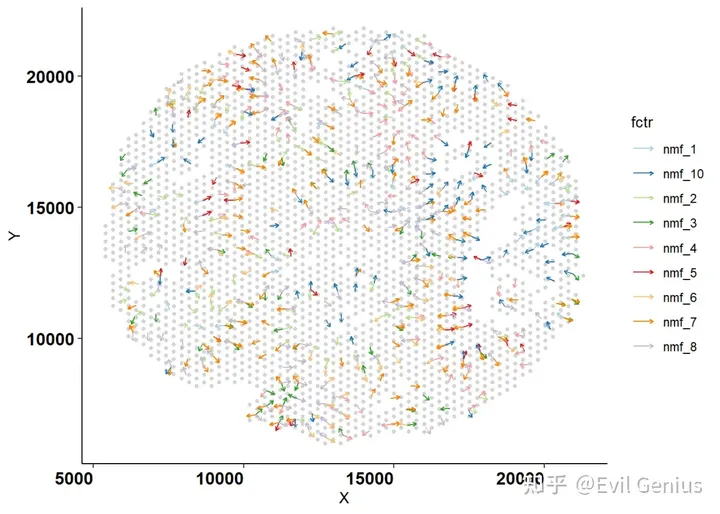
plt.factors.gradient.ind(info = lsgi.res, r_squared_thresh = 0.6,
sel.factors = c("nmf_5", "nmf_6", "nmf_8"), minimum.fctr = 10) # plot selected gradients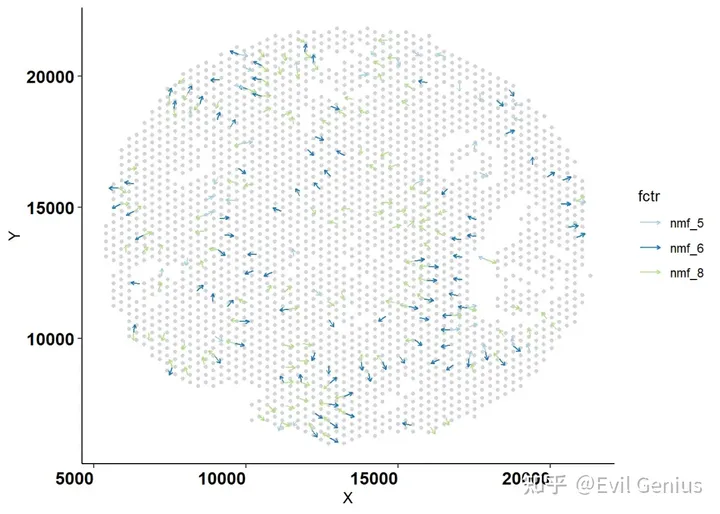
Plot single gradient with NMF loadings
# plot individual factor together with the NMF loadings
plt.factor.gradient.ind(info = lsgi.res, fctr = "nmf_1", r_squared_thresh = 0.6)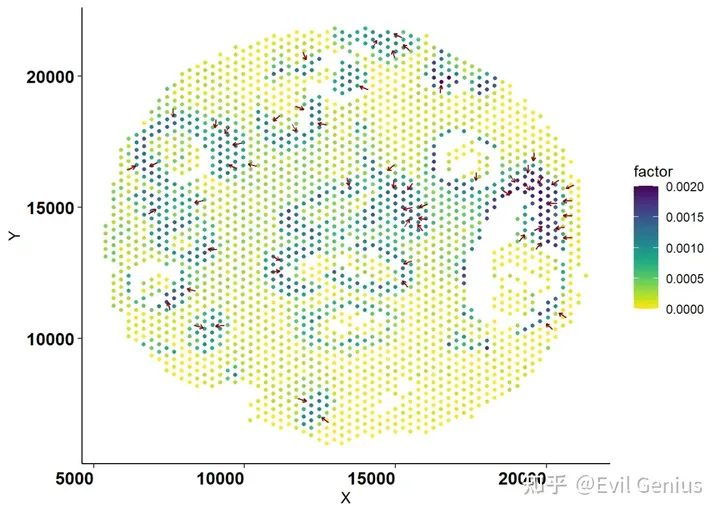
Distance analysis
dist.mat <- avg.dist.calc(info = lsgi.res, minimum.fctr = 10) # calculate average distance between NMF gradients
plt.dist.heat(dist.mat) # plot distance heatmap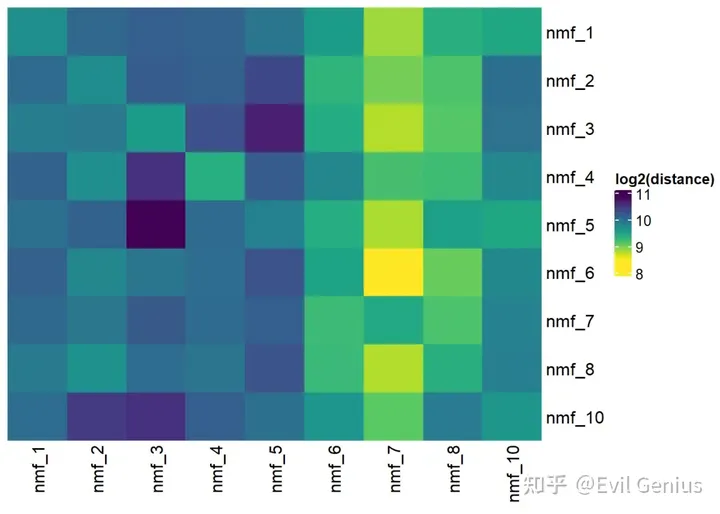
功能注释
# this can be done in the same way of NMF factor annotation
# there are different ways of doing this analysis, here we
# use hypergeometric test with top 50 genes in each NMF
# (top loadings) here we only use hallmark gene sets as a
# brief example the nmf information can be fetched from the
# Seurat object
get.nmf.info <- function(obj, top.n = 50) {
feature.loadings <- as.data.frame(obj@reductions$nmf@feature.loadings)
top.gene.list <- list()
for (i in 1:ncol(feature.loadings)) {
o <- order(feature.loadings[, i], decreasing = T)[1:top.n]
features <- rownames(feature.loadings)[o]
top.gene.list[[colnames(feature.loadings)[i]]] <- features
}
nmf.info <- list(feature.loadings = feature.loadings, top.genes = top.gene.list)
return(nmf.info)
}
nmf_info <- get.nmf.info(data)
str(nmf_info) # show the structure of nmf information extracted from the Seurat object after running NMF
#> List of 2
#> $ feature.loadings:'data.frame': 17943 obs. of 10 variables:
#> ..$ nmf_1 : num [1:17943] 0.00 5.40e-05 1.51e-05 2.56e-05 0.00 ...
#> ..$ nmf_2 : num [1:17943] 0.00 9.84e-05 8.26e-06 1.75e-05 0.00 ...
#> ..$ nmf_3 : num [1:17943] 7.65e-07 7.10e-05 2.20e-05 1.02e-05 0.00 ...
#> ..$ nmf_4 : num [1:17943] 0.00 4.47e-05 1.29e-05 3.60e-06 0.00 ...
#> ..$ nmf_5 : num [1:17943] 0.00 7.60e-05 2.17e-05 9.68e-06 0.00 ...
#> ..$ nmf_6 : num [1:17943] 3.83e-05 1.42e-05 2.75e-05 1.44e-05 0.00 ...
#> ..$ nmf_7 : num [1:17943] 1.75e-05 2.82e-05 1.85e-05 0.00 2.30e-06 ...
#> ..$ nmf_8 : num [1:17943] 0.00 2.83e-05 0.00 1.61e-05 1.45e-06 ...
#> ..$ nmf_9 : num [1:17943] 9.19e-06 3.53e-05 2.34e-05 0.00 0.00 ...
#> ..$ nmf_10: num [1:17943] 0.00 6.52e-05 0.00 8.09e-06 0.00 ...
#> $ top.genes :List of 10
#> ..$ nmf_1 : chr [1:50] "FGB" "FTH1" "LTF" "PABPC1" ...
#> ..$ nmf_2 : chr [1:50] "MUCL1" "FTH1" "AZGP1" "TMSB4X" ...
#> ..$ nmf_3 : chr [1:50] "CD74" "TMSB4X" "B2M" "HLA-DRA" ...
#> ..$ nmf_4 : chr [1:50] "FTL" "APOE" "APOC1" "CTSD" ...
#> ..$ nmf_5 : chr [1:50] "COL1A1" "POSTN" "COL1A2" "SPARC" ...
#> ..$ nmf_6 : chr [1:50] "COL1A1" "COL3A1" "COL1A2" "SPARC" ...
#> ..$ nmf_7 : chr [1:50] "IGKC" "IGHG2" "IGHA1" "IGLC1" ...
#> ..$ nmf_8 : chr [1:50] "IFI6" "MUCL1" "ISG15" "IFITM3" ...
#> ..$ nmf_9 : chr [1:50] "IGFBP7" "VWF" "AQP1" "HSPG2" ...
#> ..$ nmf_10: chr [1:50] "FTH1" "FTL" "TMSB4X" "IGKC" ...
# obtain gene sets
library(msigdbr)
library(hypeR)
mdb_h <- msigdbr(species = "Homo sapiens", category = "H")
gene.set.list <- list()
for (gene.set.name in unique(mdb_h$gs_name)) {
gene.set.list[[gene.set.name]] <- mdb_h[mdb_h$gs_name %in%
gene.set.name, ]$gene_symbol
}
# run hypeR test
mhyp <- hypeR(signature = nmf_info$top.genes, genesets = gene.set.list,
test = "hypergeometric", background = rownames(nmf_info[["feature.loadings"]]))
hyper.data <- mhyp$data
hyper.res.list <- list()
for (nmf.name in names(hyper.data)) {
res <- as.data.frame(hyper.data[[nmf.name]]$data)
hyper.res.list[[nmf.name]] <- res
}
print(head(hyper.res.list[[1]])) # here we output part of the NMF_1 annotation result
#> label pval
#> HALLMARK_COMPLEMENT HALLMARK_COMPLEMENT 8.5e-07
#> HALLMARK_APOPTOSIS HALLMARK_APOPTOSIS 7.0e-05
#> HALLMARK_INTERFERON_GAMMA_RESPONSE HALLMARK_INTERFERON_GAMMA_RESPONSE 2.0e-04
#> HALLMARK_COAGULATION HALLMARK_COAGULATION 4.7e-04
#> HALLMARK_ESTROGEN_RESPONSE_LATE HALLMARK_ESTROGEN_RESPONSE_LATE 2.0e-03
#> HALLMARK_ANDROGEN_RESPONSE HALLMARK_ANDROGEN_RESPONSE 2.3e-03
#> fdr signature geneset overlap background
#> HALLMARK_COMPLEMENT 4.2e-05 50 188 7 17943
#> HALLMARK_APOPTOSIS 1.7e-03 50 155 5 17943
#> HALLMARK_INTERFERON_GAMMA_RESPONSE 3.3e-03 50 193 5 17943
#> HALLMARK_COAGULATION 5.9e-03 50 130 4 17943
#> HALLMARK_ESTROGEN_RESPONSE_LATE 1.9e-02 50 192 4 17943
#> HALLMARK_ANDROGEN_RESPONSE 1.9e-02 50 94 3 17943
#> hits
#> HALLMARK_COMPLEMENT CFB,CLU,CP,CTSD,FN1,LTF,S100A9
#> HALLMARK_APOPTOSIS APP,CLU,ERBB2,SOD2,SQSTM1
#> HALLMARK_INTERFERON_GAMMA_RESPONSE B2M,CFB,NAMPT,SOD2,TAPBP
#> HALLMARK_COAGULATION CFB,CLU,FGG,FN1
#> HALLMARK_ESTROGEN_RESPONSE_LATE LSR,LTF,S100A9,XBP1
#> HALLMARK_ANDROGEN_RESPONSE AZGP1,B2M,KRT8
# Visualize annotation results
ggplot(hyper.res.list[[1]][1:5, ], aes(x = reorder(label, -log10(fdr)),
y = overlap/signature, fill = -log10(fdr))) + geom_bar(stat = "identity",
show.legend = T) + xlab("Gene Set") + ylab("Gene Ratio") +
viridis::scale_fill_viridis() + theme_classic() + coord_flip() +
theme(axis.text.x = element_text(color = "black", size = 12,
angle = 45, hjust = 1), axis.text.y = element_text(color = "black",
size = 8, angle = 0), panel.border = element_rect(colour = "black",
fill = NA, size = 1))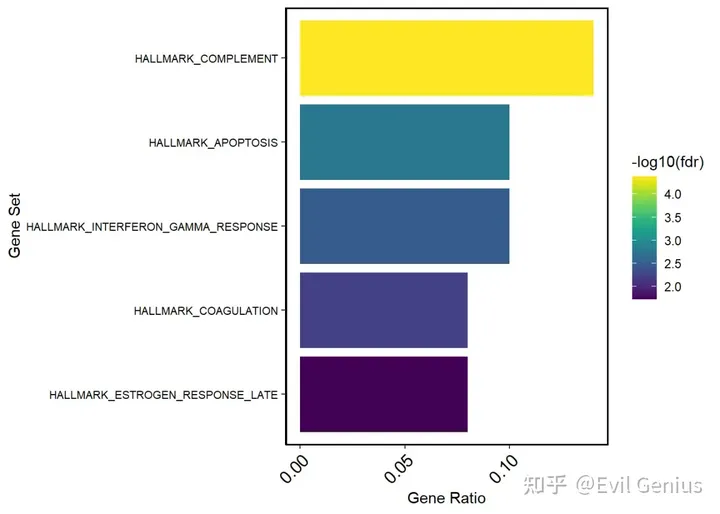
加载底片
# Finally, for Visium data only, we have the following
# functions that can plot the gradient superimposed with HE
# image
library(magick)
plot.overlay.factor <- function(object, info, sel.factors = NULL,
r_squared_thresh = 0.6, minimum.fctr = 20) {
scf <- object@images[["slice1"]]@scale.factors[["lowres"]]
object <- subset(object, cells = rownames(info$spatial_coords))
print(identical(rownames(object@meta.data), rownames(info$spatial_coords)))
object <- rotateSeuratImage(object, rotation = "L90")
object@meta.data <- cbind(object@meta.data, info$embeddings)
lin.res.df <- get.ind.rsqrs(info)
lin.res.df <- na.omit(lin.res.df)
lin.res.df <- lin.res.df[lin.res.df$rsquared > r_squared_thresh,
]
if (!is.null(sel.factors)) {
lin.res.df <- lin.res.df[lin.res.df$fctr %in% sel.factors,
]
}
lin.res.df <- lin.res.df %>%
group_by(fctr) %>%
filter(n() >= minimum.fctr) %>%
ungroup()
spatial_coords <- info$spatial_coords
spatial_coords$X <- spatial_coords$X * scf
spatial_coords$Y <- spatial_coords$Y * scf
lin.res.df$Xend <- lin.res.df$X + lin.res.df$vx.u
lin.res.df$Yend <- lin.res.df$Y + lin.res.df$vy.u
lin.res.df$X <- lin.res.df$X * scf
lin.res.df$Xend <- lin.res.df$Xend * scf
lin.res.df$Y <- lin.res.df$Y * scf
lin.res.df$Yend <- lin.res.df$Yend * scf
p <- SpatialFeaturePlot(object, features = NULL, alpha = c(0)) +
NoLegend() + geom_segment(data = as.data.frame(lin.res.df),
aes(x = X, y = Y, xend = Xend, yend = Yend, color = fctr,
fill = NULL), linewidth = 0.4, arrow = arrow(length = unit(0.1,
"cm"))) + scale_color_brewer(palette = "Paired") +
# scale_fill_gradient(low='lightgrey', high='navy')
# +
theme_classic() + theme(axis.text.x = element_text(face = "bold",
color = "black", size = 12, angle = 0, hjust = 1), axis.text.y = element_text(face = "bold",
color = "black", size = 12, angle = 0))
return(p)
}
# Adapted from this link:
# https://github.com/satijalab/seurat/issues/2702#issuecomment-1626010475
rotateSeuratImage <- function(seuratVisumObject, slide = "slice1",
rotation = "Vf") {
if (!(rotation %in% c("180", "Hf", "Vf", "L90", "R90"))) {
cat("Rotation should be either 180, L90, R90, Hf or Vf\n")
return(NULL)
} else {
seurat.visium <- seuratVisumObject
ori.array <- (seurat.visium@images)[[slide]]@image
img.dim <- dim(ori.array)[1:2]/(seurat.visium@images)[[slide]]@scale.factors$lowres
new.mx <- c()
# transform the image array
for (rgb_idx in 1:3) {
each.mx <- ori.array[, , rgb_idx]
each.mx.trans <- rotimat(each.mx, rotation)
new.mx <- c(new.mx, list(each.mx.trans))
}
# construct new rgb image array
new.X.dim <- dim(each.mx.trans)[1]
new.Y.dim <- dim(each.mx.trans)[2]
new.array <- array(c(new.mx[[1]], new.mx[[2]], new.mx[[3]]),
dim = c(new.X.dim, new.Y.dim, 3))
# swap old image with new image
seurat.visium@images[[slide]]@image <- new.array
## step4: change the tissue pixel-spot index
img.index <- (seurat.visium@images)[[slide]]@coordinates
# swap index
if (rotation == "Hf") {
seurat.visium@images[[slide]]@coordinates$imagecol <- img.dim[2] -
img.index$imagecol
}
if (rotation == "Vf") {
seurat.visium@images[[slide]]@coordinates$imagerow <- img.dim[1] -
img.index$imagerow
}
if (rotation == "180") {
seurat.visium@images[[slide]]@coordinates$imagerow <- img.dim[1] -
img.index$imagerow
seurat.visium@images[[slide]]@coordinates$imagecol <- img.dim[2] -
img.index$imagecol
}
if (rotation == "L90") {
seurat.visium@images[[slide]]@coordinates$imagerow <- img.dim[2] -
img.index$imagecol
seurat.visium@images[[slide]]@coordinates$imagecol <- img.index$imagerow
}
if (rotation == "R90") {
seurat.visium@images[[slide]]@coordinates$imagerow <- img.index$imagecol
seurat.visium@images[[slide]]@coordinates$imagecol <- img.dim[1] -
img.index$imagerow
}
return(seurat.visium)
}
}
rotimat <- function(foo, rotation) {
if (!is.matrix(foo)) {
cat("Input is not a matrix")
return(foo)
}
if (!(rotation %in% c("180", "Hf", "Vf", "R90", "L90"))) {
cat("Rotation should be either L90, R90, 180, Hf or Vf\n")
return(foo)
}
if (rotation == "180") {
foo <- foo %>%
.[, dim(.)[2]:1] %>%
.[dim(.)[1]:1, ]
}
if (rotation == "Hf") {
foo <- foo %>%
.[, dim(.)[2]:1]
}
if (rotation == "Vf") {
foo <- foo %>%
.[dim(.)[1]:1, ]
}
if (rotation == "L90") {
foo = t(foo)
foo <- foo %>%
.[dim(.)[1]:1, ]
}
if (rotation == "R90") {
foo = t(foo)
foo <- foo %>%
.[, dim(.)[2]:1]
}
return(foo)
}# then run
plot.overlay.factor(object = data, info = lsgi.res, sel.factors = NULL)
#> [1] TRUE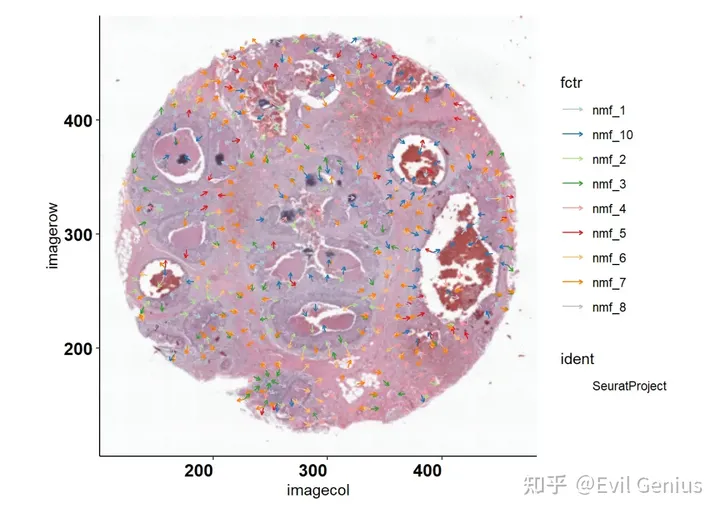
# or plot any selected factors
plot.overlay.factor(object = data, info = lsgi.res, sel.factors = c("nmf_3",
"nmf_7"))
#> [1] TRUE
参考地址Interpretable Spatial Gradient Analysis for Spatial Transcriptomics Data
参考地址LSTG
生活很好,有你更好
原创声明:本文系作者授权腾讯云开发者社区发表,未经许可,不得转载。
如有侵权,请联系 cloudcommunity@tencent.com 删除。
原创声明:本文系作者授权腾讯云开发者社区发表,未经许可,不得转载。
如有侵权,请联系 cloudcommunity@tencent.com 删除。
评论
登录后参与评论
推荐阅读
目录