技能树Day01_R环境配置_直播课day01
原创技能树Day01_R环境配置
1 R环境配置
1.1 代码
(原网址http://www.bio-info-trainee.com/3727.html)
rm(list = ls())
options()$repos
options()$BioC_mirror
#options(BioC_mirror="https://mirrors.ustc.edu.cn/bioc/")
options(BioC_mirror="http://mirrors.tuna.tsinghua.edu.cn/bioconductor/")
options("repos" = c(CRAN="https://mirrors.tuna.tsinghua.edu.cn/CRAN/"))
options()$repos
options()$BioC_mirror
# https://bioconductor.org/packages/release/bioc/html/GEOquery.html
if (!requireNamespace("BiocManager", quietly = TRUE))
install.packages("BiocManager")
BiocManager::install(c("GSEABase","GSVA","clusterProfiler" ),ask = F,update = F)
BiocManager::install(c("GEOquery","limma","impute" ),ask = F,update = F)
BiocManager::install(c("org.Hs.eg.db","hgu133plus2.db" ),ask = F,update = F)
# 下面代码被我注释了,意思是这些代码不需要运行,因为它过时了,很多旧教程就忽略
# 在代码前面加上 # 这个符号,代码代码被注释,意思是不会被运行
# source("https://bioconductor.org/biocLite.R")
# library('BiocInstaller')
# options(BioC_mirror="https://mirrors.ustc.edu.cn/bioc/")
# BiocInstaller::biocLite("GEOquery")
# BiocInstaller::biocLite(c("limma"))
# BiocInstaller::biocLite(c("impute"))
# 但是接下来的代码又需要运行啦
options()$repos
install.packages('WGCNA')
install.packages(c("FactoMineR", "factoextra"))
install.packages(c("ggplot2", "pheatmap","ggpubr"))
library("FactoMineR")
library("factoextra")
library(GSEABase)
library(GSVA)
library(clusterProfiler)
library(ggplot2)
library(ggpubr)
library(hgu133plus2.db)
library(limma)
library(org.Hs.eg.db)
library(pheatmap)1.2 注意点及报错信息
1.2.1 注意Mac需要安装xcode工具箱
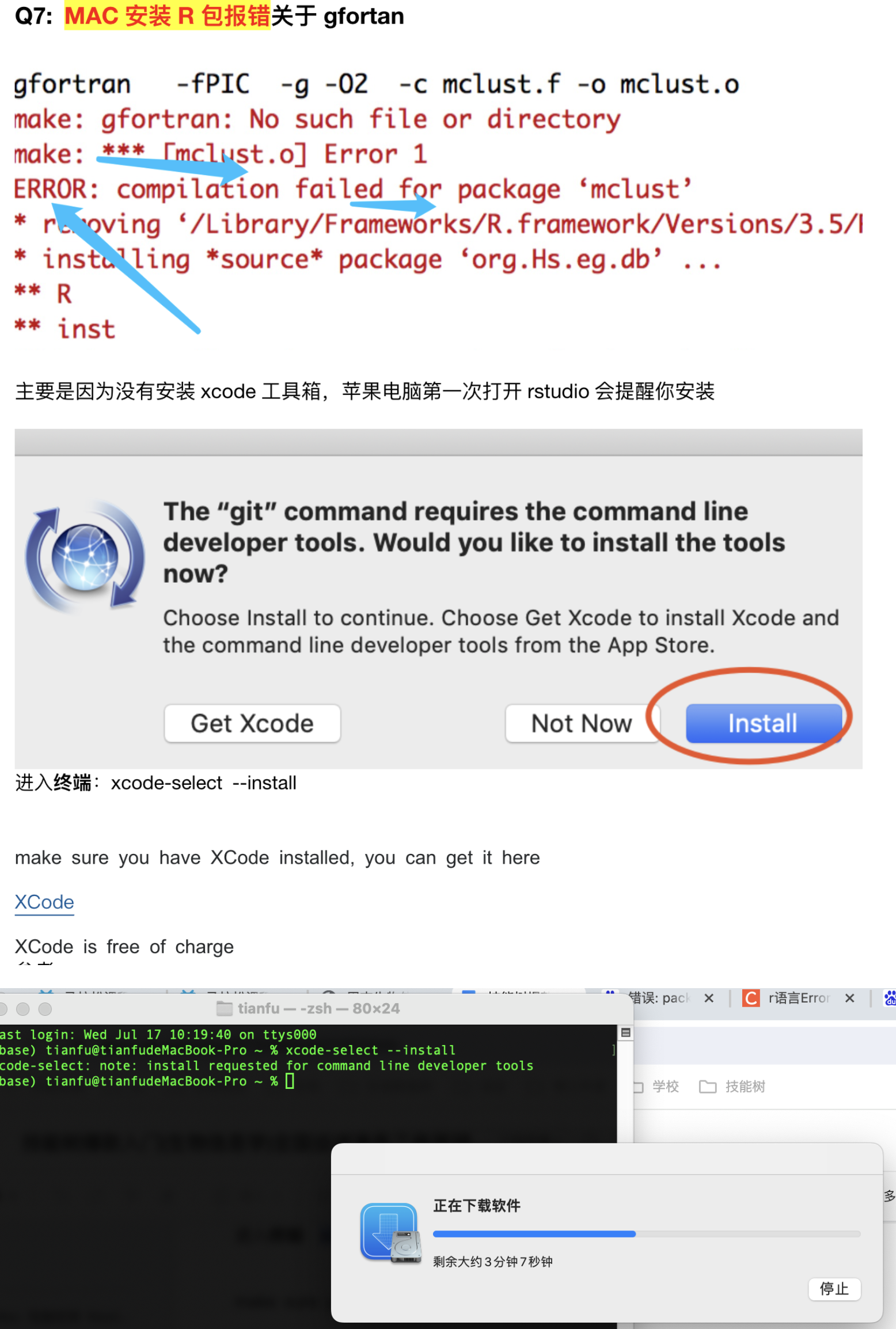
1.2.2 安装过程中依赖包版本不一致

解决办法是删除旧包,重新安装新的版本
remove.packages("cli")
install.packages("cli")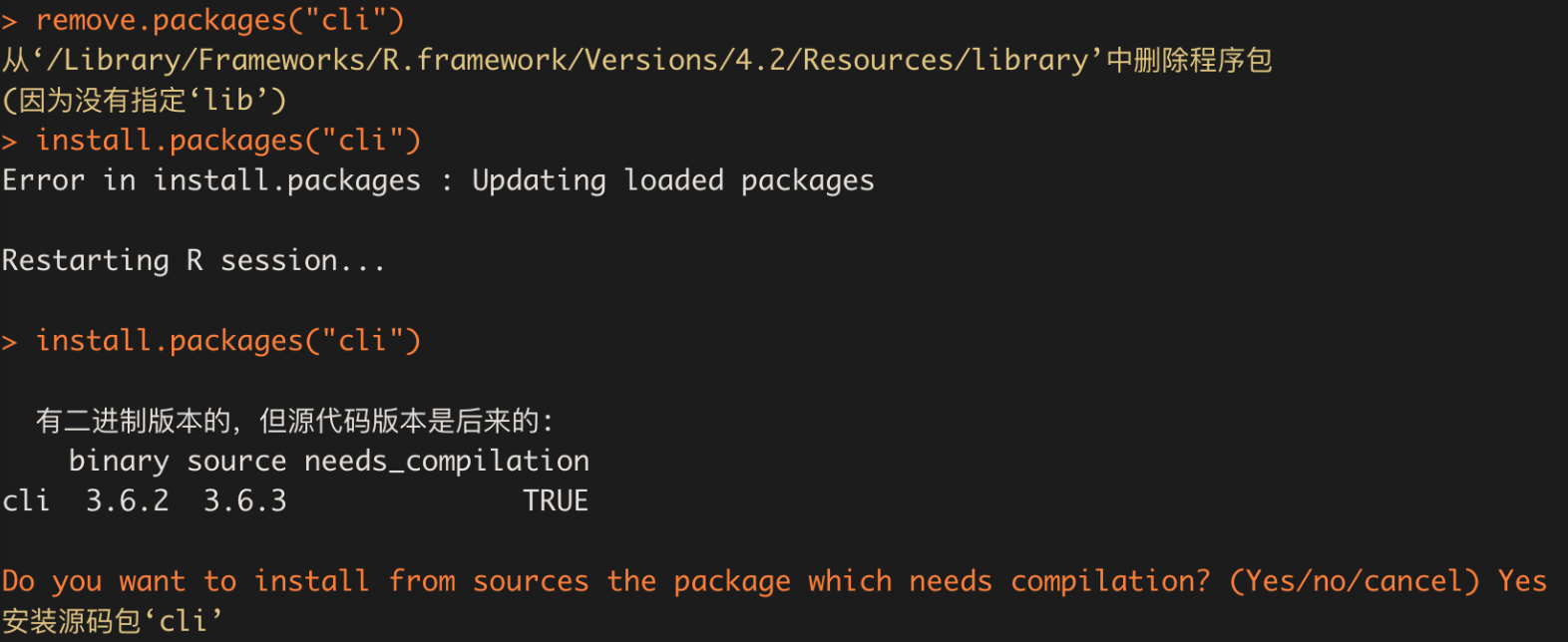
1.2.3 更新R版本到最新
才注意到自己电脑的R版本太低,可能满足不了学习的需要,因此卸载重新装了最新版本的R version 4.4.1 (2024-06-14),在重新执行上述代码时遇到新的问题。
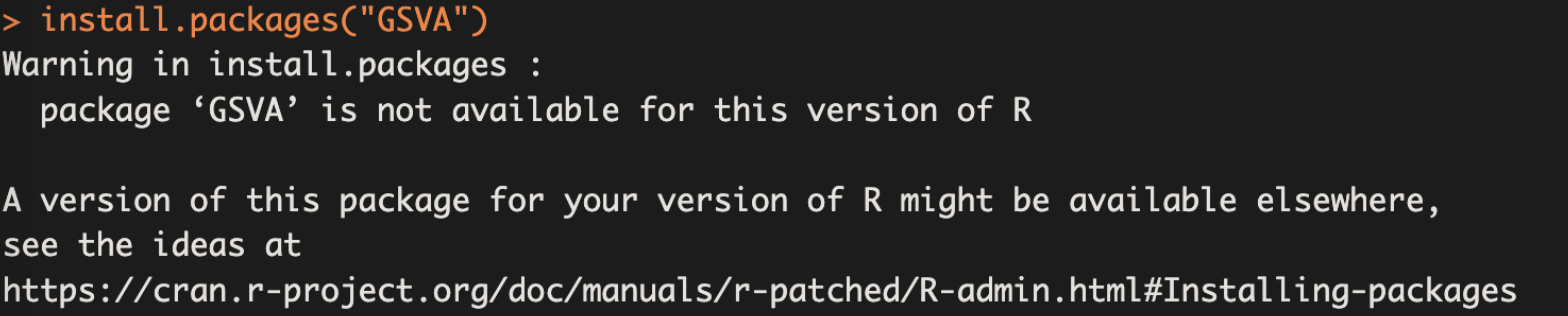
解决办法是使用Bioconductor源的安装函数
BiocManager::install("GSVA")
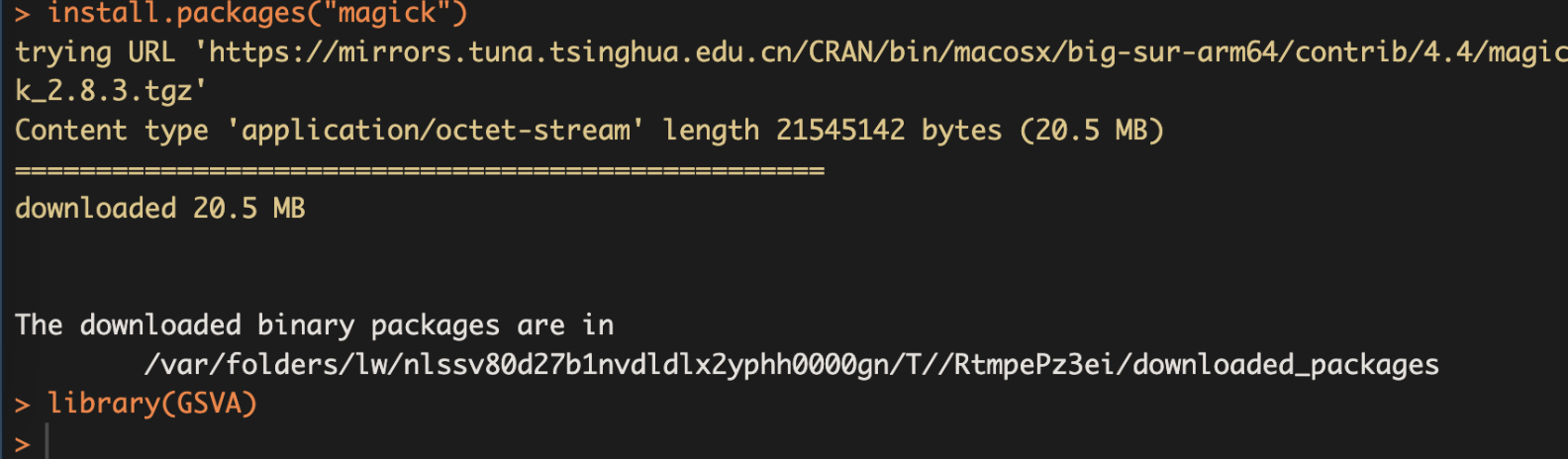
手动安装magick
1.2.4 R学习
#查看所安装的所有R包
installed.packages()
#清空控制台信息
Control + L(非 Command + L)
#清空环境变量
rm(list = ls()) 在使用R语言时,我们通常需要从CRAN(Comprehensive R Archive Network)下载和安装各种扩展包。CRAN是一个存储了大量R语言扩展包的网络服务器,用户可以通过它来获取需要的扩展包。默认情况下,R语言会使用CRAN的全球镜像源进行扩展包的下载。然而,有时候全球镜像源可能由于网络连接问题或地理位置的限制而导致下载速度较慢。为了解决这个问题,我们可以自定义指定CRAN本地镜像源,以提高下载扩展包的速度和稳定性。
生物信息学开源工具库(BioConductor)
#查看及设置CRAN,Bioconductor镜像源
options()$repos
options()$BioC_mirror
#options(BioC_mirror="https://mirrors.ustc.edu.cn/bioc/")
options(BioC_mirror="http://mirrors.tuna.tsinghua.edu.cn/bioconductor/")
options("repos" = c(CRAN="https://mirrors.tuna.tsinghua.edu.cn/CRAN/"))
options()$repos
options()$BioC_mirror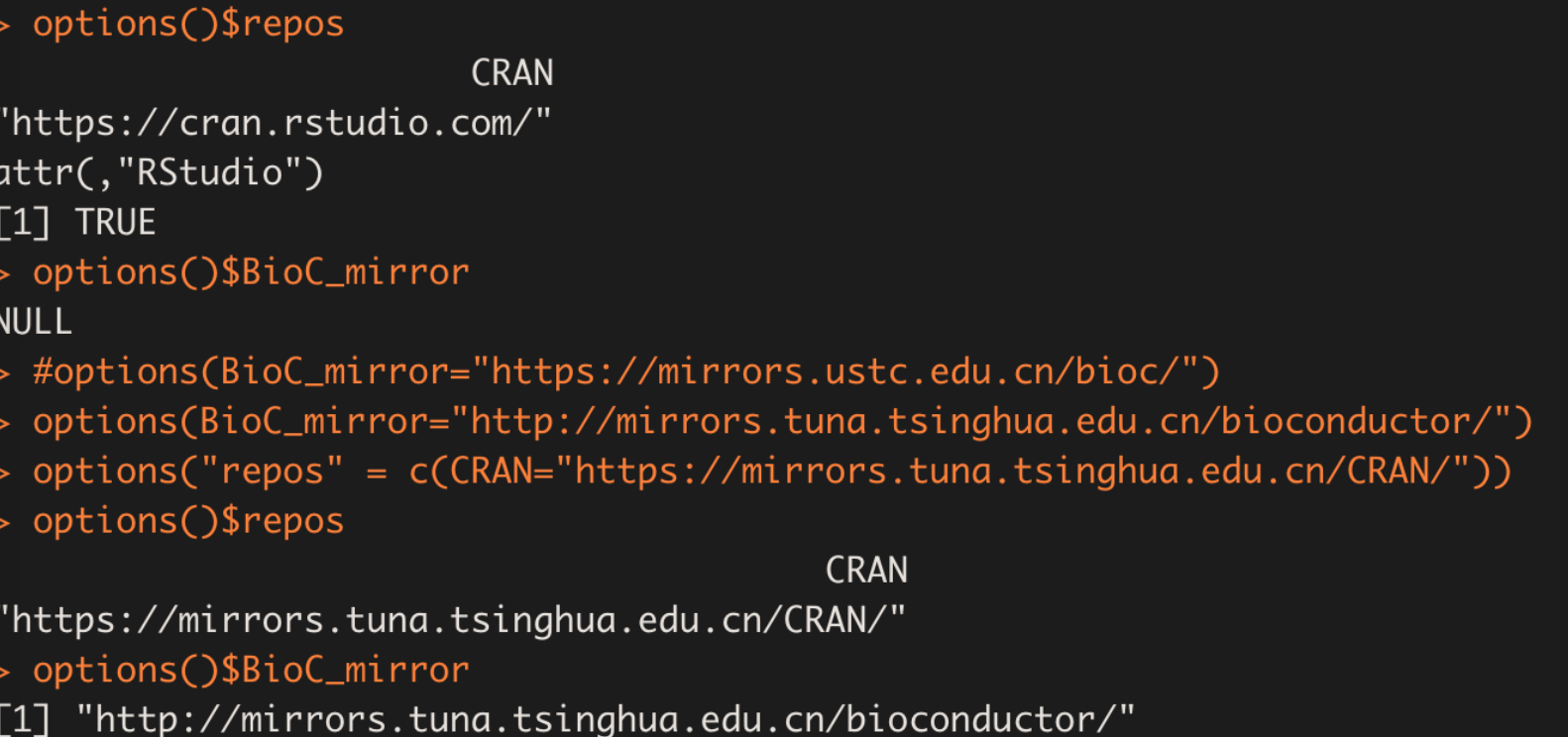
这种方法还是有问题,下次再打开Rstudio,下载Bioconductor还是会回到官方镜像,可以查询options()$BioC_mirror,如果依然是自己设置的国内镜像,就不用管了;如果发现需要再重新运行一遍代码进行设置,那么可以修改配置文件。这样就不需要重新设置了。
file.edit('~/.Rprofile')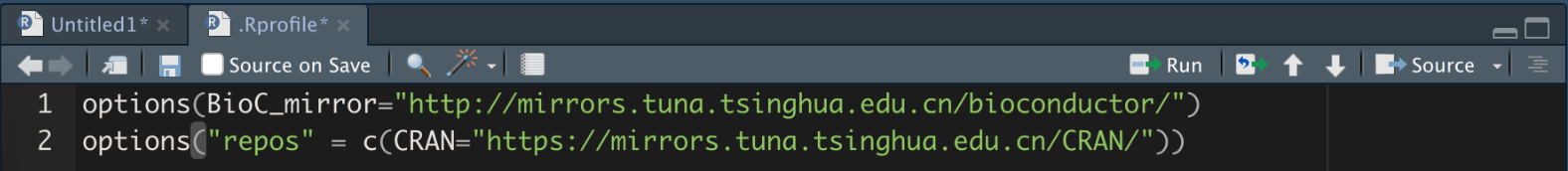
R包安装命令是install.packages(“包”)或者BiocManager::install(“包”)。取决于要安装的包存在于CRAN网站还是Biocductor,存在于哪里可以谷歌搜到。
2 生信入门直播课Day01
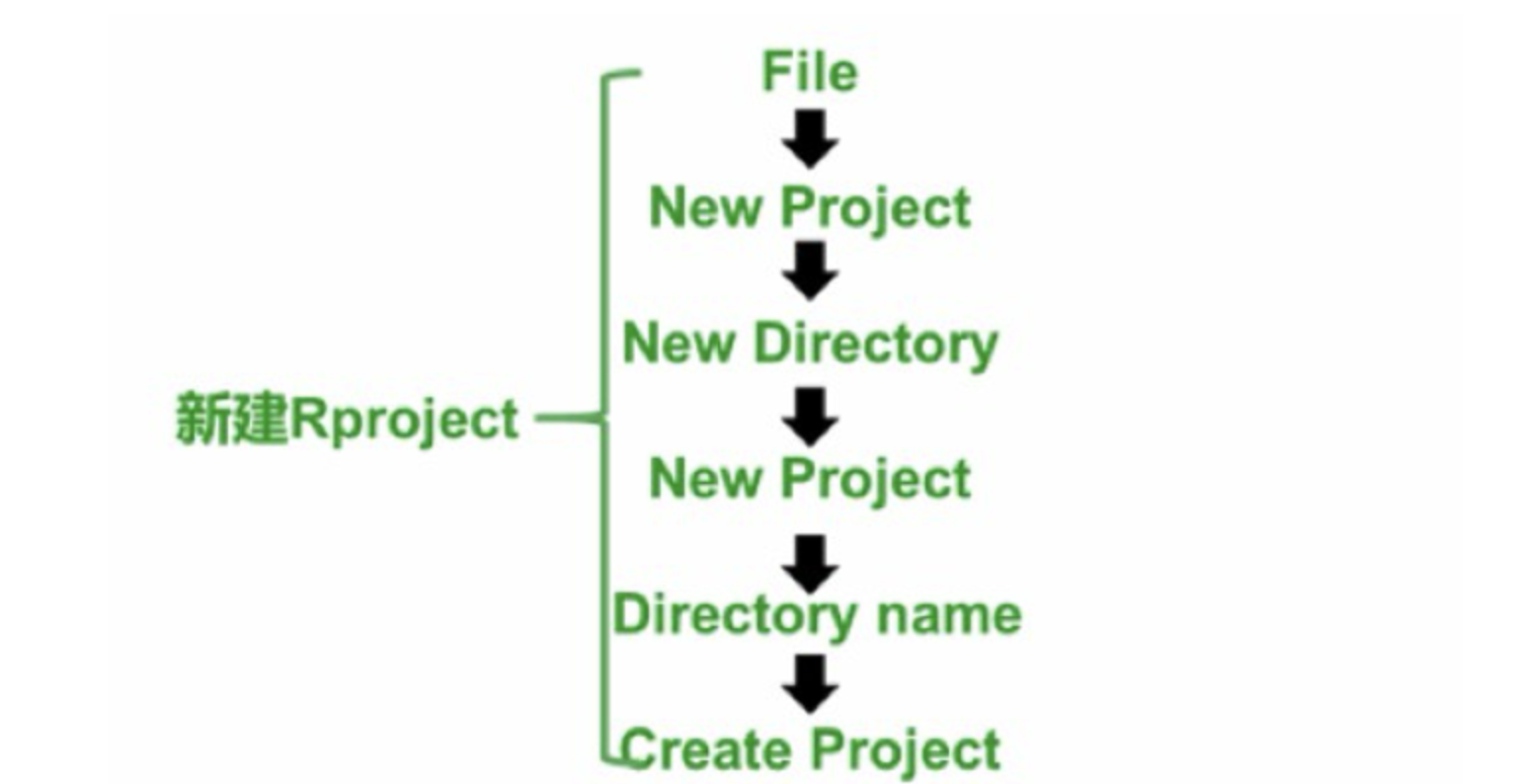
2.1 在Rstudio中新建R project及脚本(项目管理)
组织架构
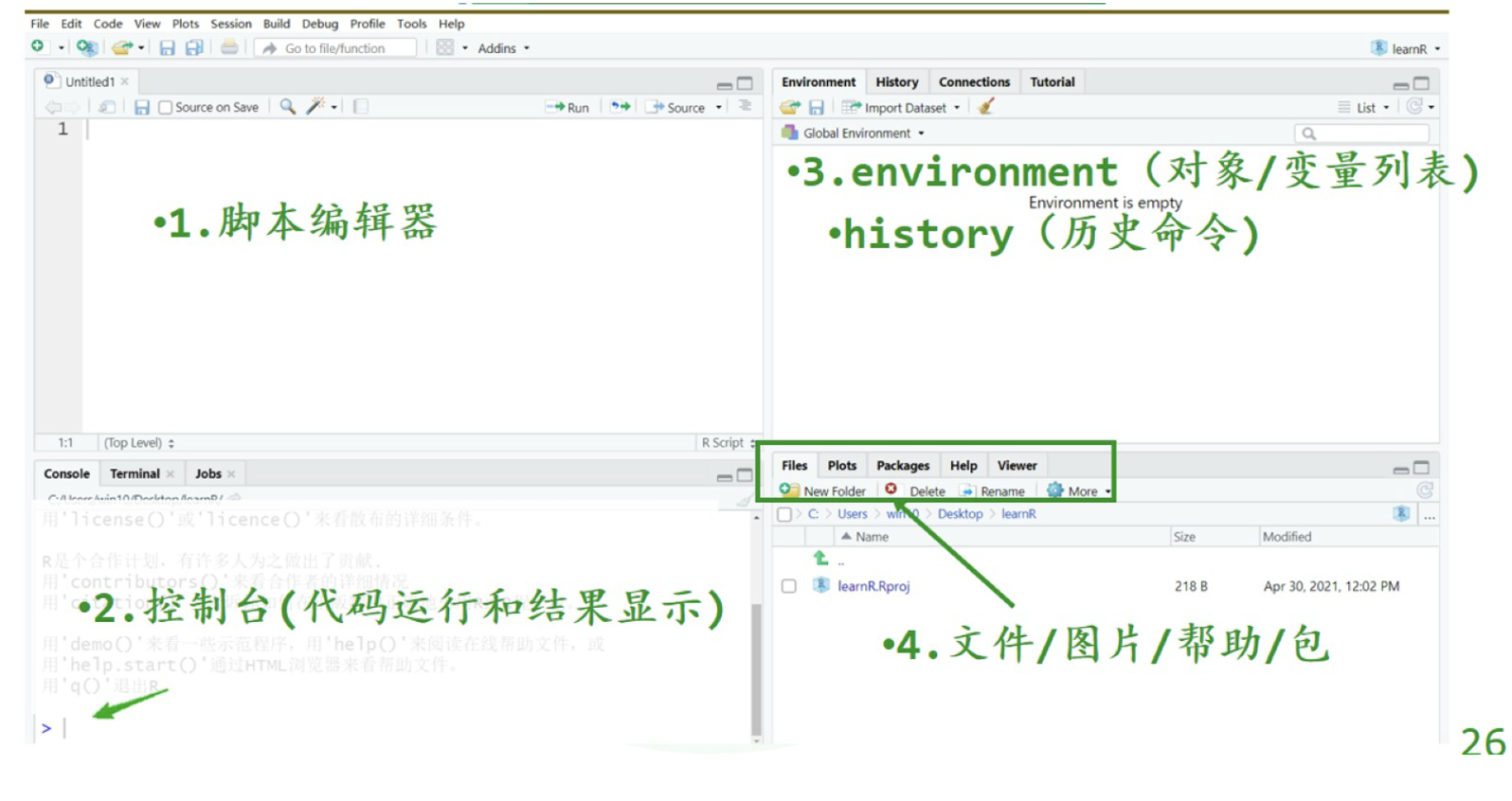
Rstudio面板介绍
部分面板消失的解决办法
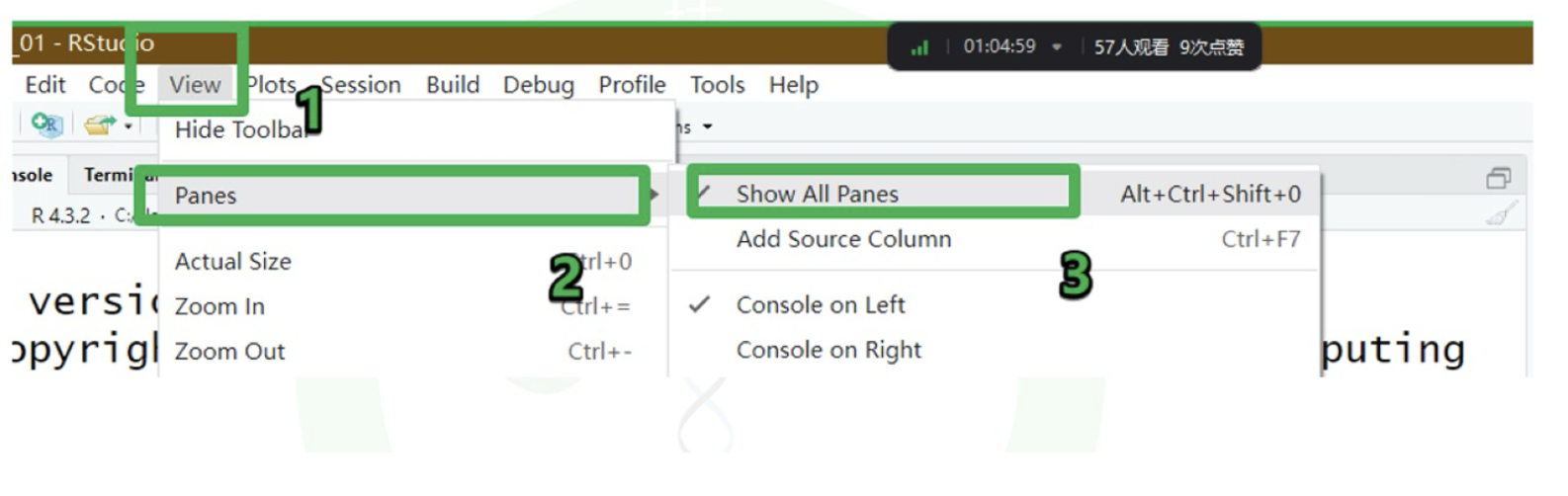
出现➕号的解决办法
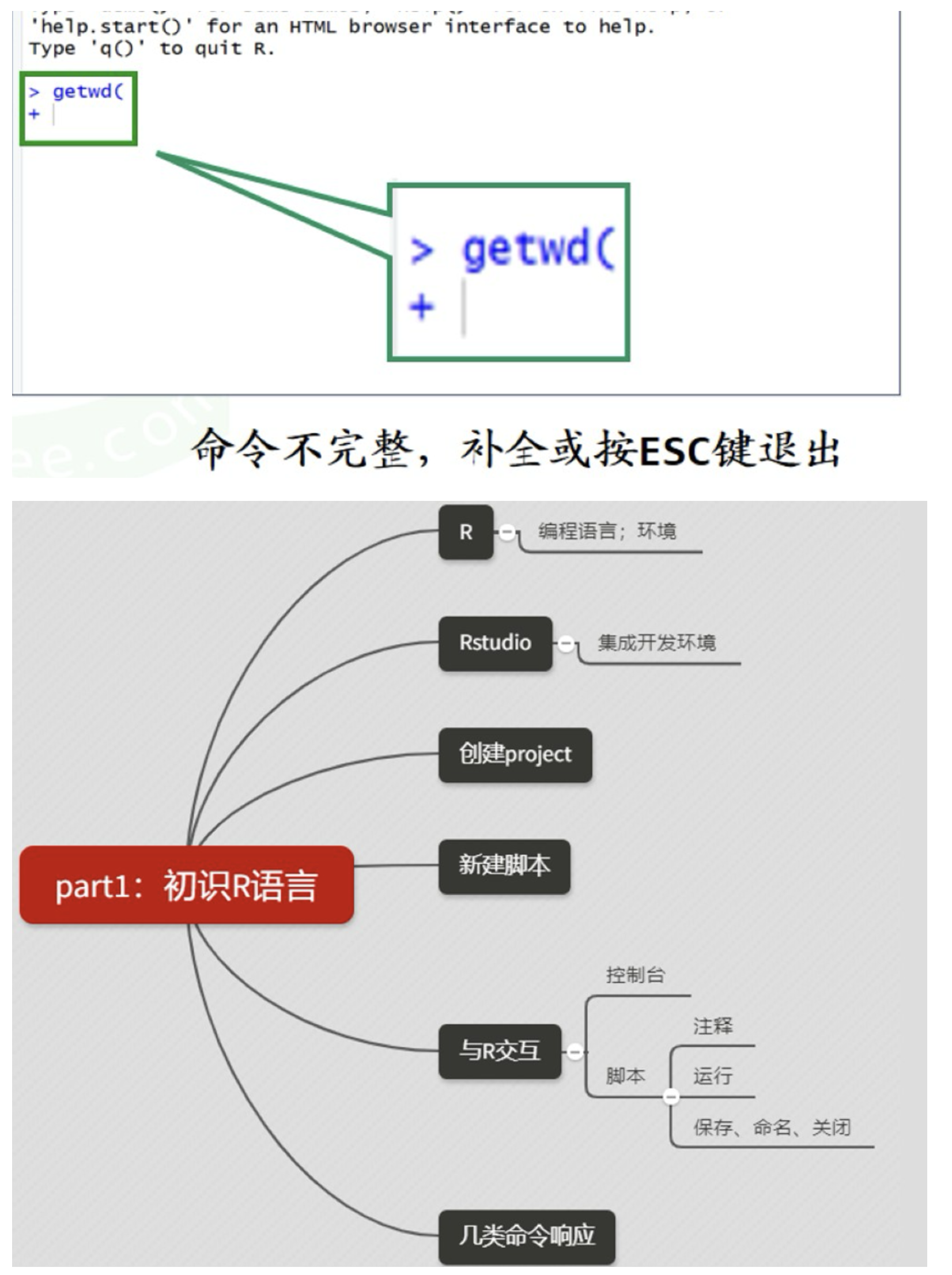
2.2 数据类型与向量
原创声明:本文系作者授权腾讯云开发者社区发表,未经许可,不得转载。
如有侵权,请联系 cloudcommunity@tencent.com 删除。
原创声明:本文系作者授权腾讯云开发者社区发表,未经许可,不得转载。
如有侵权,请联系 cloudcommunity@tencent.com 删除。

