GEO_加权共表达网络WGCNA
原创GEO_加权共表达网络WGCNA
1 前言
WGCNA(Weighted Gene Co-Expression Network Analysis,即加权基因共表达网络分析)是一种用于分析基因表达数据的系统生物学方法。WGCNA的主要目的是识别基因表达数据中的共表达模块,并研究这些模块与外部样本特征(例如,疾病状态、临床特征等)之间的关系。
下面是chatgpt给出的更为通俗易通的解释
WGCNA(加权基因共表达网络分析)是一种分析基因表达数据的方法,旨在发现一组基因是如何共同工作的。可以将其想象为一种找出基因之间“朋友圈”的方法。以下是WGCNA的一些通俗描述:
基本概念
- 基因共表达:就像某些人经常一起活动,某些基因在不同条件下(如健康与疾病状态)也表现得相似。
- 网络构建:将这些“社交关系”用网络图表示,网络中的节点代表基因,连接线(边)代表它们之间的共表达关系。
- 加权网络:不仅仅判断基因是否相关,还要看它们的关系有多紧密,用“权重”来表示这种紧密程度。
主要步骤
- 识别“朋友圈”:首先,通过计算基因表达的相似性,识别出哪些基因经常一起工作,形成一个个“朋友圈”,这些“朋友圈”叫做模块。
- 模块分析:每个模块代表了一组共同工作的基因,可以帮助我们理解它们在某种生物过程或疾病中的作用。
- 关联分析:将这些基因模块与外部特征(如疾病状态、临床数据)进行关联,看看哪些模块与这些特征相关。
2 数据预处理
2.1 数据集获取
以GSE199335数据集为例分析
library(tinyarray)
gse = "GSE199335"
geo = geo_download(gse,destdir=".",colon_remove = T)
geo$gpl
#View(geo$pd)
library(stringr)
#分组信息
Group = paste(geo$pd$genotype,geo$pd$age,sep="_") %>%
str_remove(" months of age| weeks of age") %>%
str_remove(" type") %>%
str_replace("/",".")
table(Group)
R6.1_6 R6.2_9 wild_6 wild_9
3 4 4 4
Group = factor(Group,levels = c("wild_6", "wild_9", "R6.1_6", "R6.2_9"));Group
[1] wild_6 wild_6 wild_6 R6.1_6 R6.1_6 wild_6 R6.1_6 wild_9 wild_9 wild_9 wild_9 R6.2_9
[13] R6.2_9 R6.2_9 R6.2_9
Levels: wild_6 wild_9 R6.1_6 R6.2_9
#获取探针注释,按照之前学的改动
#idmap只能获取2019年前的数据,可以采用之前的方式获取
ids <- AnnoProbe::idmap(geo$gpl,destdir = tempdir(),type = "soft")
ids = na.omit(ids)
colnames(ids) = c("probe_id","symbol")
#探针表达矩阵转换为基因表达矩阵
exp = trans_array(geo$exp,ids)
pd = geo$pd
table(Group)
save(exp,Group,pd,file = "Dat.Rdata")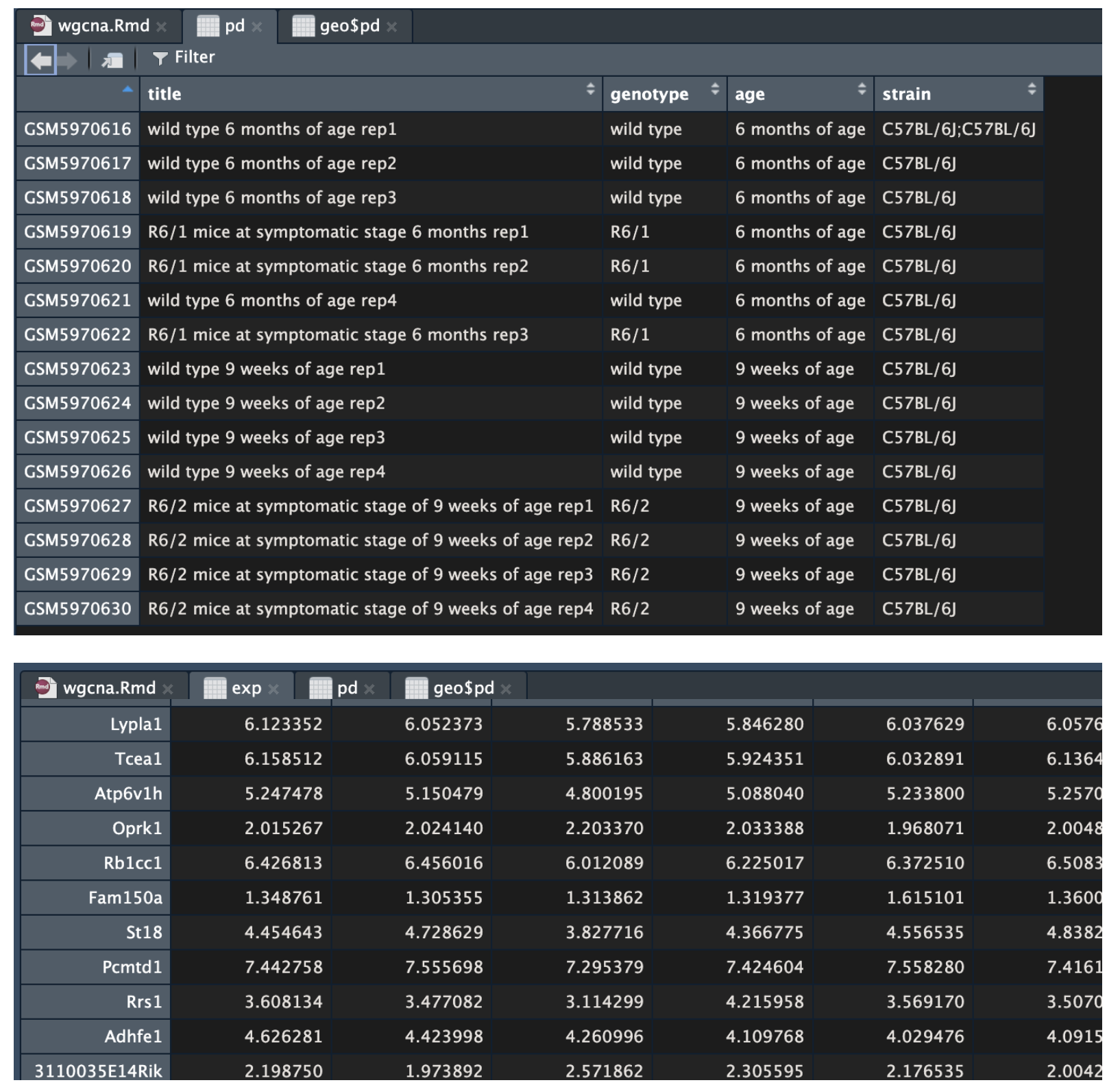
下面的代码主要自WGCNA的官方文档,进行了一些改动。
2.2 表达矩阵数据整理
首先需要有至少15个样本。
行是样本,列是基因。
不推荐使用全部基因,计算量太大
也不推荐使用差异分析所得的差异基因,包作者说的。
比较推荐的方法是按照方差或者mad去取前多少个基因,例如3500,5000,8000个,或者保留基因总数的前1/4。无标准答案。
library(WGCNA)
library(tinyarray)
load("Dat.Rdata")
#exp = log2(geo$exp+1)
boxplot(exp)#从这张图可以看出数据是取过log的,且没有异常样本,可以用
#⭐mad最大的,5000可以改为其他数字,例如3500,5000,8000,或者保留基因总数的前1/4(round(0.25*nrow(exp)))。mad可以改为var(方差)
datExpr0 = t(exp[order(apply(exp, 1, mad), decreasing = T)[1:5000],])
datExpr0[1:4,1:4]
Bpifa6 Scd3 Myh4 Opn1sw
GSM5970616 5.808989 5.964354 8.777054 7.488333
GSM5970617 4.272349 4.216835 9.052218 7.843838
GSM5970618 2.558643 4.095240 8.907886 7.464708
GSM5970619 2.081443 4.061555 7.401055 2.9124782.3 基因与样本过滤
基因过滤主要看缺失值,GEO的芯片数据大多数没缺失值。
样本过滤主要看是否有太离群的聚类
看有没有自己单独一个分支的样本, 如果有异常值就需要去掉,根据聚类图自己设置cutHeight 参数的值。F改为T;无异常值就不改
#基因过滤
gsg = goodSamplesGenes(datExpr0, verbose = 3)
gsg$allOK # 返回TRUE则继续
if (!gsg$allOK){
# 把含有缺失值的基因或样本打印出来
if (sum(!gsg$goodGenes)>0)
printFlush(paste("Removing genes:", paste(names(datExpr0)[!gsg$goodGenes], collapse = ", ")));
if (sum(!gsg$goodSamples)>0)
printFlush(paste("Removing samples:", paste(rownames(datExpr0)[!gsg$goodSamples], collapse = ", ")));
#去掉那些缺失值
datExpr0 = datExpr0[gsg$goodSamples, gsg$goodGenes]
#样本聚类
sampleTree = hclust(dist(datExpr0), method = "average")
png(file = "2.sampleClustering.png", width = 2000, height = 2000,res = 300)
par(cex = 0.6)
par(mar = c(0,4,2,0))
plot(sampleTree, main = "Sample clustering to detect outliers", sub="", xlab="", cex.lab = 1.5,cex.axis = 1.5, cex.main = 2)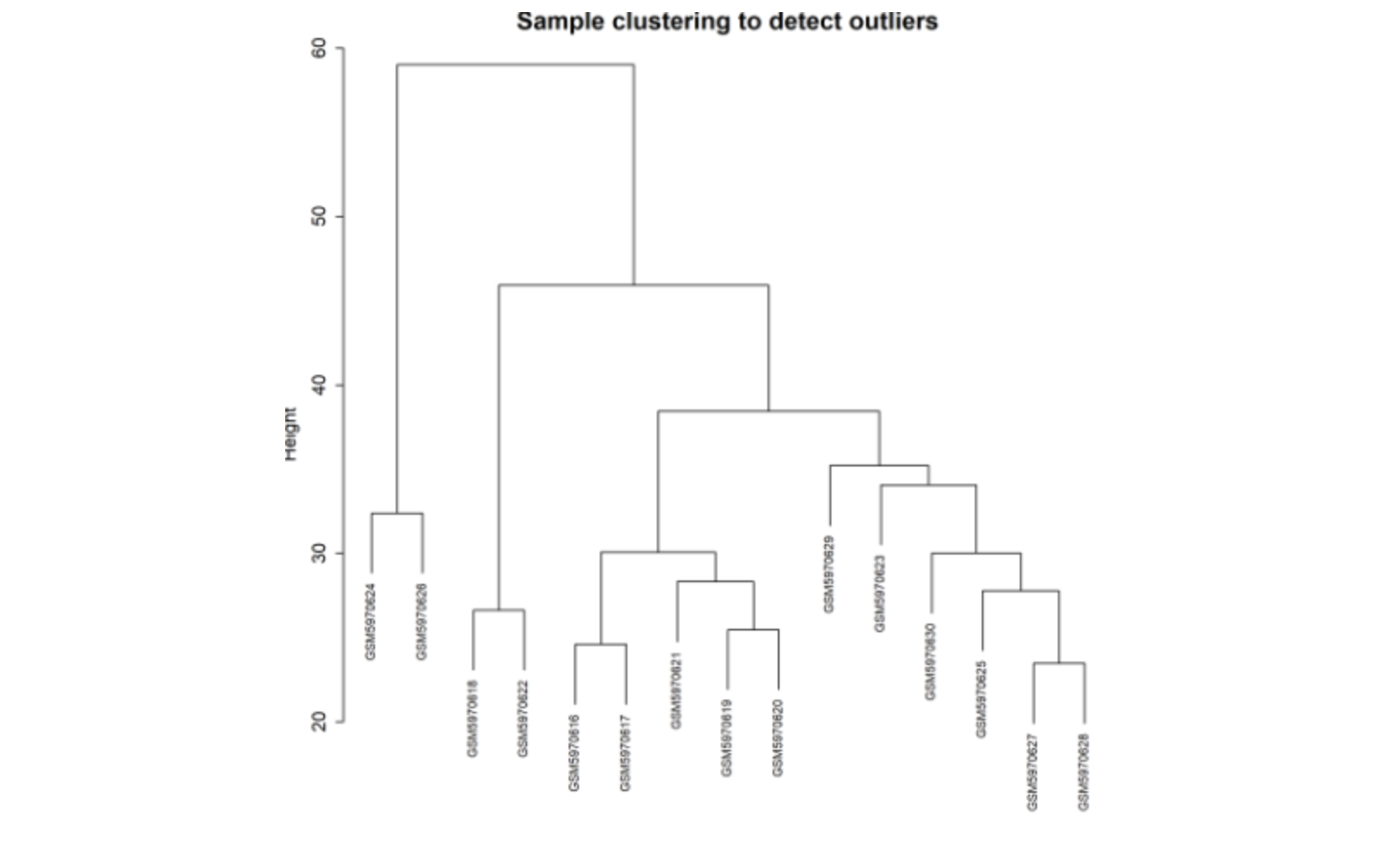
# 如果有异常值就需要去掉,根据聚类图自己设置cutHeight 参数的值。F改为T;无异常值就不改
if(F){
clust = cutreeStatic(sampleTree, cutHeight = 170, minSize = 10)
table(clust) # 0代表切除的,1代表保留的
keepSamples = (clust!=0)
datExpr = datExpr0[keepSamples, ]
}else{
#没有异常样本就不需要去除
datExpr = datExpr0
}2.4 表型信息的整理
这个信息来自芯片数据的pData,也就是上面的pd。要求必须是数值型,要么像年龄那样的数字,要么搞成0,1,或者是1,2,3等。这里利用了因子转数字的方法,转换成了数值。
library(stringr)
#自己需要的临床信息,每个一列,必须是数值型
traitData = data.frame(genotype = as.numeric(as.factor(pd$genotype)),
age = as.numeric(as.factor(pd$age)))
str(traitData)
'data.frame': 15 obs. of 2 variables:
$ genotype: num 3 3 3 1 1 3 1 3 3 3 ...
$ age : num 1 1 1 1 1 1 1 2 2 2 ...
table(traitData[,1])
1 2 3
3 4 8
names(traitData)
[1] "genotype" "age" 
即这个数据有用的表型只有两列"genotype" "age"
datTraits = traitData
sampleTree2 = hclust(dist(datExpr), method = "average")
# 用颜色表示表型在各个样本的表现: 白色表示低,红色为高,灰色为缺失
traitColors = numbers2colors(datTraits, signed = FALSE)
# 把样本聚类和表型绘制在一起
png(file = "3.Sample dendrogram and trait heatmap.png", width = 2000, height = 2000,res = 300)
plotDendroAndColors(sampleTree2, traitColors,
groupLabels = names(datTraits),
main = "Sample dendrogram and trait heatmap")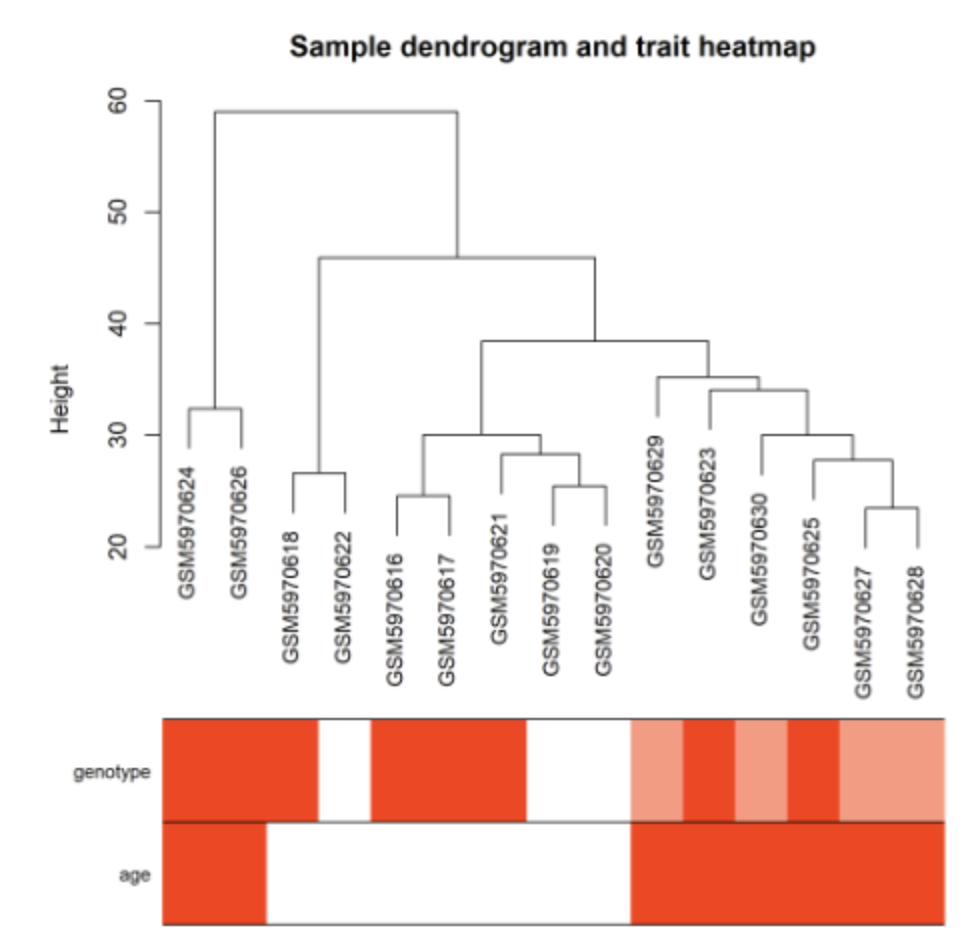
3 WGCNA
3.1 软阈值的选择
设置一系列软阈值,范围是1-30之间,后面的数没必要全部画,就seq一下。
powers = c(1:10, seq(from = 12, to=30, by=2))
sft = pickSoftThreshold(datExpr, powerVector = powers, verbose = 5)
sft$powerEstimate #推荐的软阈值
[1] 12这个结果就是推荐的软阈值,拿到了可以直接用,无视下面的图。有的数据走到这一步会得到NA,也就是没得推荐.那就要看下面的图,选拐点。根据经验,没有推荐软阈值,或者数字太大,后面跌跌撞撞走起来有些艰难,就得跑到前面重新调整表达矩阵里纳入的基因了。ex1一般设置为0.9,不太合适(就是大多数软阈值对应的纵坐标都达不到0.9)时,可以设置为0.8或者0.85。一般不能再低了。
cex1 = 0.9
png(file = "4.Soft threshold.png", width = 2000, height = 1500,res = 300)
par(mfrow = c(1,2))
plot(sft$fitIndices[,1], -sign(sft$fitIndices[,3])*sft$fitIndices[,2],
xlab="Soft Threshold (power)",
ylab="Scale Free Topology Model Fit,signed R^2",type="n",
main = paste("Scale independence"))
text(sft$fitIndices[,1], -sign(sft$fitIndices[,3])*sft$fitIndices[,2],
labels=powers,cex=cex1,col="red")
abline(h=cex1,col="red")
plot(sft$fitIndices[,1], sft$fitIndices[,5],
xlab="Soft Threshold (power)",ylab="Mean Connectivity", type="n",
main = paste("Mean connectivity"))
text(sft$fitIndices[,1], sft$fitIndices[,5], labels=powers, cex=cex1,col="red")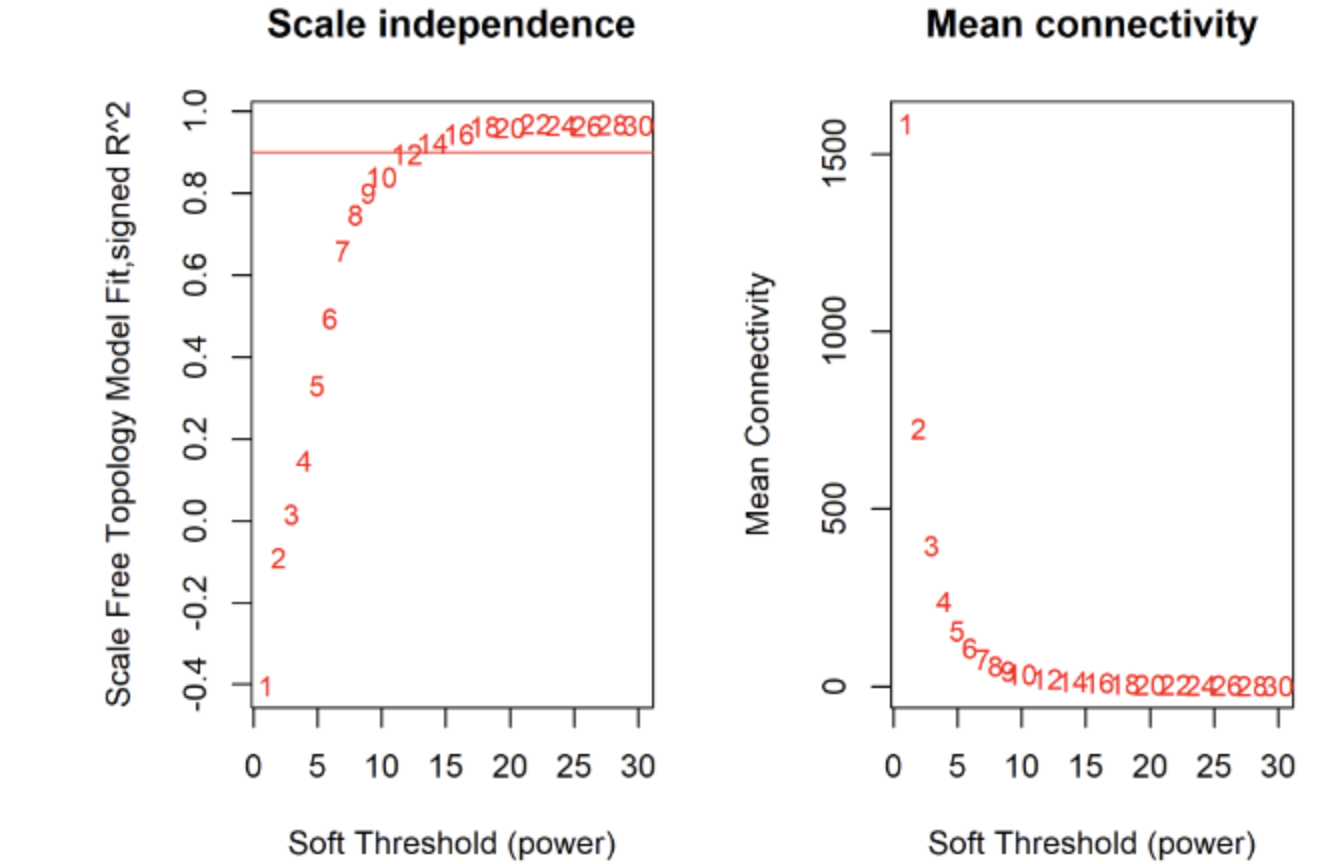
3.2 一步构建网络
如果前面没有得到推荐的软阈值,这里的power就要自己指定一个。根据上面的图,选择左图纵坐标第一个达到上面设置的cex1值的软阈值。
power = sft$powerEstimate
power
net = blockwiseModules(datExpr, power = power,
TOMType = "unsigned",
minModuleSize = 30,
reassignThreshold = 0,
mergeCutHeight = 0.25,
deepSplit = 2 ,
numericLabels = TRUE,
pamRespectsDendro = FALSE,
saveTOMs = TRUE,
saveTOMFileBase = "testTOM",
verbose = 3)后面的结果如果不理想,比如划分的模块太少,或者青色、灰色的模块占到大多数,其他颜色都很少,或者颜色模块聚类是凌乱的,可以倒回来调整几个参数。
deepSplit 默认2,调整划分模块的敏感度,值越大,越敏感,得到的模块就越多; minModuleSize 默认30,参数设置最小模块的基因数,值越小,小的模块就会被保留下来; mergeCutHeight 默认0.25,设置合并相似性模块的距离,值越小,就越不容易被合并,保留下来的模块就越多
此处展示得到了多少模块,每个模块里面有多少基因。
table(net$colors)
0 1 2 3 4 5 6 7 8 9
200 785 685 654 628 626 527 447 253 195 mergedColors = labels2colors(net$colors)
png(file = "5.DendroAndColors.png", width = 2000, height = 1200,res = 300)
plotDendroAndColors(net$dendrograms[[1]], mergedColors[net$blockGenes[[1]]],
"Module colors",
dendroLabels = FALSE, hang = 0.03,
addGuide = TRUE, guideHang = 0.05)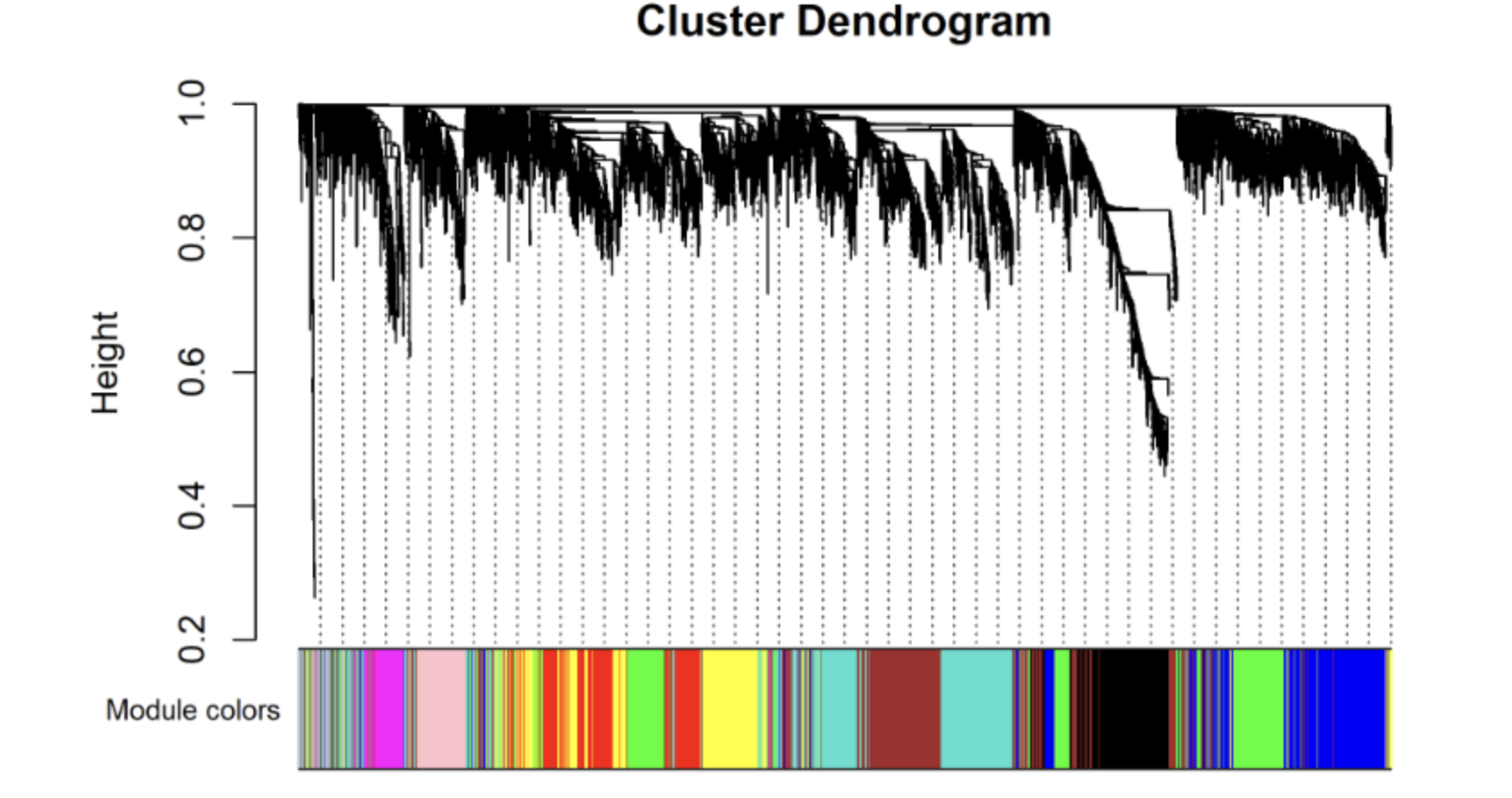
好的结果就是每个颜色都差不多在一起,(青色和灰色不在考虑范围内)。然后青色和灰色的基因不要太多。因为灰色代表没有合适的聚类,青色是基因数量的模块,比如你输入5000个基因,其中3000个都属于青色,剩下的模块基因数量太少,就很难受了。
3.3 保存每个模块对应的基因
把每个模块对应的基因存为了Rdata,用于数据挖掘下一步需求,提取基因。比如你需要某模块的基因与差异基因取交集等。
moduleLabels = net$colors
moduleColors = labels2colors(net$colors)
MEs = net$MEs
geneTree = net$dendrograms[[1]]
gm = data.frame(net$colors)
gm$color = moduleColors
head(gm)
genes = split(rownames(gm),gm$color)
save(genes,file = "genes.Rdata")3.4 模块与表型的相关性
计算基因与表型的相关性矩阵,我们希望找到和每个表型相关性较强的模块,正负相关都可。相关系数越大越好,如果能有个0.8,那结论就比较稳啦!没有的话0.6或0.7几也行,再小就不要拿来糊弄人了。
nGenes = ncol(datExpr)
nSamples = nrow(datExpr)
MEs0 = moduleEigengenes(datExpr, moduleColors)$eigengenes
MEs = orderMEs(MEs0)
moduleTraitCor = cor(MEs, datTraits, use = "p")
moduleTraitPvalue = corPvalueStudent(moduleTraitCor, nSamples)
#热图
png(file = "6.labeledHeatmap.png", width = 2000, height = 2000,res = 300)
# 设置热图上的文字(两行数字:第一行是模块与各种表型的相关系数;
# 第二行是p值)
# signif 取有效数字
textMatrix = paste(signif(moduleTraitCor, 2), "\n(",
signif(moduleTraitPvalue, 1), ")", sep = "")
dim(textMatrix) = dim(moduleTraitCor)
par(mar = c(6, 8.5, 3, 3))
# 然后对moduleTraitCor画热图
labeledHeatmap(Matrix = moduleTraitCor,
xLabels = names(datTraits),
yLabels = names(MEs),
ySymbols = names(MEs),
colorLabels = FALSE,
colors = blueWhiteRed (50),
textMatrix = textMatrix,
setStdMargins = FALSE,
cex.text = 0.5,
zlim = c(-1,1),
main = paste("Module-trait relationships"))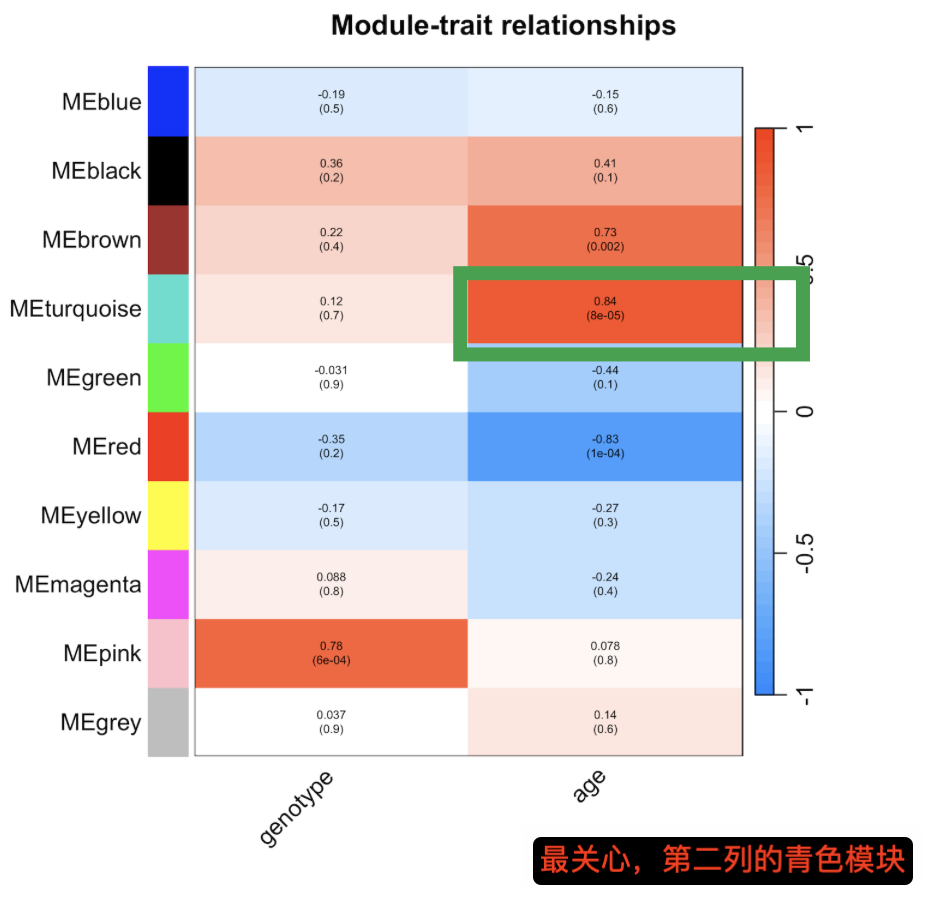
3.5 GS与MM
GS代表模块里的每个基因与形状的相关性,MM代表每个基因和所在模块之间的相关性,表示是否与模块的趋势一致。
modNames = substring(names(MEs), 3)
geneModuleMembership = as.data.frame(cor(datExpr, MEs, use = "p"))
MMPvalue = as.data.frame(corPvalueStudent(as.matrix(geneModuleMembership), nSamples))
names(geneModuleMembership) = paste("MM", modNames, sep="")
names(MMPvalue) = paste("p.MM", modNames, sep="")第几列的表型是最关心的,下面的i就设置为几。
与关心的表型相关性最高的模块赋值给下面的module。
i = 2 #
#module = "pink"
module = "turquoise"#
assign(colnames(traitData)[i],traitData[i])
instrait = eval(parse(text = colnames(traitData)[i]))
geneTraitSignificance = as.data.frame(cor(datExpr, instrait, use = "p"))
GSPvalue = as.data.frame(corPvalueStudent(as.matrix(geneTraitSignificance), nSamples))
names(geneTraitSignificance) = paste("GS.", names(instrait), sep="")
names(GSPvalue) = paste("p.GS.", names(instrait), sep="")
png(file = paste0("7.MM-GS-scatterplot.png"), width = 2000, height = 2000,res = 300)
column = match(module, modNames) #找到目标模块所在列
moduleGenes = moduleColors==module #找到模块基因所在行
par(mfrow = c(1,1))
verboseScatterplot(abs(geneModuleMembership[moduleGenes, column]),
abs(geneTraitSignificance[moduleGenes, 1]),
xlab = paste("Module Membership in", module, "module"),
ylab = "Gene significance",
main = paste("Module membership vs. gene significance\n"),
cex.main = 1.2, cex.lab = 1.2, cex.axis = 1.2, col = module)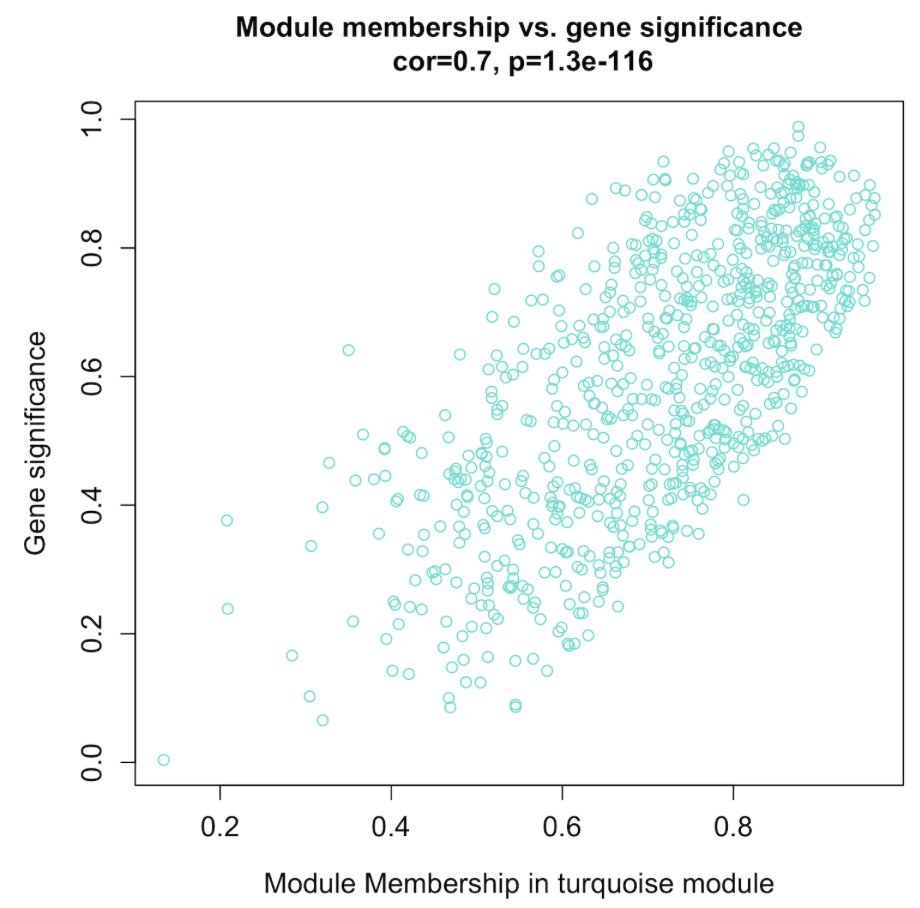
我们希望看到强悍的正相关。数据挖掘里面,可以把整个模块的基因拿出来和别的步骤结果取交集了。
也可以通过这里找到GS和MM都大的基因。
f = data.frame(GS = abs(geneModuleMembership[moduleGenes, column]),
MM = abs(geneTraitSignificance[moduleGenes, 1]))
rownames(f) = rownames(gm[moduleGenes,])
head(f)
GS MM
Rnu1b1 0.8667593 0.9482171
ND6 0.8599513 0.9310548
Vmn1r127 0.8839290 0.8336949
Snora62 0.8862433 0.9287403
Crygf 0.8759284 0.9884732
Snord68 0.8733251 0.90279533.6 TOM
用基因相关性热图的方式展示加权网络,每行每列代表一个基因。一般取400个基因画就够,拿全部基因去做电脑要烧起来了。就想看看对角线附近红彤彤的小方块,以及同一个颜色基本都在一起。
nSelect = 400
set.seed(10)
dissTOM = 1-TOMsimilarityFromExpr(datExpr, power = 6)
select = sample(nGenes, size = nSelect)
selectTOM = dissTOM[select, select]
# 再计算基因之间的距离树(对于基因的子集,需要重新聚类)
selectTree = hclust(as.dist(selectTOM), method = "average")
selectColors = moduleColors[select]
library(ggplot2)
myheatcol = colorpanel(250,'red',"orange",'lemonchiffon')
png(file = "8.Sub400-netheatmap.png", width = 2000, height = 2000,res = 300)
plotDiss = selectTOM^7
diag(plotDiss) = NA #将对角线设成NA,在图形中显示为白色的点,更清晰显示趋势
TOMplot(plotDiss, selectTree, selectColors, col=myheatcol,main = "Network heatmap plot, selected genes")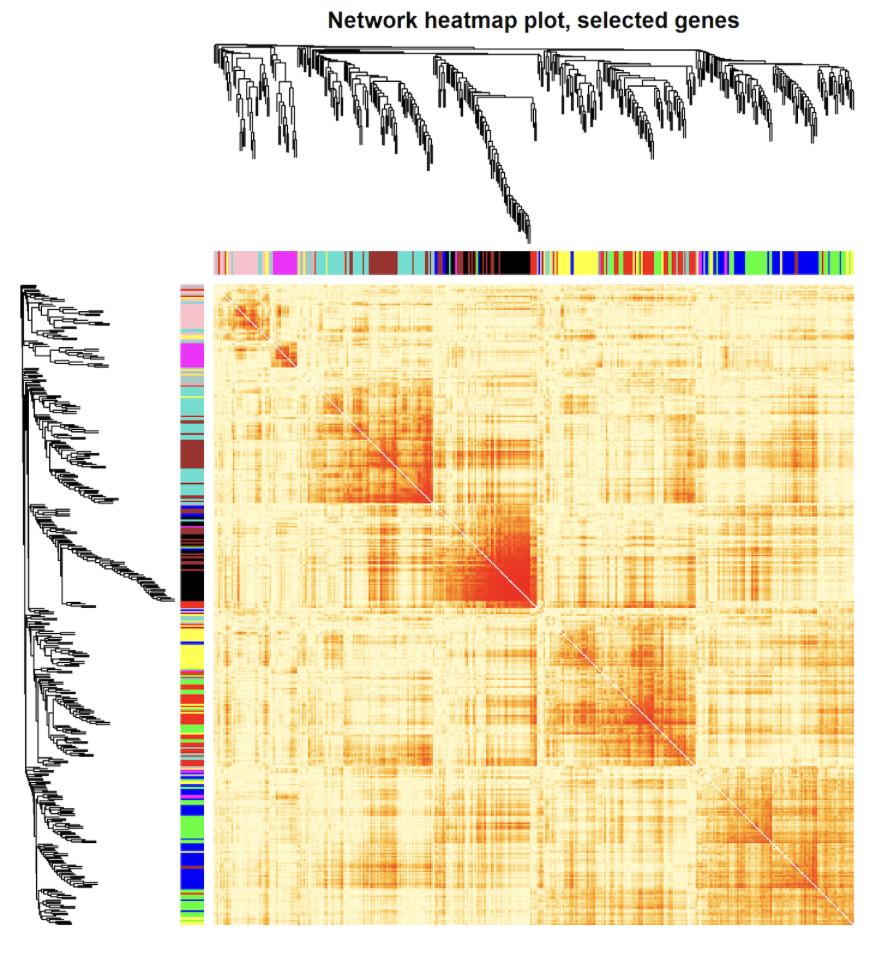
3.7 模块与表型的相关性
MEs = moduleEigengenes(datExpr, moduleColors)$eigengenes
MET = orderMEs(cbind(MEs, instrait))
png(file = "9.Eigengene-dengro-heatmap.png", width = 2000, height = 2000,res = 300)
par(cex = 0.9)
plotEigengeneNetworks(MET, "", marDendro = c(0,4,1,2), marHeatmap = c(4,4,1,2), cex.lab = 0.8, xLabelsAngle = 90)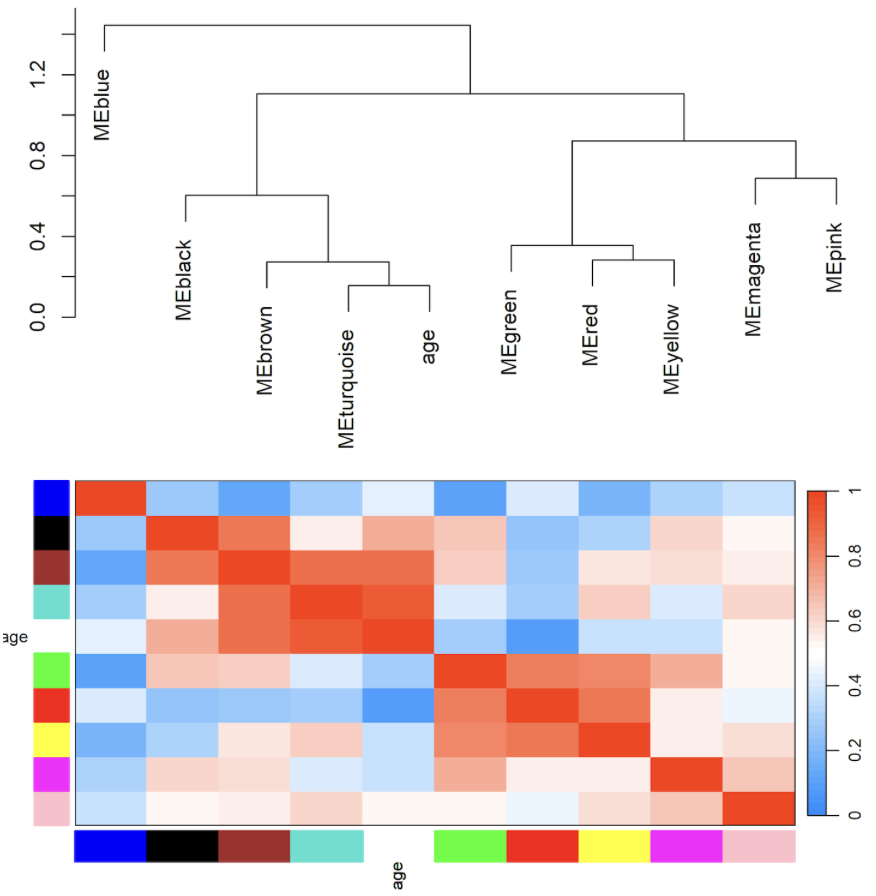
原创声明:本文系作者授权腾讯云开发者社区发表,未经许可,不得转载。
如有侵权,请联系 cloudcommunity@tencent.com 删除。
原创声明:本文系作者授权腾讯云开发者社区发表,未经许可,不得转载。
如有侵权,请联系 cloudcommunity@tencent.com 删除。

