【生物信息学】基因富集分析enrichment
【生物信息学】基因富集分析enrichment

一、实验介绍
本实验完成了基因富集分析
二、实验环境
本系列实验使用了PyTorch深度学习框架,相关操作如下(基于深度学习系列文章的环境):
1. 配置虚拟环境
深度学习系列文章的环境
conda create -n DL python=3.7 conda activate DLpip install torch==1.8.1+cu102 torchvision==0.9.1+cu102 torchaudio==0.8.1 -f https://download.pytorch.org/whl/torch_stable.htmlconda install matplotlibconda install scikit-learn新增加
conda install pandasconda install seabornconda install networkxconda install statsmodelspip install pyHSICLasso注:本人的实验环境按照上述顺序安装各种库,若想尝试一起安装(天知道会不会出问题)
2. 库版本介绍
软件包 | 本实验版本 | 目前最新版 |
|---|---|---|
matplotlib | 3.5.3 | 3.8.0 |
numpy | 1.21.6 | 1.26.0 |
python | 3.7.16 | |
scikit-learn | 0.22.1 | 1.3.0 |
torch | 1.8.1+cu102 | 2.0.1 |
torchaudio | 0.8.1 | 2.0.2 |
torchvision | 0.9.1+cu102 | 0.15.2 |
新增
networkx | 2.6.3 | 3.1 |
|---|---|---|
pandas | 1.2.3 | 2.1.1 |
pyHSICLasso | 1.4.2 | 1.4.2 |
seaborn | 0.12.2 | 0.13.0 |
statsmodels | 0.13.5 | 0.14.0 |
3. IDE
建议使用Pycharm(其中,pyHSICLasso库在VScode出错,尚未找到解决办法……)
win11 安装 Anaconda(2022.10)+pycharm(2022.3/2023.1.4)+配置虚拟环境_QomolangmaH的博客-CSDN博客
https://blog.csdn.net/m0_63834988/article/details/128693741
三、实验内容
1. 导入必要的库
from scipy.stats import hypergeom
import numpy as np
import pandas as pd
from statsmodels.stats.multitest import fdrcorrection
import matplotlib.pyplot as plt2. 定义一些变量
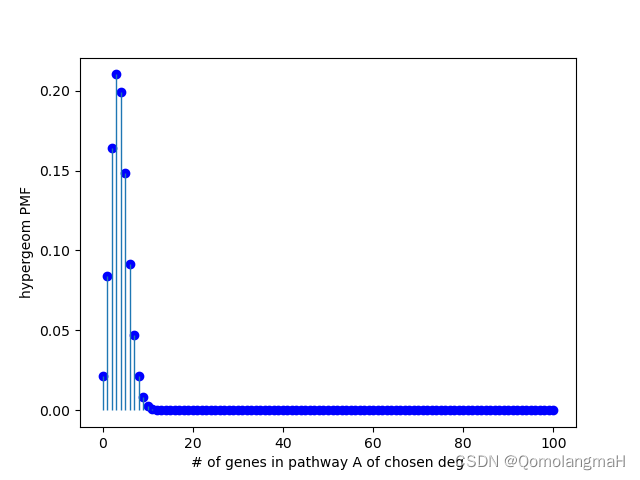
[M, n, N] = [8000, 100, 300]定义了三个变量,分别表示总基因数(M)、通路A基因数(n)和差异基因数(N)。
3. 计算富集分析的p值
x = np.arange(0, n + 1)
pmf_deg = []
for i in x:
pmf_deg.append(hypergeom.pmf(i, 8000, 100, 300))
p_A = np.sum(pmf_deg[30:300]) 使用超几何分布计算了富集分析的p值。循环计算了在不同基因数下的概率质量函数值,并将结果存储在pmf_deg列表中。最后,计算了在基因数为30到300之间的概率之和,即富集分析的p值。
4. 绘制概率质量函数图
fig = plt.figure()
ax = fig.add_subplot(111)
ax.plot(x, pmf_deg, 'bo')
ax.vlines(x, 0, pmf_deg, lw=1)
ax.set_xlabel('# of genes in pathway A of chosen deg')
ax.set_ylabel('hypergeom PMF')
plt.show()绘制概率质量函数图,展示了不同基因数下的概率分布情况。
5. 读取数据
all_pathway_canonica_GO_bp_unify = pd.read_csv("all_pathway_canonica_GO_bp_unify.csv", index_col=0, header=0)
deg_gene = pd.read_csv('deg_gene.csv', index_col=0, header=0)
gene_symbol = pd.read_table("gene_symbol_list.txt", header=None)all_pathway_canonica_GO_bp_unify = pd.read_csv("all_pathway_canonica_GO_bp_unify.csv", index_col=0, header=0)
deg_gene = pd.read_csv('deg_gene.csv', index_col=0, header=0)
gene_symbol = pd.read_table("gene_symbol_list.txt", header=None)
gene_symbol.columns = ['gene']
gene_symbol['label'] = 0
gene_symbol.index = gene_symbol['gene']
gene_symbol.loc[deg_gene['0'], 'label'] = 16. 定义富集分析函数
def Enrichment(gene_symbol, all_pathway_canonica_GO_bp_unify):
data = gene_symbol
up = data[data['label'] == 1].index.tolist()
all_pathway = all_pathway_canonica_GO_bp_unify
all_symbol = data.index
all_list = np.unique(all_pathway['V1'])
pathway_enrich_up = pd.DataFrame()
pvalue = []
for i in range(len(all_list)):
# 获取每一个通路的基因集合
gene_pathway = all_pathway[all_pathway['V1'] == all_list[i]]['V2']
# 通路基因与差异表达基因交集
up_no = len(set(gene_pathway).intersection(set(up)))
# 背景基因与通路基因的交集
hit_no = len(set(gene_pathway).intersection(set(all_symbol)))
# 做统计模型时,统计不是分布概率,而是累积概率,所以计算的p_value 值计算公式为P(X>x-1)或者 1- P(X≤x)
# sf(k, M, n, N) = 1-cdf
# cdf ,hypergeom.cdf表示:总共有M件产品,n件次品,从M件中随机挑出N件,这N件中最多包含n件中的k件的概率
# M is the total number of objects
# n is total number of Type I objects
# Scipy中的stats.hypergeom.sf(x, m+n, m, k)相当于R中的phyper(x, m, n, k, lower.tail=FALSE)(P X>x)
pval_up = hypergeom.sf(up_no - 1, len(all_symbol), hit_no, len(up))
pvalue.append(pval_up)
res = pd.DataFrame((all_list[i], pval_up, up_no, hit_no, set(gene_pathway).intersection(set(up)))).transpose()
pathway_enrich_up = pd.concat([pathway_enrich_up, res], axis=0)
pathway_enrich_up.columns = ['pathway', 'pval_up', 'up_no', 'hit_no', 'gene_list']
# 在富集分析中,每一个结果都进行了很多次的差异比较(一个通路一次),这种多重比较下的假阳性会急剧升高(这个假阳性的比例:FDR,false discovery rate,
# 其含义是拒绝零假设的事件中错误拒绝事件的所占比例),因此要校正p值。
# 举个栗子,如果检验1000次,设定的阈值为0.05(5%),那么无论得到多少个显著富集通路,这些显著富集通路出现假阳性的概率保持在2%之内,这就叫FDR<0.02
# BH: Benjamini & Hochberg法,简称BH法。将n个p值按照从小到大排序,k为p值的顺序值,找到一个最大的k使得p*n/k <α ,认为1,2,...k个通路是显著富集的
# 最大的p值保持不变,对于排名n-1的,p_adj(n-1) = min( p * n / k, p_adj(n))。其影响是除了最大的p值保持不变外,所有的其他p值都会被增加,变得没那么显著
fdr = fdrcorrection(pvalue, alpha=0.05, is_sorted=False)
p_adjust = fdr[1]
pathway_enrich_up['fdr_p_adjust'] = list(p_adjust)
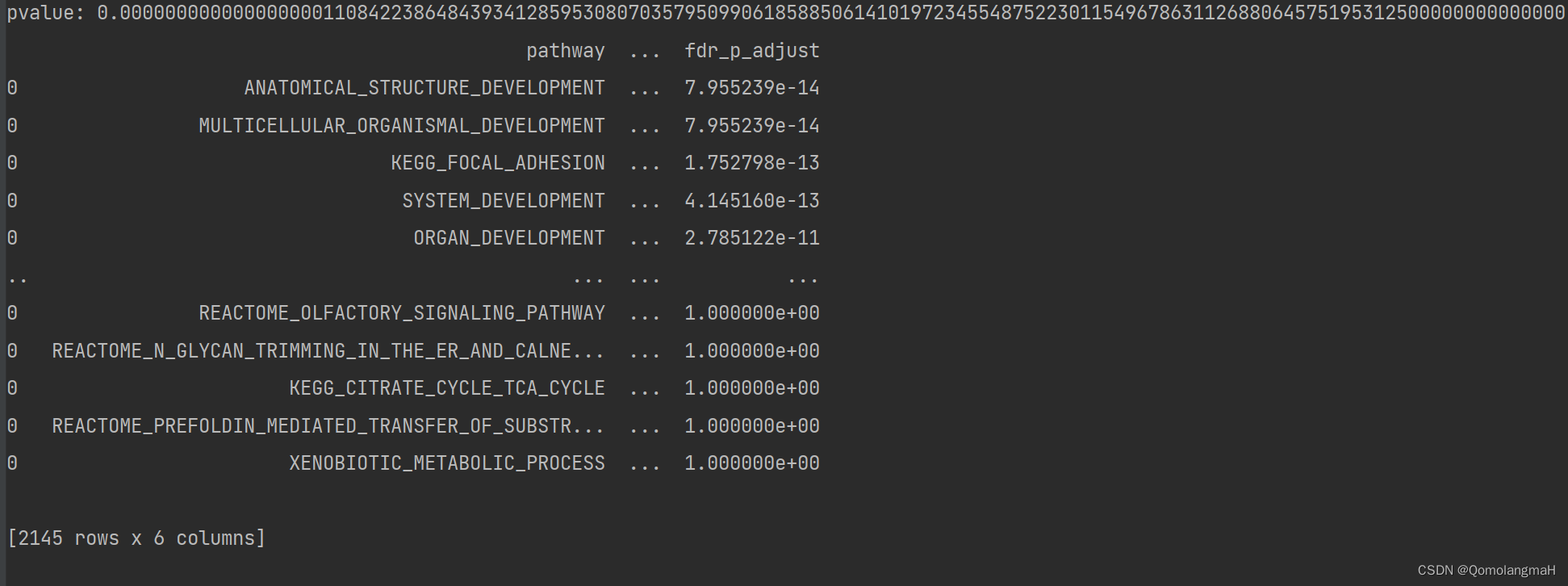
final_pathway_enrich_up = pathway_enrich_up.sort_values(by='pval_up', ascending=True)
return (final_pathway_enrich_up)7.富集分析
res = Enrichment(gene_symbol, all_pathway_canonica_GO_bp_unify) 调用Enrichment函数进行富集分析,并将结果存储在res变量中。
8. 将结果保存到文件并打印
res.to_csv('enrichment.csv')
print(res)将富集分析的结果保存到CSV文件中,并打印结果。
请注意,以上只是代码的分析和解释,并非在DAVID Functional Annotation Bioinformatics Microarray Analysis网站上的具体操作。要详细了解如何在该网站上进行功能注释和富集分析,请访问该网站并参考其提供的文档和教程。
四、DAVID Functional Annotation Bioinformatics Microarray Analysis
DAVID Functional Annotation Bioinformatics Microarray Analysis (ncifcrf.gov)
ChatGPT
DAVID(Database for Annotation, Visualization, and Integrated Discovery)是一个功能注释和生物信息学分析工具。它提供了一套强大的功能,用于解释和理解高通量生物学数据,例如基因表达谱、蛋白质组学和组织芯片数据等。 DAVID的功能注释模块可以帮助研究人员对基因或蛋白质列表进行功能注释和富集分析。它通过使用多种生物信息学数据库和资源,如Gene Ontology(GO)数据库、KEGG(Kyoto Encyclopedia of Genes and Genomes)通路数据库、PubMed文献数据库等,来解释和理解给定的基因或蛋白质集合。 使用DAVID进行功能注释分析的一般步骤如下:
- 输入数据:将感兴趣的基因或蛋白质列表输入到DAVID系统中。这些列表可以是通过实验获得的差异表达基因、蛋白质质谱数据或其他高通量数据分析得到的结果。
- 功能注释:DAVID将根据输入的基因或蛋白质列表,使用不同的数据库和算法进行功能注释。它会将输入的基因或蛋白质与已知的功能注释信息进行比较,包括基因本体论(Gene Ontology)注释、通路注释、疾病关联等。
- 富集分析:DAVID还会对输入的基因或蛋白质列表进行富集分析,以确定在给定的功能注释数据库中是否存在显著富集的功能条目。这有助于确定与特定生物学过程、分子功能或细胞组分相关的功能集合。
- 结果解释和可视化:DAVID提供了丰富的结果解释和可视化工具,以帮助研究人员理解分析结果。它可以生成图表、图形和交互式网络,以展示功能注释和富集分析的结果。

