转录组GSE122709—KEGG 富集不出?
原创转录组GSE122709—KEGG 富集不出?
在分析GSE122709时候,取D1组、D2组分别与NC组进行基因差异与富集的分析时候,遇到一个问题就是D2/NC比较,进行KEGG分析时候什么结果都没有。查找原因时候遇到了一些问题,这里做简单的记录。
1 D1 Vs NC
差异基因
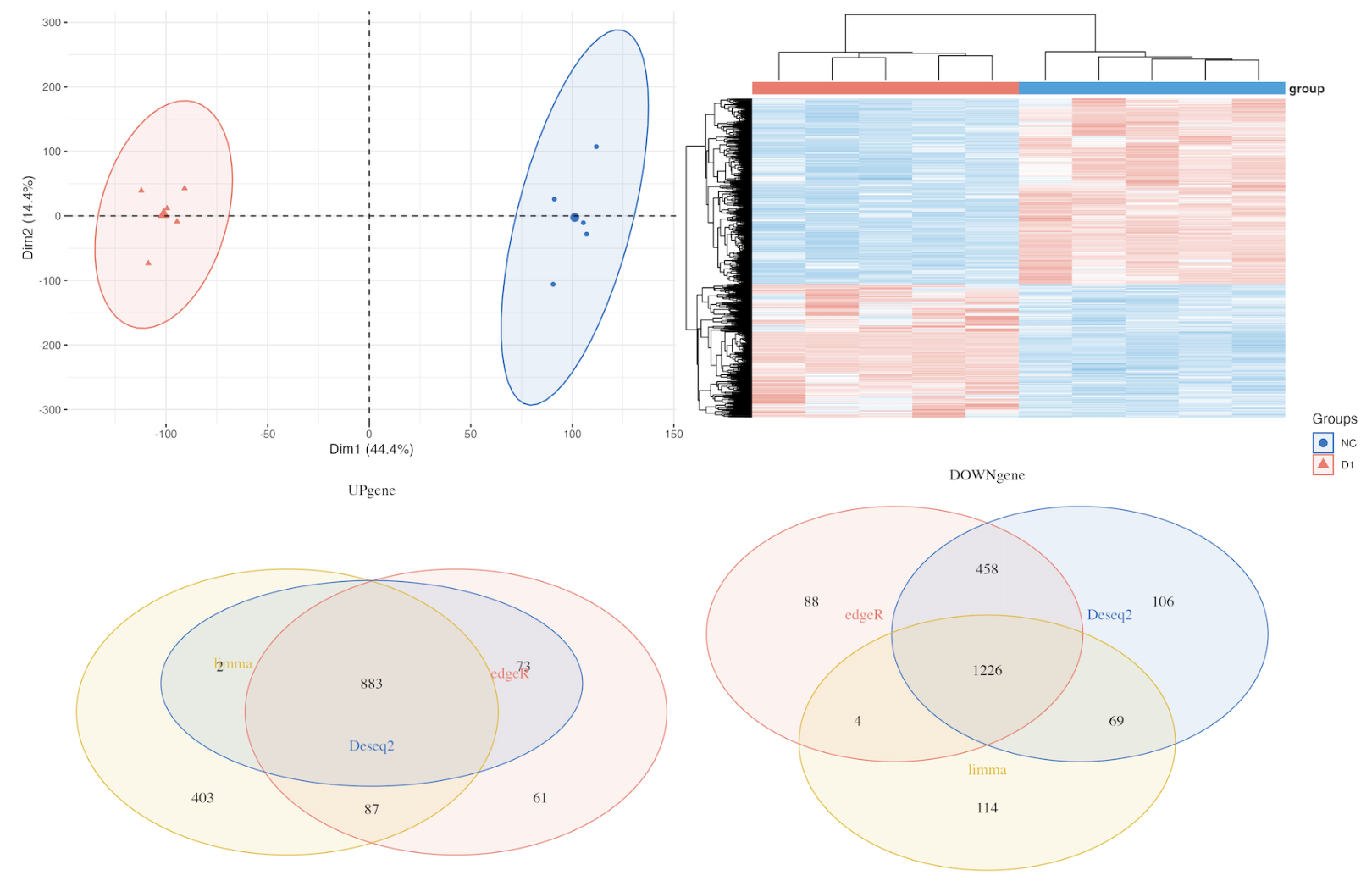
富集分析
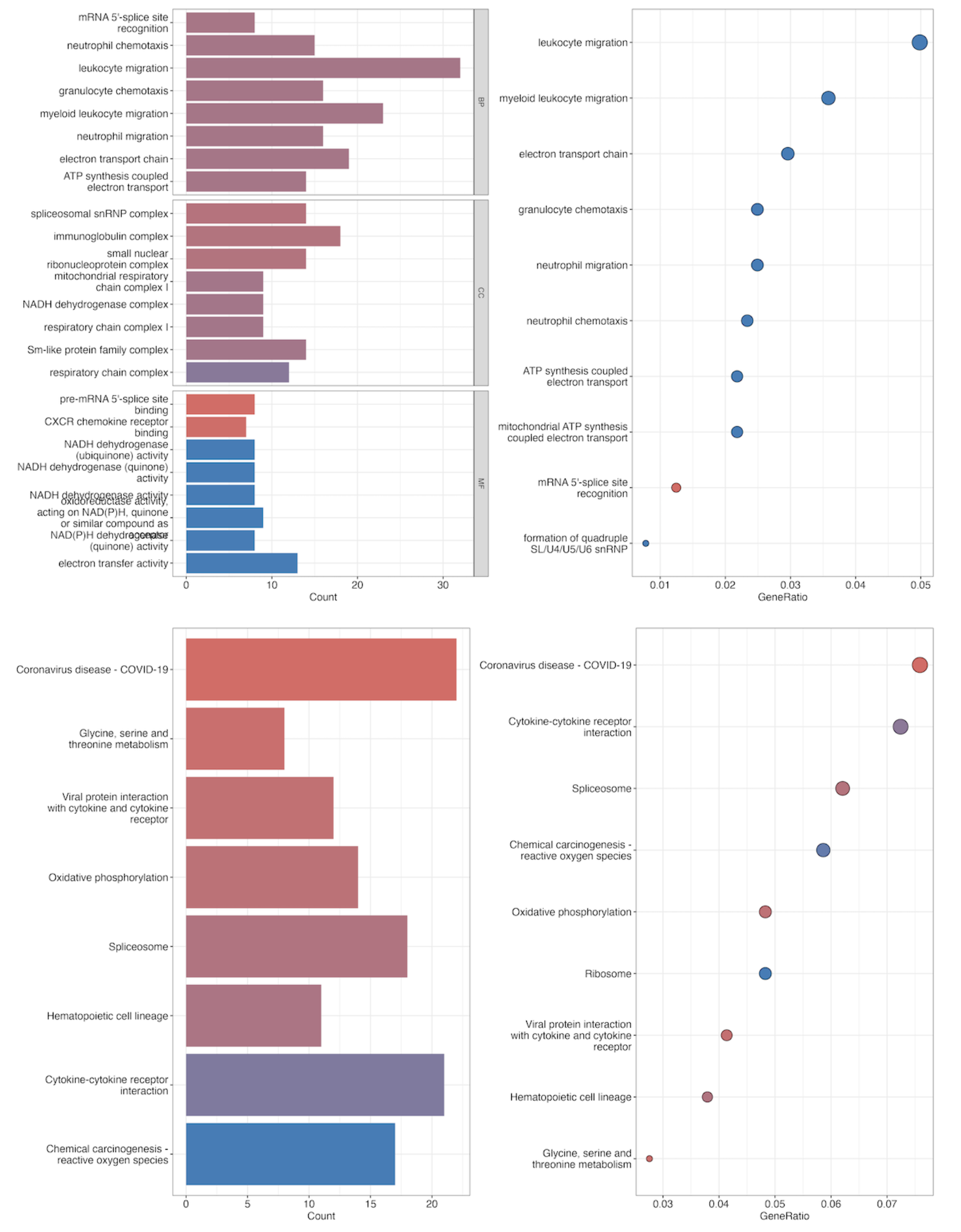
2 D2 Vs NC
差异基因
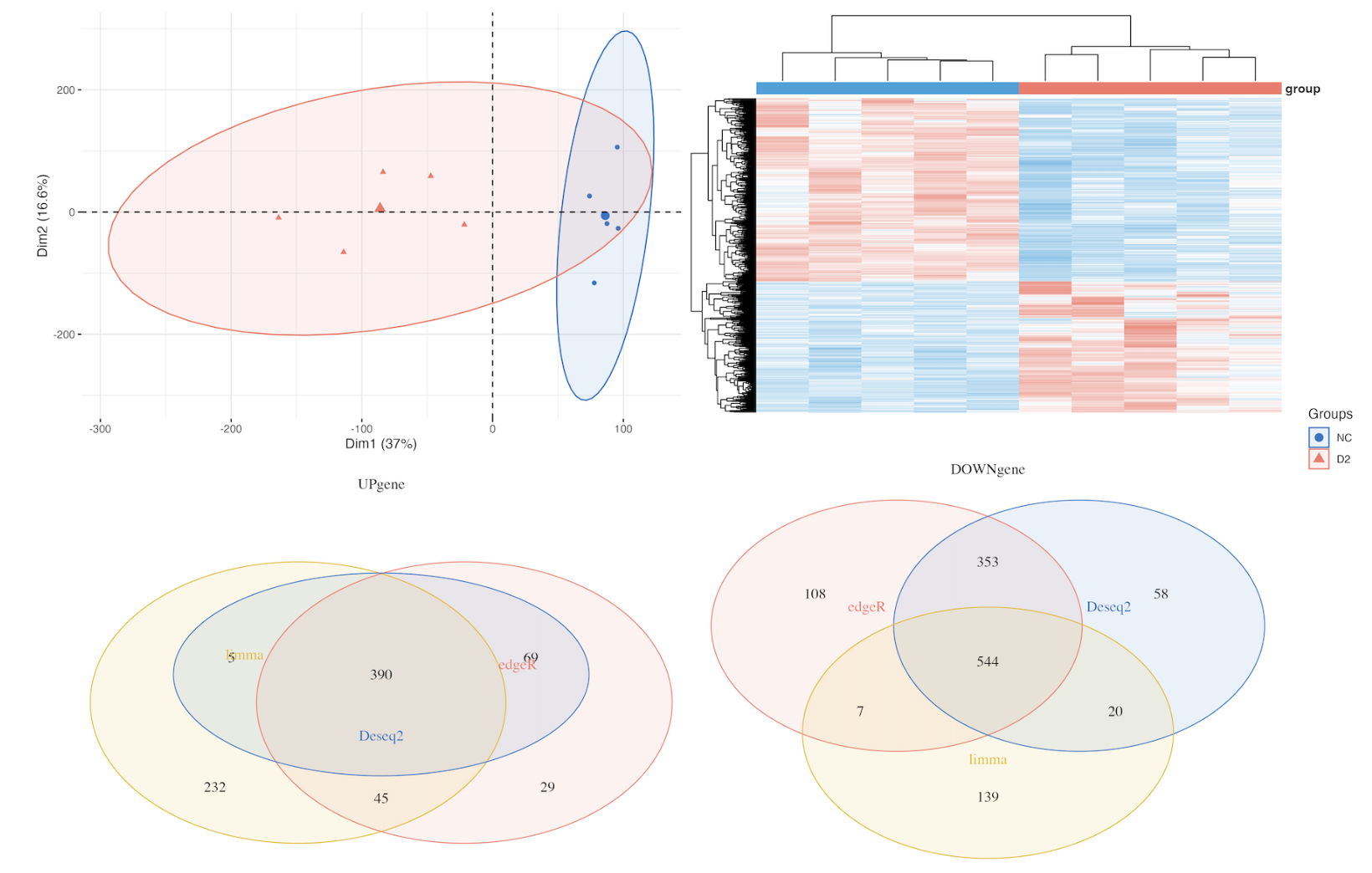
富集分析
#(1)输入数据
load("./D2/GSE122709_DEG.Rdata")
proj <- "GSE122709"
library(tinyarray)
g = intersect_all(rownames(DEG1)[DEG1$change!="NOT"],
rownames(DEG2)[DEG2$change!="NOT"],
rownames(DEG3)[DEG3$change!="NOT"])
output <- bitr(g,
fromType = 'SYMBOL',
toType = 'ENTREZID',
OrgDb = 'org.Hs.eg.db')
gene_diff = output$ENTREZID
#(2)富集
ekk <- enrichKEGG(gene = gene_diff,
organism = 'hsa')
ekk <- setReadable(ekk,
OrgDb = org.Hs.eg.db,
keyType = "ENTREZID")
#'select()' returned 1:many mapping between keys and columns
#Warning message:
#In bitr(g, fromType = "SYMBOL", toType = "ENTREZID", OrgDb = "org.Hs.eg.db") :
# 20.24% of input gene IDs are fail to map...
#可视化
p3 <- barplot(ekk)
p4 <- dotplot(ekk)注:
在进行可视化p3 <- barplot(ekk)时候报错:
Error in ansypos <- rep(yes, length.out = len)ypos :
replacement has length zero
In addition: Warning message:
In rep(yes, length.out = len) : 'x' is NULL so the result will be NULL
这种问题是没有富集到通路,因此可视化不了。
View(ekk@result),后查看kegg发现是有结果的,结果如下:
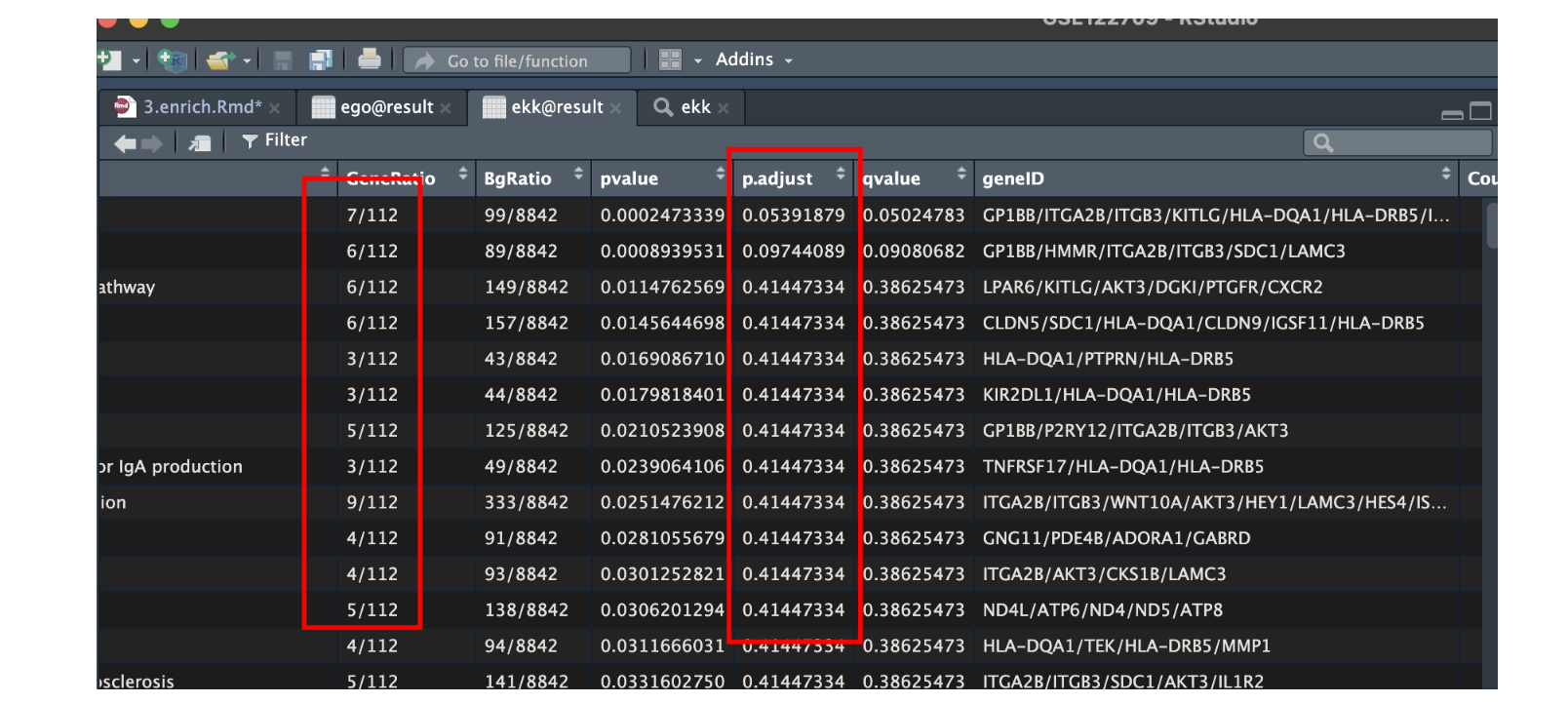
尝试的探索:
pvalue or p.adjust?
pathways <- subset(ekk@result, p.adjust< 0.05)
pathways
[1] category subcategory ID Description GeneRatio BgRatio pvalue
[8] p.adjust qvalue geneID Count
<0 rows> (or 0-length row.names)观察上述矩阵,可以看到pvalue还是比较小的,adjust pvalue偏大,因此筛选测试下,发现如果设置p.adjust < 0.05的话,一条都满足不了。这个说明,barplot()函数默认是取p.adjust的进行绘图的。
GeneRatio?
按照之前的学习,我知道GeneRatio,BgRatio是这样定义的:

因此不太理解为什么我用来做富集的基因只有112个?
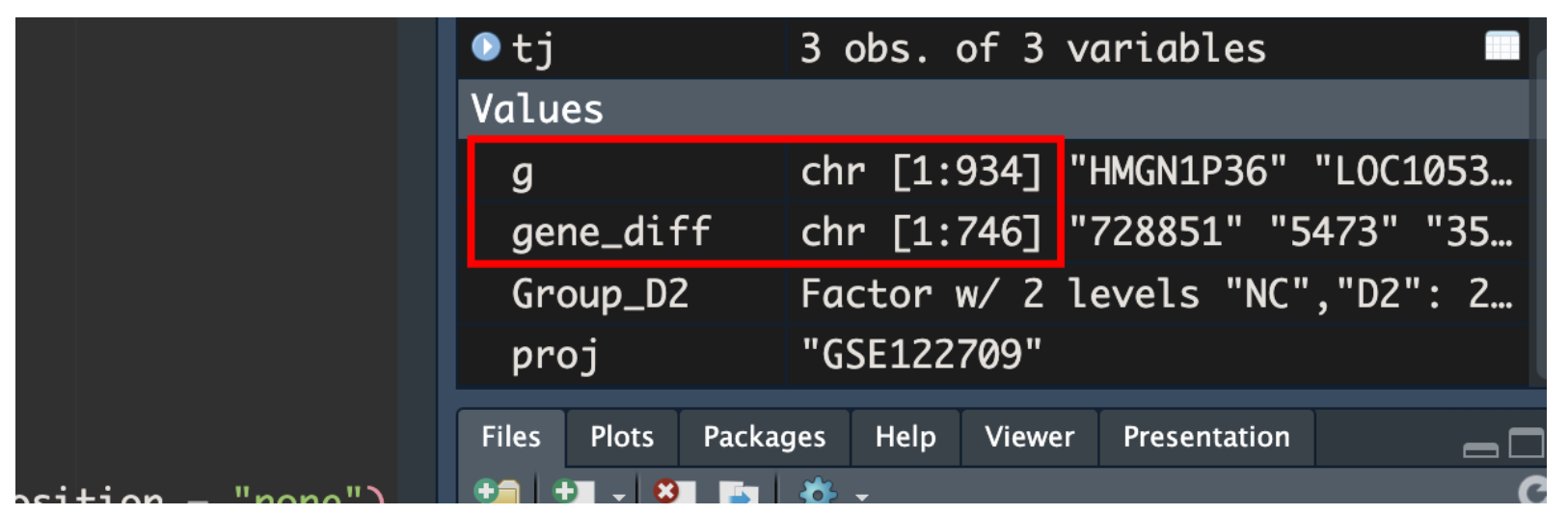
由图中可以看到:
用3个R包得到的差异基因交集g有934个对象,正好是差异基因中(390Up + 544Down),在进行ID转换后,一部分基因丢失后,gene_diff还剩746个基因才对。
那么在进行富集分析时
ekk <- enrichKEGG(gene = gene_diff,
organism = 'hsa')为什么出来的结果只有112呢?
猜想之一的是只有被kegg收录的基因才会被富集,那么挑出来的700个基因只有100个基因被收录到,这个运气也太差了。
想不明白就先记录下。
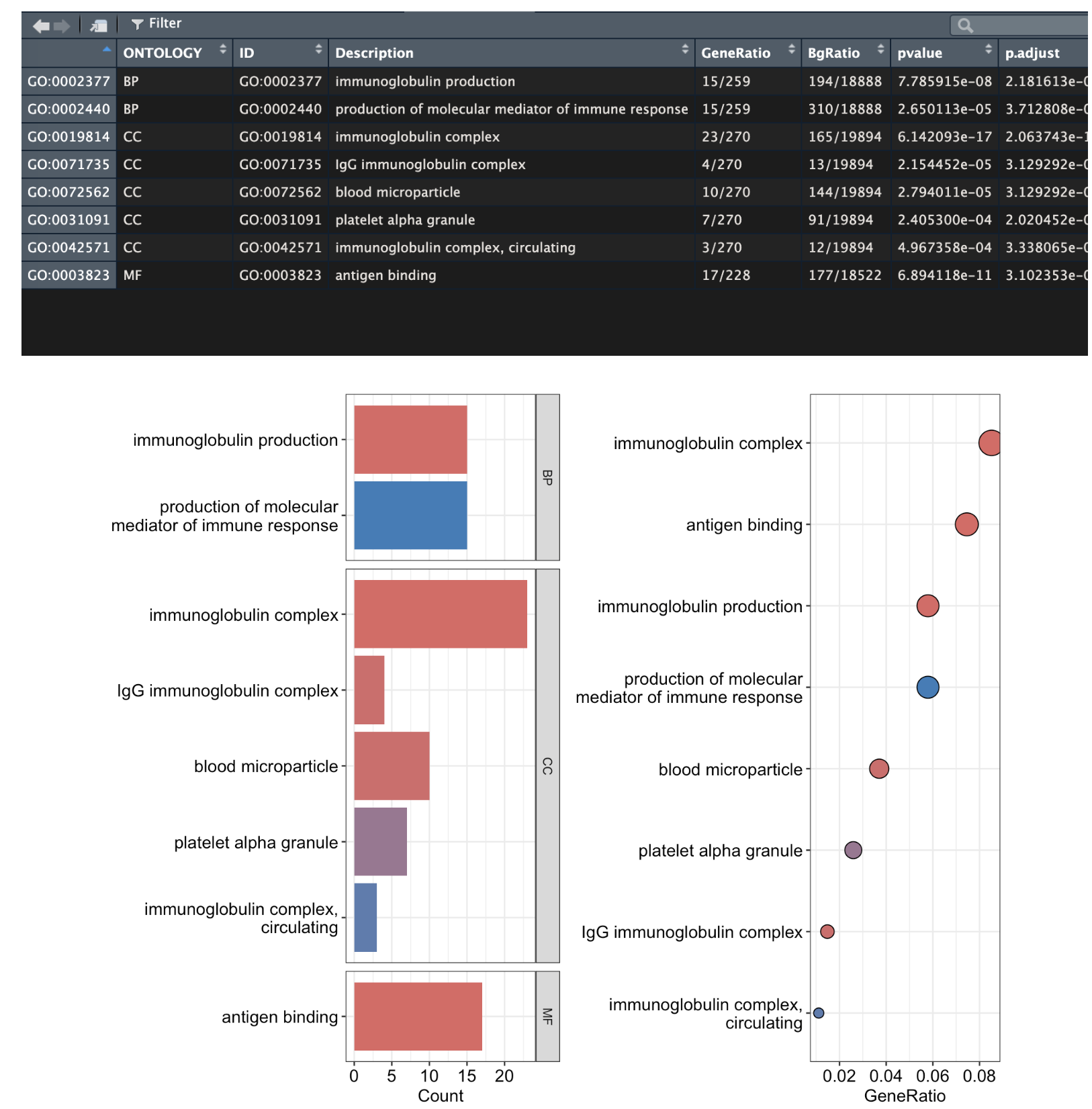
GO是可以出一点结果的:
原创声明:本文系作者授权腾讯云开发者社区发表,未经许可,不得转载。
如有侵权,请联系 cloudcommunity@tencent.com 删除。
原创声明:本文系作者授权腾讯云开发者社区发表,未经许可,不得转载。
如有侵权,请联系 cloudcommunity@tencent.com 删除。

