10X Visium(55um)进行高分辨率降维分析
原创10X Visium(55um)进行高分辨率降维分析
原创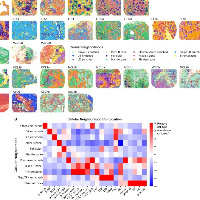
追风少年i
发布于 2024-09-23 15:36:24
发布于 2024-09-23 15:36:24
作者,Evil Genius
感受一下空间转录组精度分析上的变化
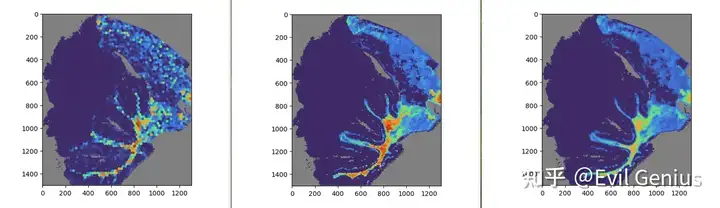
那么如何实现visium低精度实现高精度的数据分析呢?毕竟现在都是HD等高精度的天下了,分析也需要跟进。
10XVisium的空间分辨率仍然有限。每个ST spot通常包含多个细胞,并且测序spot没有覆盖大面积的组织。较新的技术,如Stereo-seq和Xenium,实现了更高的分辨率,但产生更稀疏和噪声数据。通常需要将spot聚合为较大的meta-spot,以提高信噪比,但不幸的是,这显著降低了空间分辨率。
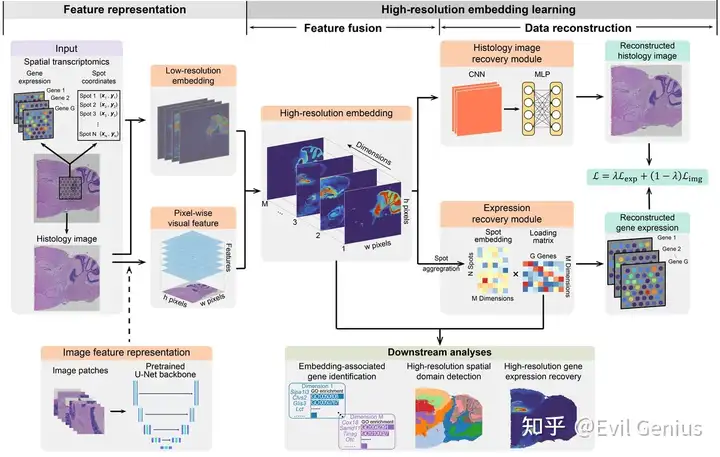
ST 数据是高维的,并且通常表现出相当大的噪声和稀疏性,因此需要将其作为分析的关键步骤进行降维。由于传统的降维方法没有考虑空间信息,因此最近开发了专门适用于ST数据的方法。通过利用空间信息,这些方法可以生成具有空间感知的低维表示,更好地保留细胞或spot之间的空间关系。
ST 数据通常伴随着高分辨率的组织学图像,如苏木精-伊红(H&E)染色图像或免疫荧光图像,这些图像在更高的分辨率下提供了有关组织形态细胞组成和空间组织的有价值的信息。
基因表达 + 组织学图像进行高分辨率降维
更精确的嵌入和空间聚类,多方法比较
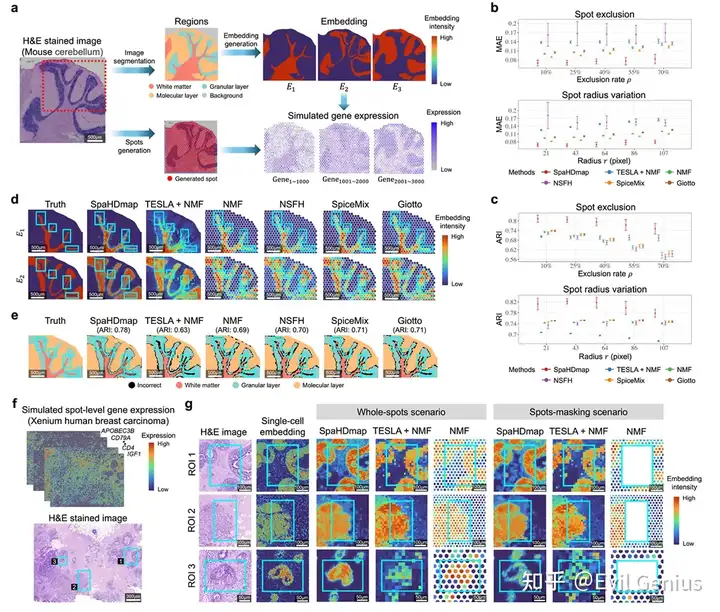
小鼠大脑数据集测试效果
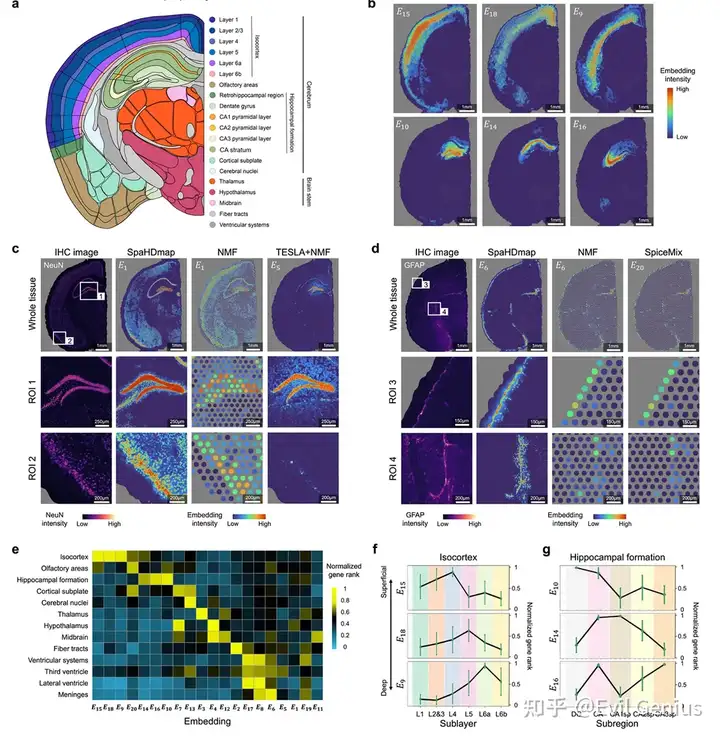
更精细的空间结构
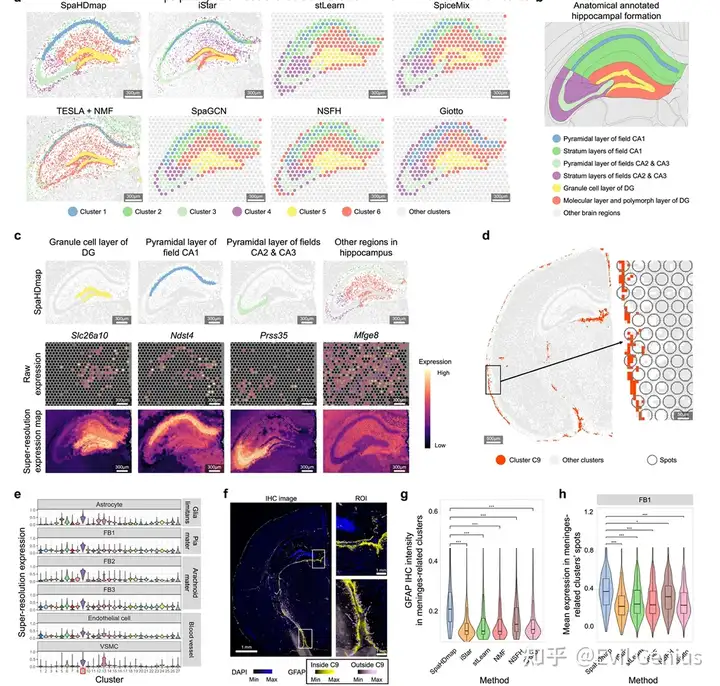
多样本的整合效果
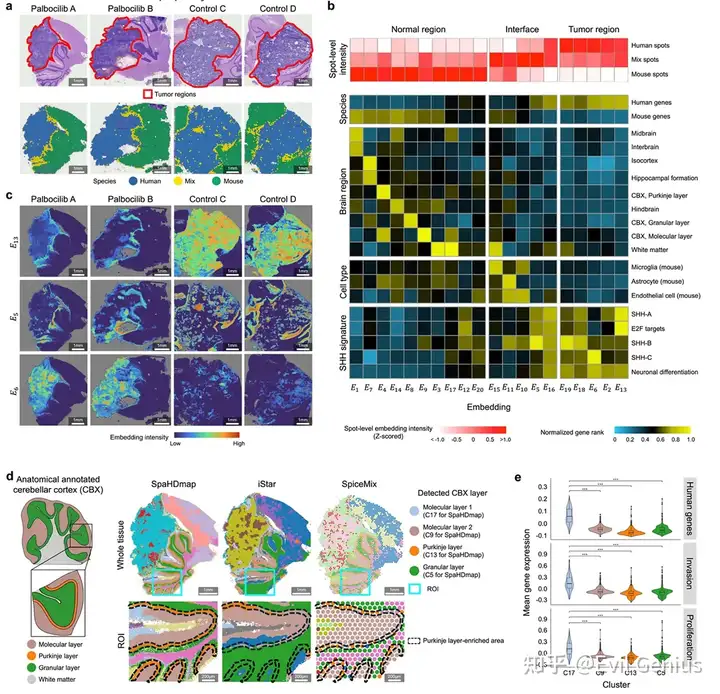
CRC样本中的肿瘤异质性和免疫活性
示例代码,10X数据
###pip install SpaHDmap
import torch
import numpy as np
import scanpy as sc
import SpaHDmap as hdmap
rank = 20
seed = 123
verbose = True
np.random.seed(seed)
torch.manual_seed(seed)
root_path = '../experiments/'
project = 'MPBS01'
results_path = f'{root_path}/{project}/Results_Rank{rank}/'
radius = 45
scale_factor = 1
# Load the data (This data has been preprocessed, including normalization, swapping the coordinates and selecting SVGs)
mouse_posterior = hdmap.prepare_stdata(section_name='mouse_posterior',
image_path='../data/MPBS01/HE.tif',
spot_coord_path='../data/MPBS01/spot_coord.csv', # The coordinates must be in the first two columns
spot_exp_path='../data/MPBS01/expression_nor.csv', # Has been normalized in this data
scale_factor=scale_factor,
radius=radius,
swap_coord=False) # Has been swapped in the data
section_id = 'V1_Mouse_Brain_Sagittal_Posterior'
# Download the data from the 10X website (set include_hires_tiff=True to download the hires image)
adata = sc.datasets.visium_sge(section_id, include_hires_tiff=True)
image_path = adata.uns["spatial"][section_id]["metadata"]["source_image_path"]
# or load the data from a local folder
# adata = sc.read_visium(f'data/{section_id}')
# image_path = f'data/{section_id}/image.tif'
# Load the data from the 10X Visium folder
mouse_posterior = hdmap.prepare_stdata(adata=adata,
section_name='mouse_posterior',
image_path=image_path,
scale_factor=scale_factor)
hdmap.select_svgs(mouse_posterior, n_top_genes=3000)
# Initialize the SpaHDmap runner
mapper = hdmap.Mapper(mouse_posterior, results_path=results_path, rank=rank, verbose=verbose)
# Run all steps in one function
mapper.run_SpaHDmap(save_score=False, save_model=True, visualize=True)
# Run NMF on concatenated data
mapper.get_NMF_score(save_score=False)
print(mouse_posterior.scores['NMF'].shape)
mapper.visualize(mouse_posterior, score='NMF', index=2)
# mapper.visualize('mouse_posterior', score='NMF', index=2) # visualize given the name
# mapper.visualize(score='NMF', index=2) # ignore the section name if only one section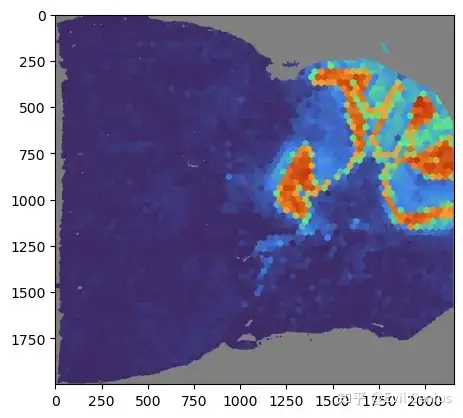
# Save all NMF scores into `results_path/section_name/NMF`
mapper.visualize(score='NMF')
# Pre-train the SpaHDmap model via reconstructing the HE image
mapper.pretrain(save_model=True)
# Train the GCN model and get GCN score
mapper.get_GCN_score(save_score=False)
print(mouse_posterior.scores['GCN'].shape)
# Visualize the GCN score
mapper.visualize(mouse_posterior, score='GCN', index=2)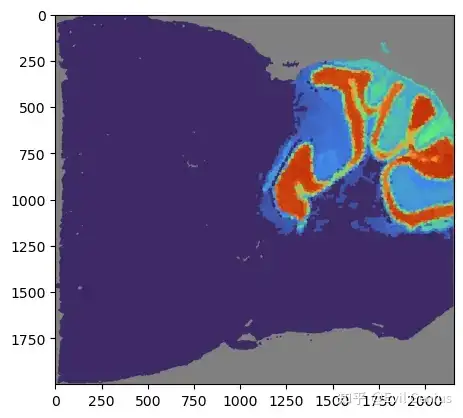
# Save all GCN scores into `results_path/section_name/GCN`
mapper.visualize(score='GCN')
# The refined metagene matrix based on the GCN score
print(mapper.metagene_GCN.shape)
# Get the VD score
mapper.get_VD_score(use_score='GCN')
# Train the SpaHDmap model
# If train_path is not empty, SpaHDmap will load the trained model from the train_path
mapper.train(save_model=True)
# Get the SpaHDmap score
mapper.get_SpaHDmap_score(save_score=False)
print(mouse_posterior.scores['SpaHDmap'].shape)
# Visualize the SpaHDmap score
mapper.visualize(mouse_posterior, score='SpaHDmap', index=2)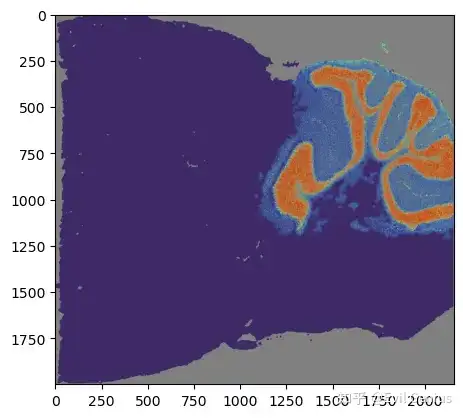
生活很好,有你更好
原创声明:本文系作者授权腾讯云开发者社区发表,未经许可,不得转载。
如有侵权,请联系 cloudcommunity@tencent.com 删除。
原创声明:本文系作者授权腾讯云开发者社区发表,未经许可,不得转载。
如有侵权,请联系 cloudcommunity@tencent.com 删除。
评论
登录后参与评论
推荐阅读
目录
