【拿捏热图/详细注释】pyComplexHeatmap系列(一)之基础绘制之差异基因聚类热图并标注关键基因
【拿捏热图/详细注释】pyComplexHeatmap系列(一)之基础绘制之差异基因聚类热图并标注关键基因

KS科研分享与服务-TS的美梦
发布于 2025-12-20 17:19:38
发布于 2025-12-20 17:19:38
热图是生物数据分析最常见、最炙手可热的数据展示形式,我们之前专门出过很多热图系列,以及衍生的热图修饰,五花八门,非常齐全,公众号检索‘热图’可以浏览基础用法以及个性化修饰,部分比较优秀的应用可参考:
复现NC图表:相关性分析气泡图(热图)---同时展示正负调控关系和显著性
nature级别图表:一个注释气泡热图函数(适用于单细胞及普通数据)
Monocle3个性化分析作图:拟时热图/拟时基因GO分析/拟时基因趋势分析
Complexheatmap有很多隐藏功能:marker基因平均表达量热图添加基因分面注释
(视频教程-复现Science图表)做热图无限添加文本注释及修饰
更新:单细胞基因热图多重注释函数-可标记基因及添加富集分析结果
【代码调整】复现《Cell》文章图表:气泡热图展示基因表达+分组添加
【视频-python一键函数】:热图标注特定基因及不同颜色设置展示marker基因
pyCirclize一个包可以干很多事【2】:基因/蛋白环形表达量热图+互作+分组注释
在R中热图主要由pheatmap、ComplexHeatmap、ggplot2以及ggheatmap等包完成,已经介绍过各种各样的形式,或者各种修饰;在很多小伙伴单细胞分析或者整合数据分析向python迁移的过程中,在数据可视化上也希望python能够轻松完成热图绘制。python中也有各种库具有绘制热图功能,例seaborn、matplotlib等,但是对于初次接触者或者灵活性而言,没有R中那么顺手。这里我们介绍一款专一的用于热图绘制的py库pyComplexHeatmap,可以将其理解为R版ComplexHeatmap包的py对应版,在使用上,功能上有很类似,且比较好理解。但是PyComplexHeatmap的底层绘图完全基于matplotlib。这里先介绍他的基础用法,以转录组差异基因数据展示为例:
Github: https://github.com/DingWB/PyComplexHeatmap
1-安装加载数据
#安装
pip install PyComplexHeatmap
import pandas as pd
import matplotlib.pyplot as plt
import numpy as np
import PyComplexHeatmap
from PyComplexHeatmap import * #导入所有类与函数#加载数据:这个数据基因有点多,选择部分进行演示
df = pd.read_csv('./df_heat.csv',index_col=0)
df = df.iloc[:30]
df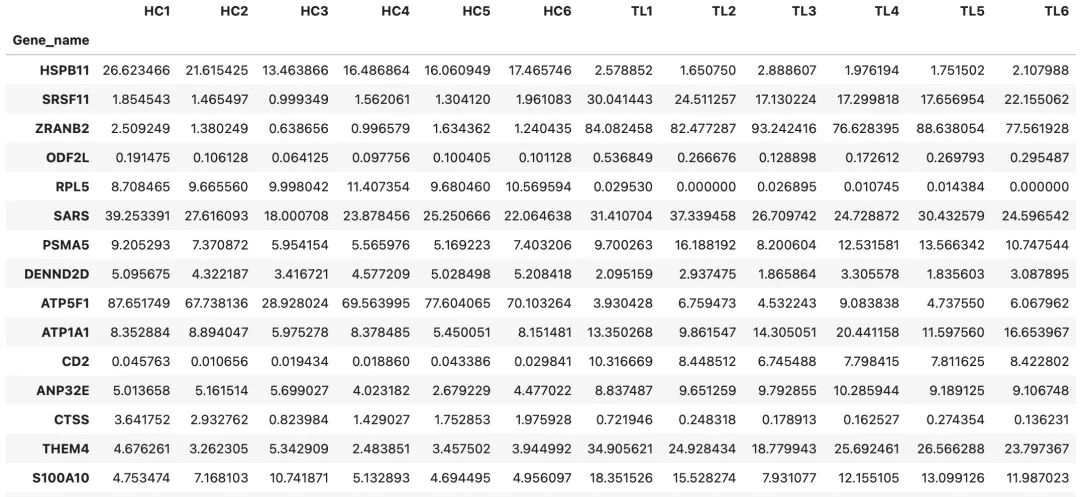
2-正常热图绘制,了解函数参数
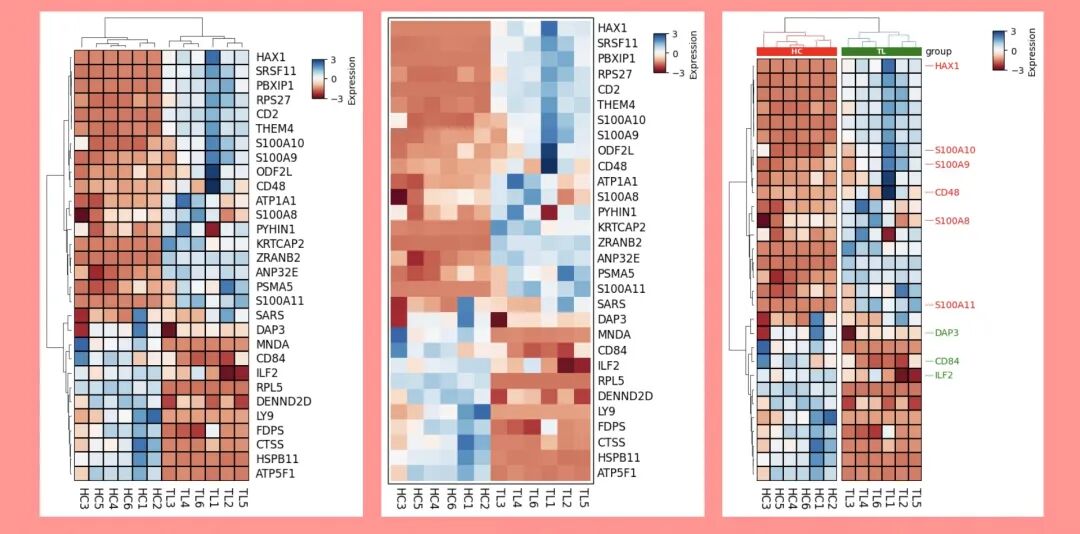
#轻松绘制一个简单的聚类热图
plt.figure(figsize=(4,8))#图宽、高
cm = ClusterMapPlotter(data=df, #表达矩阵
z_score=0,#是否对数据计算z-score,0表示对按行scale、1表示按列scale
col_cluster=True,#列聚类
row_cluster=True,#行聚类
row_dendrogram=True, #是否展示行聚类树
col_dendrogram=True,#是否展示列聚类树
show_rownames=True,#是否显示行名
show_colnames=True,#是否显示列名
row_names_side='right',#行名位置
col_names_side='bottom',#列名位置
label='Expression',#热图legend标题
#plot_legend=False, #是否plot热图legend
legend_kws={'vmax':3,'vmin':-3},#字典,设置legend相关修饰参数
cmap='RdBu',#热图颜色设置,plt.colormaps()查看,有百种选择
xticklabels_kws={'labelrotation':-90,'labelcolor':'black','labelsize':12},#字典,x轴标签设置
yticklabels_kws = {'labelcolor':'black','labelsize':12},#字典,y轴标签设置
linewidths=0.01,#热图格子线粗细,设置0则不展示
linecolor='black'#热图格子线条颜色
)
##pyComplexHeatmap目前没有参数能够为整个热图外部添加边框
##不过可以使用plt添加,不要展示聚类树
plt.figure(figsize=(4,8))#图宽、高
cm = ClusterMapPlotter(data=df, #表达矩阵
z_score=0,#是否对数据计算z-score,0表示对按行scale、1表示按列scale
col_cluster=True,#列聚类
row_cluster=True,#行聚类
row_dendrogram=False, #是否展示行聚类树
col_dendrogram=False,#是否展示列聚类树
show_rownames=True,#是否显示行名
show_colnames=True,#是否显示列名
row_names_side='right',#行名位置
col_names_side='bottom',#列名位置
label='Expression',#热图legend标题
#plot_legend=False, #是否plot热图legend
legend_kws={'vmax':3,'vmin':-3},#字典,设置legend相关修饰参数
cmap='RdBu',#热图颜色设置,plt.colormaps()查看,有百种选择
xticklabels_kws={'labelrotation':-90,'labelcolor':'black','labelsize':12},#字典,x轴标签设置
yticklabels_kws = {'labelcolor':'black','labelsize':12},#字典,y轴标签设置
)
heatmap_ax = cm.ax
# 为热图添加边框
heatmap_ax.set_frame_on(True) # 显示边框
heatmap_ax.patch.set_edgecolor('black') # 边框颜色
heatmap_ax.patch.set_linewidth(1) # 边框宽度
plt.show()
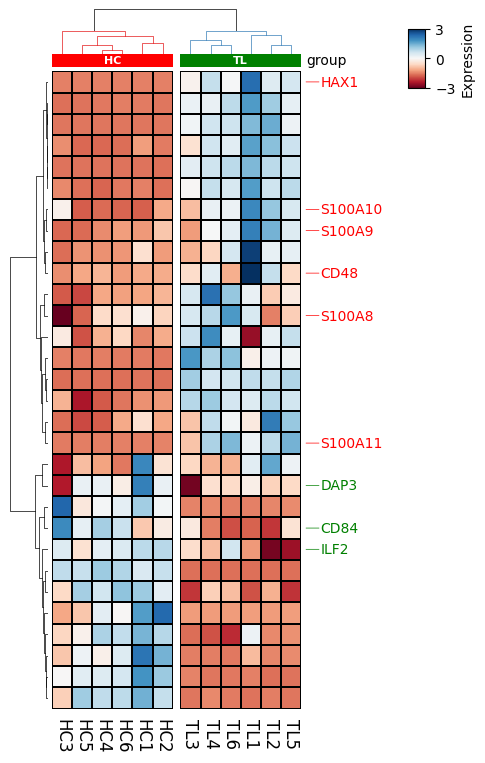
3-标注感兴趣基因+分组注释
很多时候,展示的基因过多,我们只希望展示一些感兴趣的基因
#比如这些基因我们需要label
lable_genes= ['S100A8','S100A9','S100A10','S100A11', 'HAX1','CD48',
'CD84','ILF2','CSTT','DAP3']
#还可以设置不同的颜色展示
colors_dict={}
for g in lable_genes:
if g in ['S100A8','S100A9','S100A10','S100A11', 'HAX1','CD48']:
colors_dict[g]='red'
else:
colors_dict[g]='green'
label_rows = df.apply(lambda x:x.name if x.name in lable_genes else None,axis=1)
label_rows #将label作为注释添加到热图
row_ha_right = HeatmapAnnotation(
selected=anno_label(label_rows, colors=colors_dict,relpos=(0,0.5)),
axis=0,verbose=1,
label_kws={'rotation':0,'horizontalalignment':'right','verticalalignment':'top', },
orientation='right'
)
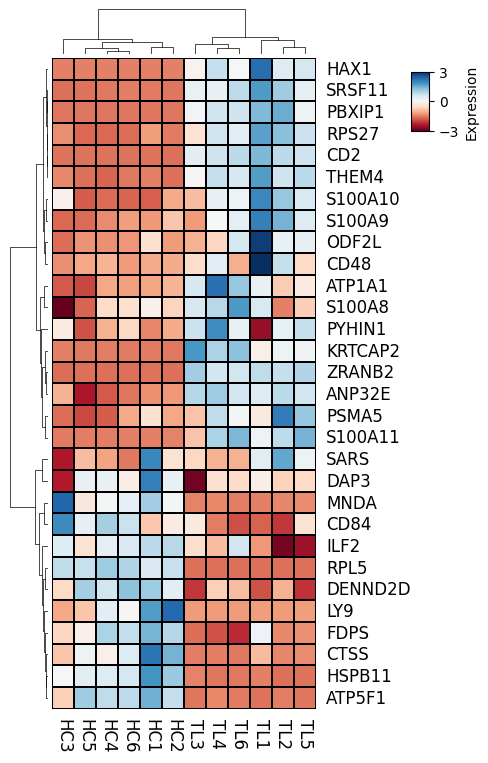
plt.figure(figsize=(4,8))#图宽、高
cm = ClusterMapPlotter(data=df, #表达矩阵
z_score=0,#是否对数据计算z-score,0表示对按行scale、1表示按列scale
col_cluster=True,#列聚类
row_cluster=True,#行聚类
row_dendrogram=True, #是否展示行聚类树
col_dendrogram=True,#是否展示列聚类树
show_rownames=True,#是否显示行名
show_colnames=True,#是否显示列名
row_names_side='right',#行名位置
col_names_side='bottom',#列名位置
label='Expression',#热图legend标题
#plot_legend=False, #是否plot热图legend
legend_kws={'vmax':3,'vmin':-3},#字典,设置legend相关修饰参数
cmap='RdBu',#热图颜色设置,plt.colormaps()查看,有百种选择
xticklabels_kws={'labelrotation':-90,'labelcolor':'black','labelsize':12},#字典,x轴标签设置
yticklabels_kws = {'labelcolor':'black','labelsize':12},#字典,y轴标签设置
linewidths=0.01,#热图格子线粗细,设置0则不展示
linecolor='black',#热图格子线条颜色
right_annotation=row_ha_right
)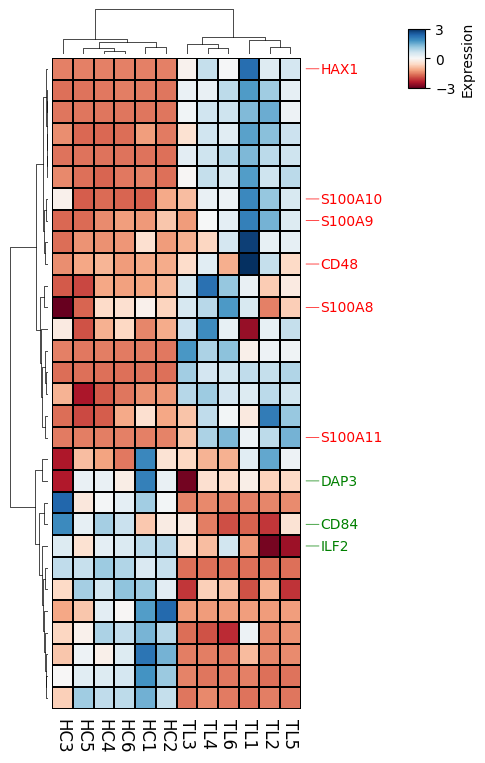
对列进行注释,首先需要生成一个列分组文件,对于行的注释是一样的操作
sample_group = {
"sample": df.columns.to_list(),
"group": ["HC"] * 6 + ["TL"] * 6
}
sample_group = pd.DataFrame(sample_group)
sample_group = sample_group.set_index(sample_group.columns[0])top_anno = HeatmapAnnotation(
group =anno_simple(sample_group.group,#分组信息
colors={'HC': 'red', 'TL': 'green'},#颜色设置
add_text=True,#添加分组名称
legend=False,#不展示legend
height=3,#注释高度
text_kws={'color':'white', 'fontsize':8,'fontweight':'bold',
'rotation':0})
)plt.figure(figsize=(4,8))#图宽、高
cm = ClusterMapPlotter(data=df, #表达矩阵
z_score=0,#是否对数据计算z-score,0表示对按行scale、1表示按列scale
col_cluster=True,#列聚类
row_cluster=True,#行聚类
row_dendrogram=True, #是否展示行聚类树
col_dendrogram=True,#是否展示列聚类树
show_rownames=True,#是否显示行名
show_colnames=True,#是否显示列名
row_names_side='right',#行名位置
col_names_side='bottom',#列名位置
label='Expression',#热图legend标题
#plot_legend=False, #是否plot热图legend
legend_kws={'vmax':3,'vmin':-3},#字典,设置legend相关修饰参数
cmap='RdBu',#热图颜色设置,plt.colormaps()查看,有百种选择
xticklabels_kws={'labelrotation':-90,'labelcolor':'black','labelsize':12},#字典,x轴标签设置
yticklabels_kws = {'labelcolor':'black','labelsize':12},#字典,y轴标签设置
linewidths=0.01,#热图格子线粗细,设置0则不展示
linecolor='black',#热图格子线条颜色
right_annotation=row_ha_right,
top_annotation=top_anno,
col_split=sample_group['group'],
col_split_gap=2,
tree_kws={'col_cmap': 'Set1','colors':'black'}
)
本文参与 腾讯云自媒体同步曝光计划,分享自微信公众号。
原始发表:2025-11-23,如有侵权请联系 cloudcommunity@tencent.com 删除
评论
登录后参与评论
推荐阅读
目录

