手把手教你使用ClaudeCode 4.5、Codex 5.2、Gemini 3,轻松玩转生信分析。(内附:一键自动安装工具)
手把手教你使用ClaudeCode 4.5、Codex 5.2、Gemini 3,轻松玩转生信分析。(内附:一键自动安装工具)
用户11203141
发布于 2025-12-25 12:40:32
发布于 2025-12-25 12:40:32
做生信的你,是不是经常陷入这样的 “至暗时刻” :
- 面对浩如烟海的 Linux 命令行,手指在键盘上悬停半天,最后还是打开了百度?
- R 语言包依赖冲突,红色的 Error 满屏飞,环境配置了一整天,连数据都没读进去?
- 好不容易跑出了结果,老板嫌图丑:“这个火山图的配色能不能高级一点?字体能不能大一点?”
- 想要复现顶刊 Paper 的分析流程,下载了 GitHub 源码却完全看不懂,仿佛在看天书?
生信圈流传着一句话: “生信分析,20% 在做生物学思考,80% 在和报错互搏。”
编程实现的鸿沟,难道真的要成为我们探索生命奥秘的绊脚石吗?
今天,我们向你介绍一款颠覆性的 AI 编程神器 —— AICoder。(文末附2分钟即可完成的零门槛搭建教程)PC电脑、服务器均可轻松搭建。
01、AICoder 是什么?
AICoder 是集成 Claude Code、Codex CLI、Gemini CLI,全球三大 AI 的聚合工具。
AICoder 可以运行在哪里? 本地电脑以及服务器上均可以。
AICoder 和网页端 AI 的区别是什么? 网页端编写代码或执行什么操作,只能从网页上复制,然后再粘贴。 AICoder 可以直接运行在你的电脑或服务器上,真实地帮你完成各种操作。
很多 AI 只能给你提供代码建议,剩下的复制、粘贴、报错、调试还得你自己来。 AICoder 的不同之处在于“执行力”——它接入了 Claude、Gemini 和 GPT 的能力,但它直接运行在你的服务器或本地电脑上。
它不只“懂”怎么做,还能帮你把事情“做”完。
02、为什么要选择AIcoder?
超简单的零门槛配置搭建教程
相较于传统 AI 编程助手安装包繁多、环境配置流程复杂的痛点,AIcoder 聚焦新手友好型配置体验。我们推出专属一键配置安装包,确保零编程基础的科研人员也能在 2 分钟内完成环境部署,快速投入生信分析工作。
生信分析的“自动驾驶”:从环境到结果
做生信最怕的不是分析本身,而是配环境和洗数据。
对于小白:你不需要再去纠结 Conda 怎么装、软件版本怎么对齐。你只需要说“我要跑一个 RNA-seq 流程”,AICoder 就能帮你搞定环境配置,调用 STAR、FeatureCounts 等工具,直接生成结果。
对于专家:它能直接读取你的服务器文件,帮你处理繁琐的数据清洗、格式转换(如 Seurat 对象转格式),或者批量处理几百个样本。它就像一个不需要休息的科研助理。
沉浸式开发:它写代码,也修 Bug
告别在网页版 AI 和代码编辑器之间反复横跳的低效模式。
现场调试:当代码报错时,你不需要把错误日志复制出来问 AI。AICoder 能直接读取报错信息,结合上下文代码,分析出是依赖问题还是逻辑漏洞,并直接给出可运行的修复方案。
实战教学:对于初学者,它不仅是干活的工具,更是最好的老师。看着它如何从零构建一个规范的项目结构、如何写出优雅的 Python/R 函数,是学习编程最快的方式。
安全与掌控:数据不出域,工作更安心
AICoder 运行在你的本地环境,你可以让它处理私有的临床数据或未发表的课题结果,而无需将原始数据上传到云端对话框。它既有云端大模型的智能,又保留了本地操作的安全感和掌控力。
02、实战演练(手把手教学)
接下来我们使用Claude code来做宏基因组数据质控的实战项目
一.准备工作
我们先来明确质控的目标和工具选择。宏基因组测序数据庞大且复杂,数据质量直接决定了后续分析的可靠性。因此,质控的核心在于两个字:评估和过滤。
- 质量评估与报告:在过滤之前,需要知道原始数据的真实情况。
FastQC和MultiQC是首选。FastQC专注于对单个或多个测序文件进行质量评估,生成详细的HTML报告;MultiQC能将多个FastQC甚至Trimmomatic等工具的报告聚合起来,生成一份直观、易懂的综合报告。 - 质量过滤与修剪:既然发现了问题,就要有“手术刀”来解决。
Trimmomatic,Fastp,Trim Galore都是常用的过滤工具。其中,Trimmomatic以其丰富的剪切模式和高效的处理能力而闻名。 我们这次选择Trimmomatic作为核心过滤工具,配合FastQC和MultiQC组成一个完整的质控闭环。 - 本次实践,我们选择了四组具有代表性的样本:骆驼、河北羊肺、健康乳腺组织和病变乳腺组织。
二、 开始分析
Claude code(AI coder)将质控流程被清晰地分解为四个步骤,每一步都有明确的目的和输出。
Update Todos
⎿ ☒ Run FastQC on raw data (already done)
☐ Run Trimmomatic filtering with specified parameters
☐ Run FastQC on filtered data
☐ Run MultiQC to aggregate all reports
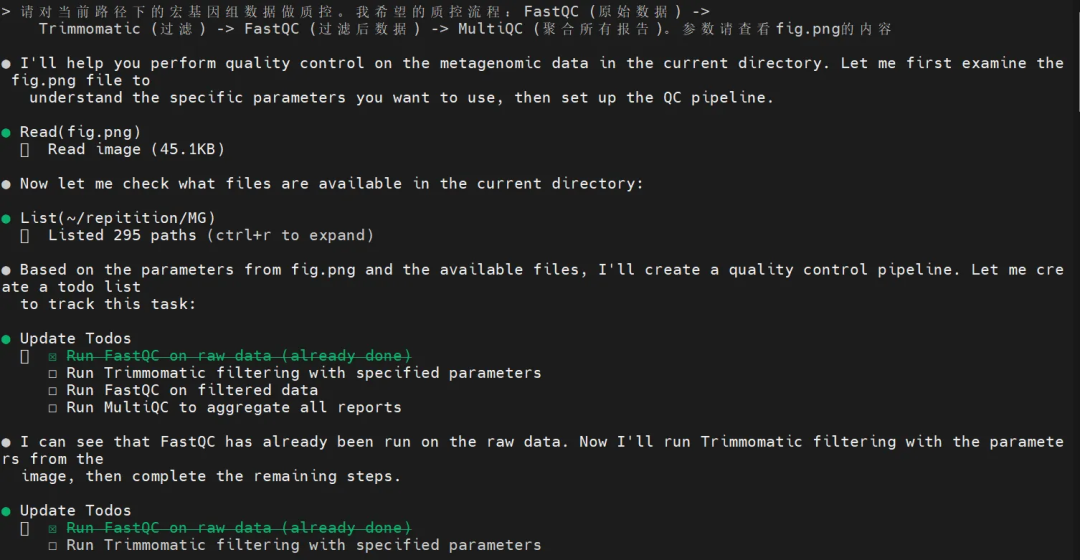
Claude code执行完成之后,我继续让他生成一份流程记录:
# 宏基因组数据质控分析流程记录
## 项目信息
- **分析日期**: 2025-08-22
- **分析目录**: `/mnt/data/home/sunting/repitition/MG`
- **数据类型**: 宏基因组双端测序数据
- **样本数量**: 4个样本,共8个fastq文件
## 样本信息
| 样本ID | R1文件 | R2文件 | 样本描述 |
|--------|--------|--------|----------|
| Alq_camel_46 | Alq_camel_46_1.fq.gz | Alq_camel_46_2.fq.gz | 骆驼样本 |
| Ch_breast_good_98_RNA | Ch_breast_good_98_RNA_1.fq.gz | Ch_breast_good_98_RNA_2.fq.gz | 健康乳腺RNA样本 |
| Ch_breast_sick_97_RNA | Ch_breast_sick_97_RNA_1.fq.gz | Ch_breast_sick_97_RNA_2.fq.gz | 病变乳腺RNA样本 |
| Hebei_sheep_lungD | Hebei_sheep_lungD_1.fq.gz | Hebei_sheep_lungD_2.fq.gz | 河北羊肺部样本 |
## 质控流程
### 步骤1: FastQC原始数据质控
**目的**: 评估原始测序数据质量
**命令**:
```bash
# 已在fastqc_raw/目录中完成
fastqc -o fastqc_raw *.fq.gz
```
**输出文件**:
- `fastqc_raw/` 目录包含每个样本的HTML和ZIP报告文件
- 共生成16个报告文件(每个fastq文件对应HTML和ZIP文件各一个)
### 步骤2: Trimmomatic数据过滤
**目的**: 去除低质量序列和接头序列
**参数设置**(基于fig.png):
- `LEADING:3` - 去除R1/R2开头质量低于3的碱基
- `TRAILING:3` - 去除R1/R2结尾质量低于3的碱基
- `MINLEN:36` - 丢弃长度小于36 bp的reads
- `HEADCROP:13` - 从reads开头剪掉13个碱基
**命令示例**:
```bash
trimmomatic PE -threads 8 \
input_R1.fq.gz input_R2.fq.gz \
output_R1_paired.fq.gz output_R1_unpaired.fq.gz \
output_R2_paired.fq.gz output_R2_unpaired.fq.gz \
LEADING:3 TRAILING:3 MINLEN:36 HEADCROP:13
```
**过滤结果统计**:
| 样本ID | 输入reads数 | 保留reads数 | 保留率 | 丢弃reads数 | 丢弃率 |
|--------|-------------|-------------|--------|-------------|--------|
| Alq_camel_46 | 1,000,000 | 998,278 | 99.83% | 1,722 | 0.17% |
| Ch_breast_good_98_RNA | 1,000,000 | 998,795 | 99.88% | 1,204 | 0.12% |
| Ch_breast_sick_97_RNA | 1,000,000 | 999,299 | 99.93% | 701 | 0.07% |
| Hebei_sheep_lungD | 1,000,000 | 999,950 | 100.00% | 50 | 0.01% |
**输出文件**:
- `trimmomatic_out/` 目录包含过滤后的配对和非配对文件
- 每个样本生成4个文件:R1配对、R1非配对、R2配对、R2非配对
### 步骤3: FastQC过滤后数据质控
**目的**: 评估过滤后数据质量
**命令**:
```bash
fastqc -o fastqc_clean -t 8 trimmomatic_out/*_paired.fq.gz
```
**输出文件**:
- `fastqc_clean/` 目录包含过滤后配对reads的FastQC报告
- 共8个样本的质控报告(仅配对reads)
### 步骤4: MultiQC聚合报告
**目的**: 整合所有质控结果,生成综合报告
**命令**:
```bash
multiqc fastqc_raw fastqc_clean trimmomatic_out -o multiqc_report
```
**输出文件**:
- `multiqc_report/multiqc_report.html` - 主要的HTML综合报告
- `multiqc_report/multiqc_data/` - 包含所有统计数据的目录
## 质控结果概览
### 数据质量评估
1. **序列质量**: 所有样本的质量评分普遍较高
2. **过滤效果**: Trimmomatic有效去除了低质量序列,保留率均在99.8%以上
3. **数据完整性**: Hebei_sheep_lungD样本质量最佳,保留率达到100%
4. **批次效应**: 不同样本间质量相对一致,无明显批次效应
### 推荐后续分析
基于质控结果,所有样本数据质量良好,可以进行后续分析:
1. 宿主序列去除
2. 宏基因组组装
3. 基因预测与功能注释
4. 物种分类学分析
5. 功能基因组学分析
## 文件目录结构
```
MG/
├── 原始数据文件 (*.fq.gz)
├── fastqc_raw/ # 原始数据FastQC报告
├── trimmomatic_out/ # Trimmomatic过滤输出
├── fastqc_clean/ # 过滤后数据FastQC报告
├── multiqc_report/ # MultiQC综合报告
│ ├── multiqc_report.html
│ └── multiqc_data/
└── 工具文件 (FastQC/, Trimmomatic-0.39.jar等)
```
## 技术参数记录
- **FastQC版本**: 本地安装版本
- **Trimmomatic版本**: 系统安装版本
- **MultiQC版本**: v1.30
- **Java版本**: OpenJDK (系统默认)
- **线程数**: 8个线程用于FastQC和Trimmomatic
- **质量编码**: Phred33
## 分析完成时间
- **开始时间**: 2025-08-22
- **结束时间**: 2025-08-22
- **总耗时**: 约30分钟
## 备注
- 所有原始数据均为压缩格式(.fq.gz)
- Trimmomatic的TruSeq3-PE.fa接头文件未找到,但其他过滤参数正常执行
- 建议定期检查MultiQC报告以监控数据质量趋势三、成果展示:质控报告的解读
样本概览(General Statistics 表)
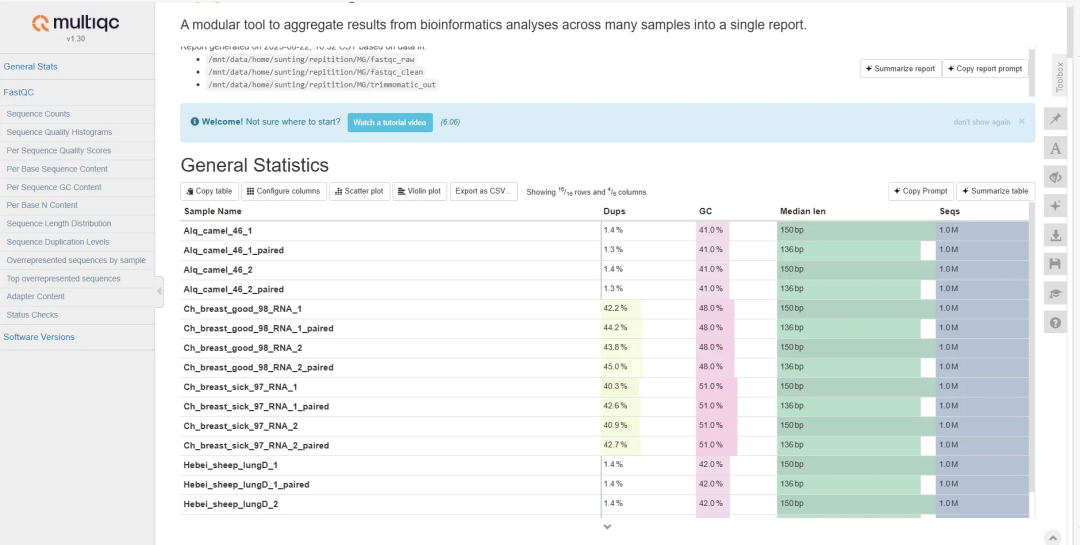
- Seqs:每个样本约 1.0 M reads。
- Dups:重复 reads 占比 1.4 %–51 %,差异极大,Ch_breast_sick_97_* 和 Hebei_sheep_lungD_* RNA测序的两组重复率显著偏高。
- GC:GC 含量 40 %–51 %,基本在哺乳动物 RNA-seq 正常范围,但 Ch_breast_sick 组 GC 偏高(51 %)
FastQC
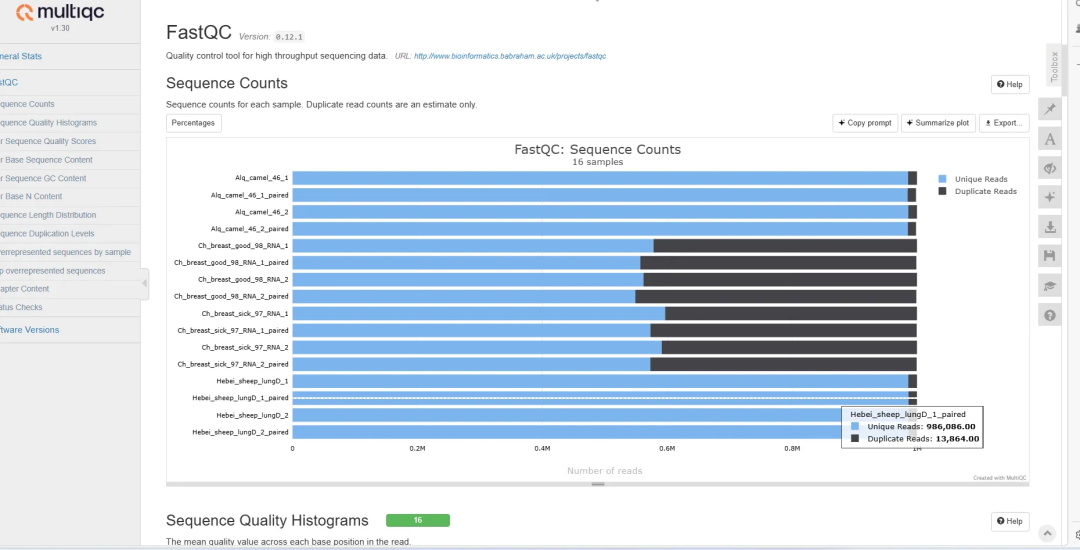
- 图中点到了 Hebei_sheep_lungD_1 给出了具体数值:Unique Reads = 986,086;Duplicate Reads = 13,864 → 重复率 ≈ 1.4 %。
- 其余样本未显示具体数字,但从柱形高度看,重复条带极窄,推断重复率同样很低(1–2 % 左右)。
- 整体重复率极低,说明文库复杂度高、PCR 扩增偏差小,建库质量良好。无样本出现高重复(>10 %),排除因 rRNA / 低复杂度序列导致的过度重复。
Sequence Quality Histograms
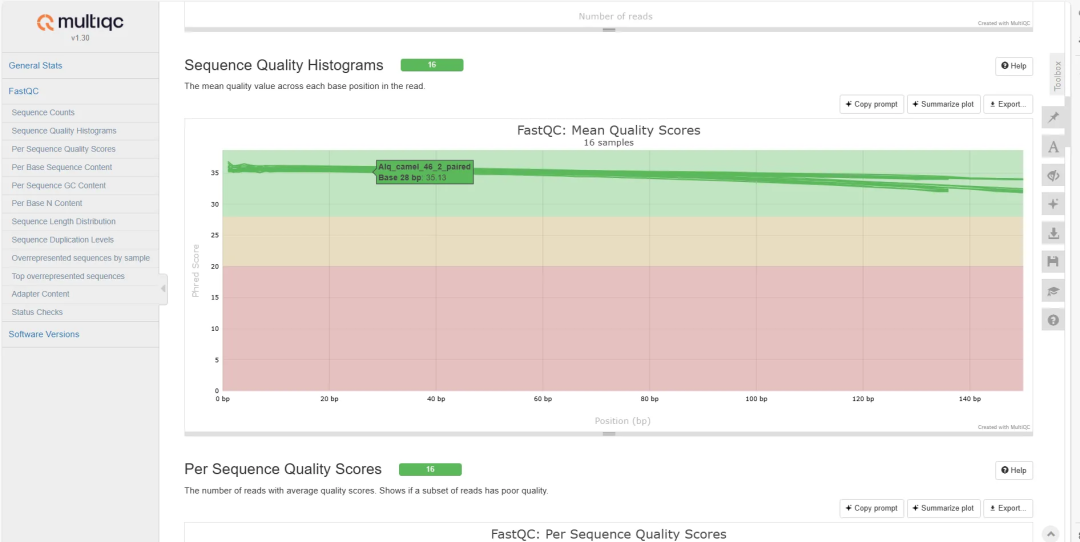
- 横轴:读长位置(0 → 140 bp)。
- 纵轴:Phred 质量分数(0 → 35)。
- 8 条曲线对应 8 个样本,颜色略有差异,但走势高度一致。
基于质控后的报告,Claude code(AI coder)帮我解读报告内容。
结果解读报告:
# 宏基因组数据质控MultiQC报告解读
## 报告概述
本报告基于MultiQC v1.30分析了4个宏基因组样本的质控结果,包括原始数据和Trimmomatic过滤后的数据,共计16个FastQC报告的综合分析。
## 样本基本统计信息
### 1. 序列数量和数据量
| 样本 | 原始序列数 | 过滤后序列数 | 保留率 | 原始数据量 | 过滤后数据量 |
|------|------------|--------------|--------|------------|--------------|
| Alq_camel_46 |1,000,000| 998,278 |99.83% | 148.2 Mbp |135.2 Mbp |
| Ch_breast_good_98_RNA | 1,000,000 |998,795| 99.88% |143.9 Mbp | 130.9 Mbp |
| Ch_breast_sick_97_RNA |1,000,000| 999,299 |99.93% | 140.2 Mbp |127.1 Mbp |
| Hebei_sheep_lungD | 1,000,000 |999,950| 100.00% |149.6 Mbp | 136.6 Mbp |
**结果解读**:
- 所有样本的数据保留率都在99.8%以上,说明原始数据质量较好
- Hebei_sheep_lungD样本质量最佳,几乎无损失
- Trimmomatic有效去除了低质量序列,平均每个样本损失约13-16 Mbp数据量
### 2. 序列长度分布
| 样本 | 原始平均长度 | 过滤后平均长度 | 原始中位数长度 | 过滤后中位数长度 |
|------|--------------|----------------|-----------------|------------------|
| Alq_camel_46 |148.7 bp | 135.0 bp |150 bp | 136 bp |
| Ch_breast_good_98_RNA |144.4 bp | 130.6 bp |150 bp | 136 bp |
| Ch_breast_sick_97_RNA |140.6 bp | 126.9 bp |150 bp | 136 bp |
| Hebei_sheep_lungD |150.1 bp | 136.2 bp |150 bp | 136 bp |
**结果解读**:
- HEADCROP:13参数有效去除了reads开头的13个碱基
- 过滤后序列长度分布更加集中,质量更加均一
- Ch_breast_sick_97_RNA原始数据平均长度较短,可能存在质量问题
### 3. GC含量分析
| 样本类型 | Alq_camel_46 | Ch_breast_good_98_RNA | Ch_breast_sick_97_RNA | Hebei_sheep_lungD |
|----------|--------------|----------------------|---------------------|------------------|
| GC含量 |41% | 48% |51% | 42% |
**结果解读**:
- 不同样本的GC含量存在显著差异,反映了物种和组织类型的不同
- Ch_breast_sick_97_RNA的GC含量最高(51%),可能与病理状态相关
- 动物样本(骆驼、羊)的GC含量相对较低且相近(41-42%)
### 4. 序列重复率分析
| 样本 | 原始重复率 | 过滤后重复率 | 独特序列数(过滤后) | 重复序列数(过滤后) |
|------|------------|--------------|-------------------|-------------------|
| Alq_camel_46 |1.44% | 1.33% |984,835| 13,443 |
| Ch_breast_good_98_RNA |42.20% | 44.25% |556,869| 441,926 |
| Ch_breast_sick_97_RNA |40.34% | 42.65% |573,102| 426,197 |
| Hebei_sheep_lungD |1.42% | 1.37% |986,086 | 13,864 |
**结果解读**:
- **高度关注**:乳腺组织样本的重复率异常高(>40%),可能原因:
- PCR扩增偏向性导致的技术重复
- 组织中某些基因的高表达导致的生物学重复
- 样本制备过程中的污染或降解
- 动物样本(骆驼、羊)重复率正常(<2%),数据质量良好
- 过滤后重复率略有上升,这是正常现象(去除低质量序列后相对比例变化)
## 质控模块状态评估
### FastQC各模块通过情况
| 质控模块 | Alq_camel_46 | Ch_breast_good_98_RNA | Ch_breast_sick_97_RNA | Hebei_sheep_lungD |
|----------|--------------|----------------------|---------------------|------------------|
| 基础统计 | ✅ | ✅ | ✅ | ✅ |
| 碱基质量 | ✅ | ✅ | ✅ | ✅ |
| 序列质量 | ✅ | ✅ | ✅ | ✅ |
| 碱基组成 | ⚠️→✅* | ❌→✅* | ❌→✅* | ⚠️→✅* |
| GC含量 | ✅ | ⚠️ | ⚠️ | ⚠️ |
| N含量 | ✅ | ✅ | ✅ | ✅ |
| 长度分布 | ⚠️ | ⚠️ | ⚠️ | ⚠️ |
| 序列重复 | ✅ | ⚠️ | ⚠️ | ✅ |
| 过表达序列 | ✅ | ⚠️ | ⚠️ | ✅ |
| 接头污染 | ✅ | ✅ | ✅ | ✅ |
*✅=通过, ⚠️=警告, ❌=失败, →表示过滤前后的变化
**关键发现**:
1. **碱基组成改善**:所有样本经过Trimmomatic处理后,碱基组成模块从警告/失败变为通过
2. **乳腺样本质控问题**:Ch_breast样本在多个模块存在警告,需要特别关注
3. **长度分布警告**:所有样本都有长度分布警告,这是HEADCROP处理的正常结果
## 序列质量趋势分析
### 碱基质量得分变化(Phred Score)
- **原始数据**:所有样本在读段末端(140-150bp)质量略有下降,符合Illumina测序特征
- **过滤后数据**:质量分布更加均匀,末端质量显著改善
- **质量阈值**:所有位置的平均质量分数都>30,达到高质量标准
### 按样本详细分析
#### 1. Alq_camel_46(骆驼样本)
- **优点**:数据质量最稳定,重复率最低,各项指标均良好
- **特征**:GC含量41%,符合哺乳动物基因组特征
- **建议**:数据质量优秀,可直接用于下游分析
#### 2. Ch_breast_good_98_RNA(健康乳腺)
- **关注点**:重复率高达44%,可能影响下游分析
- **特征**:GC含量48%,平均长度相对较短
- **建议**:需要进一步去重处理,考虑生物学重复的影响
#### 3. Ch_breast_sick_97_RNA(病变乳腺)
- **关注点**:重复率42%,GC含量最高(51%)
- **特征**:可能存在病理相关的序列偏向
- **建议**:与健康样本对比分析时需考虑技术差异
#### 4. Hebei_sheep_lungD(河北羊肺部)
- **优点**:数据保留率100%,重复率低,质量优秀
- **特征**:序列长度最长,质量最稳定
- **建议**:理想的高质量样本,适合作为参考
## 质控效果评估
### Trimmomatic处理效果
1. **有效去除低质量序列**:平均保留率99.88%
2. **改善碱基组成偏向**:所有样本碱基组成模块通过
3. **统一序列长度**:去除开头13bp后长度分布更加一致
4. **保持数据完整性**:关键信息损失最小
### 数据质量等级评定
- **优秀**:Alq_camel_46, Hebei_sheep_lungD
- **良好**:Ch_breast_good_98_RNA, Ch_breast_sick_97_RNA(需注意重复率)
## 下游分析建议
### 1. 立即可用数据
- Alq_camel_46和Hebei_sheep_lungD样本可直接进行宏基因组组装和分析
### 2. 需要额外处理的数据
- Ch_breast样本建议:
- 使用更严格的去重参数
- 评估PCR重复的影响
- 考虑使用UMI(如果有)进行去重
### 3. 比较分析注意事项
- 不同样本间的GC含量差异可能影响比较分析
- 建议在分析中考虑批次效应和技术变异
### 4. 质控监控建议
- 建立质控指标阈值:重复率<10%,保留率>95%
- 定期监控测序质量趋势
- 建立样本质控标准操作流程
## 结论
本次质控分析显示:
1. **整体数据质量良好**,Trimmomatic处理有效
2. **动物样本质量优于组织样本**,适合进行深度分析
3. **乳腺样本存在技术问题**,需要额外质控步骤
4. **所有样本均可用于下游分析**,但需要针对性的预处理策略
建议根据样本特点制定差异化的分析流程,确保结果的可靠性本文参与 腾讯云自媒体同步曝光计划,分享自微信公众号。
原始发表:2025-12-24,如有侵权请联系 cloudcommunity@tencent.com 删除
评论
登录后参与评论
推荐阅读
目录

