代码分享 | 空间数据(Xenium, CosMx, Visium HD, STO)识别肿瘤交界区扩展--免疫浸润带
代码分享 | 空间数据(Xenium, CosMx, Visium HD, STO)识别肿瘤交界区扩展--免疫浸润带

生信大杂烩
发布于 2025-12-29 11:43:00
发布于 2025-12-29 11:43:00
前言
在空间转录组数据分析中,我们经常需要研究肿瘤细胞(Tumor)与正常组织(Normal)的交界区域。这个区域被称为浸润边缘(Invasive Margin),一般这个肿瘤边缘交界区可以使用10x的Xenium Browser(如果是VisiumHD细胞分割数据,则可以使用Loupe Browser)根据HE染色结果,手动圈选,但是细胞混杂,如何在密密麻麻的细胞中,精准地把那些在交界处的细胞挑出来比较考验耐心,就想着能不能基于细胞自动识别出肿瘤交界区,拟合一条边界线出来,便于后续研究,比如想看某些基因是否随着与交界处距离的变化它的表达也会发生变化等。
之前我们也有介绍过一篇23年的空间文章,研究的一个核心概念是肿瘤边缘的"侵袭区",文章中定义的是以肿瘤边缘为中心的500微米宽的区域,这里是肿瘤细胞侵袭和转移的活跃前沿,包含复杂的细胞成分及独特的分子特征,存在免疫抑制微环境、肿瘤细胞代谢重编程及受损肝细胞,对肿瘤进展和患者预后有关键影响。这个区域在肿瘤侵袭和转移中具有重要地位,文章后续都是围绕其位置、细胞组成、分子特征以及功能作用进行详细描述。
Cell Res | Stereo-seq揭示人类肝癌浸润区促进肝细胞-肿瘤细胞串扰、局部免疫抑制和肿瘤进展
主要就是找到肿瘤边缘,向肿瘤内部和外部扩展一定宽度,定义侵袭区
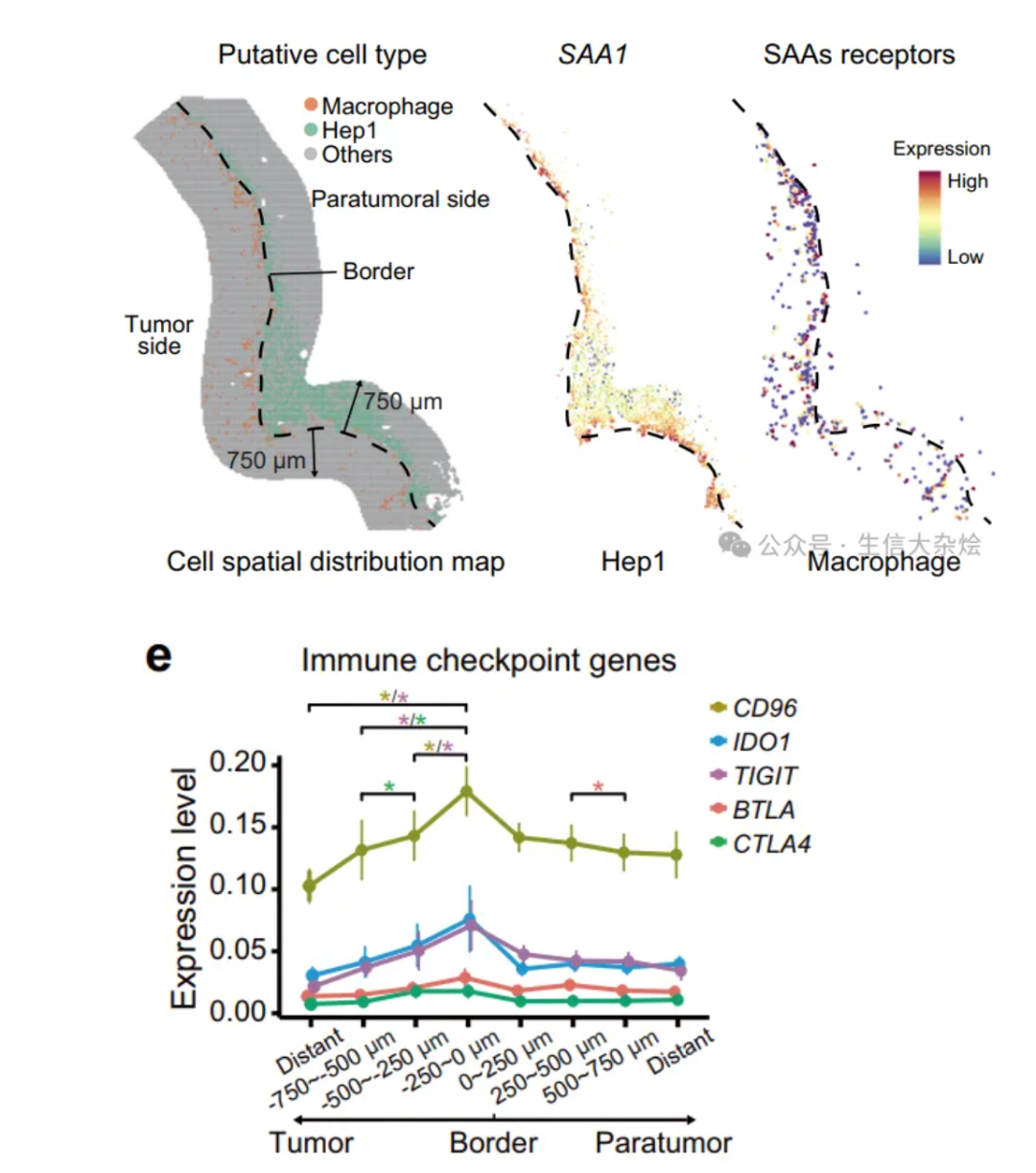
以下是这篇文章中关于肿瘤边缘的"侵袭区"的实现描述

文章方法描述中用到的是图像识别,这里我们介绍基于细胞空间位置实现的方法
第一部分:识别边界细胞
1. 原理
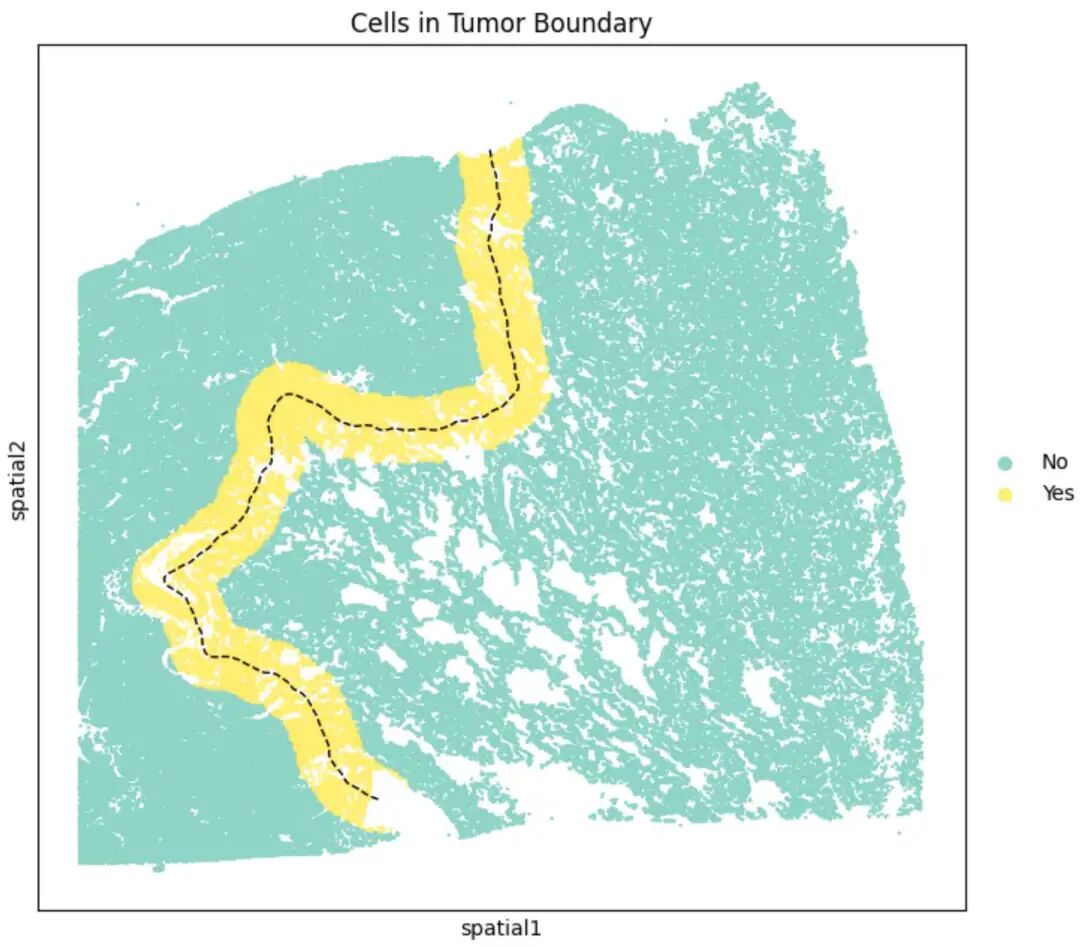
利用K-近邻算法 (KNN) 的思想。如果一个细胞是“肿瘤细胞”,但是离它最近的N个邻居细胞中里出现了“正常”细胞(或者反过来),那么我们就可以确认这个细胞就处于交界区,我们可以给它打个boundary cell的标签。
如图所示,首先定义好肿瘤核心区域和正常区域
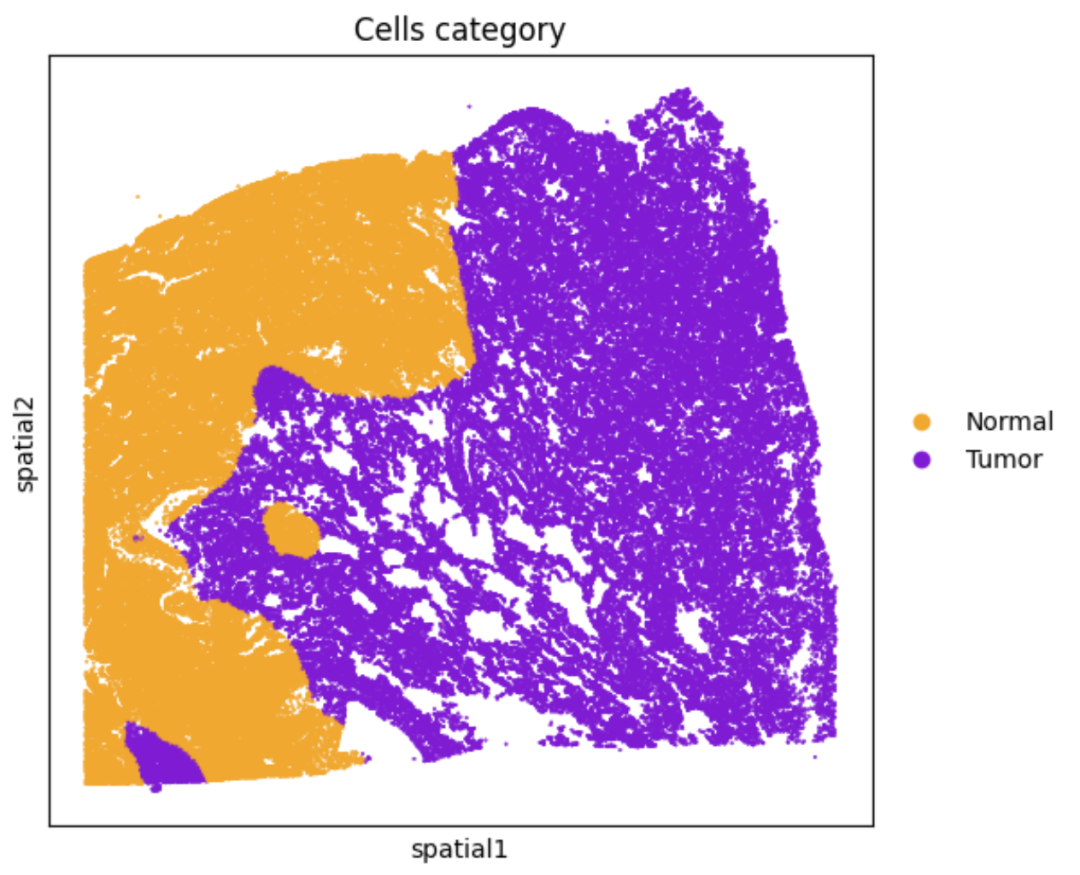
2. 实现逻辑
- 获取坐标与标签:读取
adata.obsm['spatial'](空间坐标) 和adata.obs['CN_category'](细胞类型)。 - 构建邻居图:使用
NearestNeighbors对所有细胞的坐标构建索引,找出每个细胞最近的n_neighbors个邻居。 - 遍历检查:
- 遍历每一个细胞。
- 如果当前细胞是
Tumor,检查它的邻居里有没有Normal。 - 如果当前细胞是
Normal,检查它的邻居里有没有Tumor。 - 只要满足上述条件,就将该细胞标记为
True(是边界细胞)。
- 保存结果:将布尔值保存在
adata.obs['is_boundary']中。
3. 关键参数
- **
n_neighbors=10**:- 含义:判断边界时考虑周围多少个细胞。
- 影响:数值越小,边界越细碎、对噪声越敏感;数值越大,识别出的边界带越宽(因为隔得较远的细胞也会被算作邻居)。
这里黄色的细胞就是我们找到的处于边界上的细胞
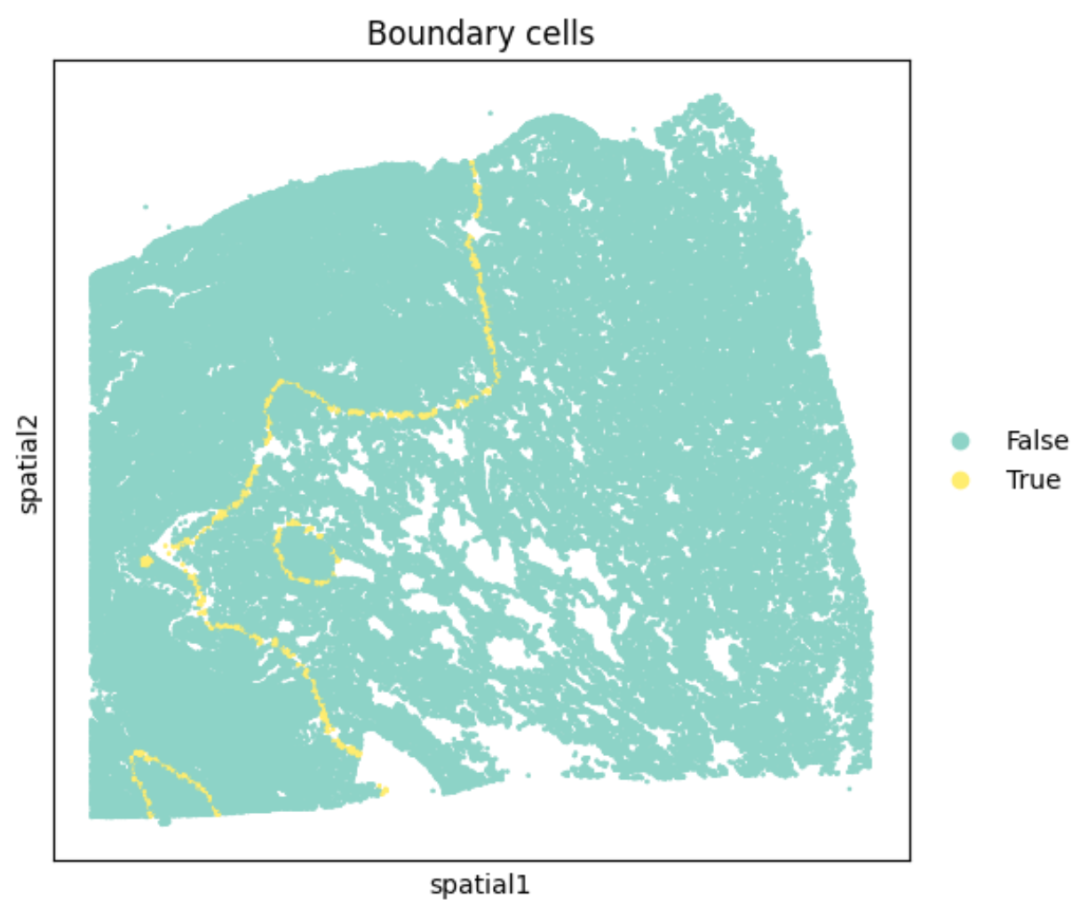
第二部分:拟合平滑边界线 (plot_smooth_boundary_masked)
这一步比较复杂,它不是简单地把黄点连起来(那样会很乱),而是采用“分类概率等高线”的方法。
1. 原理
将空间看作一个平面,训练一个分类器来预测平面上任意一点是 Tumor 的概率。在 Tumor 概率为 0.5 的地方,就是理论上的分界线。为了好看,增加了平滑处理;为了真实,增加了空白区域屏蔽。
2. 实现逻辑
- 准备数据:提取坐标 和类别 (Tumor=1, Normal=0)。
- 绘制散点:先画出背景的 Tumor/Normal 细胞,再把第一步识别出的
is_boundary细胞用红色高亮画出来。 - **训练分类器 (KNN Classifier)**:
- 用
KNeighborsClassifier训练。 - 作用:给它平面上任意一个坐标,它能根据周围的细胞投票,算出这个坐标属于 Tumor 的概率(0到1之间)。
- 用
- **创建网格 (Meshgrid)**:在整个坐标范围内生成密集的网格点(比如 500x500 个点),覆盖整个组织区域。
- 计算距离掩膜 (Distance Masking) —— 这里是处理“空白处不画线”的关键:
- 计算网格上每个点距离最近的真实细胞有多远。
- 如果网格点在组织外的空白区域,离真实细胞很远,这个距离就会很大。
- 预测与平滑:
- 预测:计算网格上每个点的 Tumor 概率。此时得到的概率图是阶梯状的(比较锯齿)。
- **高斯模糊 (
gaussian_filter)**:对概率图进行模糊处理。这会让 0 和 1 之间的过渡变得平滑,从而让画出来的线是圆润的曲线,而不是锯齿线。
- 应用掩膜:
- 利用第5步计算的距离。如果某网格点距离真实细胞超过
max_dist_threshold,将其概率设为NaN。 - 这样在画线时,这些区域就会被跳过(断开)。
- 利用第5步计算的距离。如果某网格点距离真实细胞超过
- **提取轮廓 (Contour)**:
- 使用
ax.contour寻找概率为 0.5 的等高线。这就是 Tumor 和 Normal 分庭抗礼的分界线。 - 筛选掉太短的线段(
min_vertex_count),只保留主边界。
- 使用
3. 关键参数详解
- **
smooth_sigma=2(或6)**:- 越大:边界线越平滑、越圆润,但可能会丢失细节,导致线与真实细胞点的贴合度下降(过于“概括”)。
- 越小:边界线越紧贴细胞分布,但可能会出现锯齿或不必要的抖动。
- 含义:高斯模糊的核大小。
- 影响:
- **
min_vertex_count=300**:- 含义:最小顶点数。也就是画出来的线如果不满足包含300个点,就丢弃。
- 作用:去噪。通常我们只想要主边界线。组织内部偶尔会有几个杂散的细胞导致分类器在内部画出小圆圈,这个参数可以把这些孤立的小圈圈过滤掉。
- **
max_dist_threshold=300**:- KNN 分类器的特性是,即使在离组织很远的空白处,它也会根据遥远的细胞强行预测一个类别,导致边界线延伸到画布边缘。
- **如果改小 (e.g., 100)**:网格点必须离细胞很近才保留。如果组织中间有较大的裂隙或空洞,边界线可能会断开。
- **如果改大 (e.g., 500)**:允许网格点离细胞较远。能把断裂的边界连起来,但可能会在明明没有细胞的背景区域画出多余的线。
- 含义:最大距离阈值。
- 作用:控制边界线的延伸范围(防止“脑补”过度)。
- 调节指南:
黑色线即为你拟合出来的肿瘤细胞边界
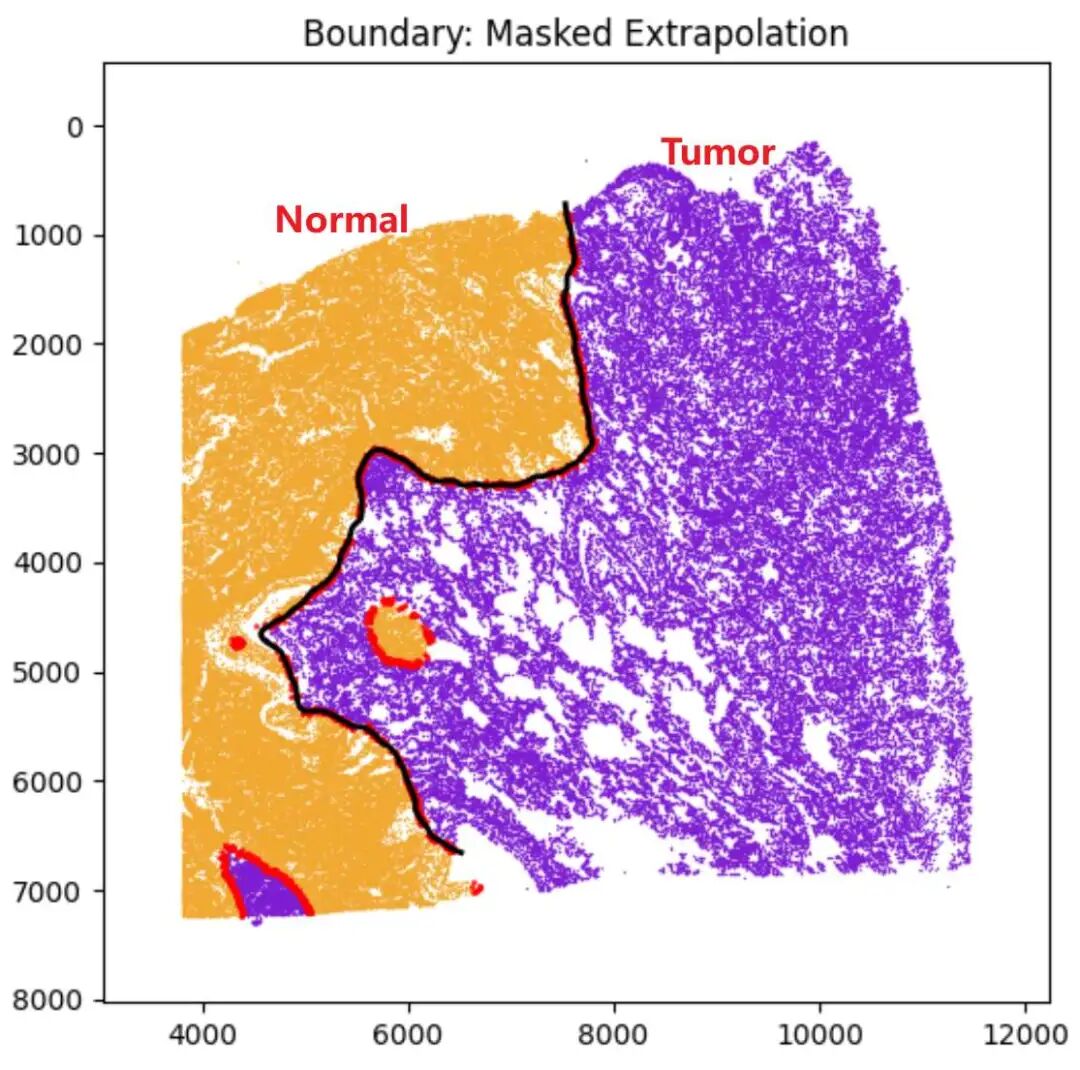
第三部分:计算所有细胞到边界线的物理距离
既然已经拟合出了边界线,那么我们就可以计算每个细胞到这条线的最近距离,这个是最关键的,有了每个细胞到边界线的距离后,我们就很容易能够去自定义划定这个浸润带,比如上述说的定义一条50um宽的浸润带,那么我们只要去看,哪些细胞跟边界线的距离小于25um,这些细胞所在的区域就组成了这条50um宽的浸润带。接下来就可以去看这个区域中细胞的组成、与其他区域基因表达差异、功能差异、细胞间通讯等。
1. 以肿瘤边界线为中心,扩展50um画出一条带浸润带
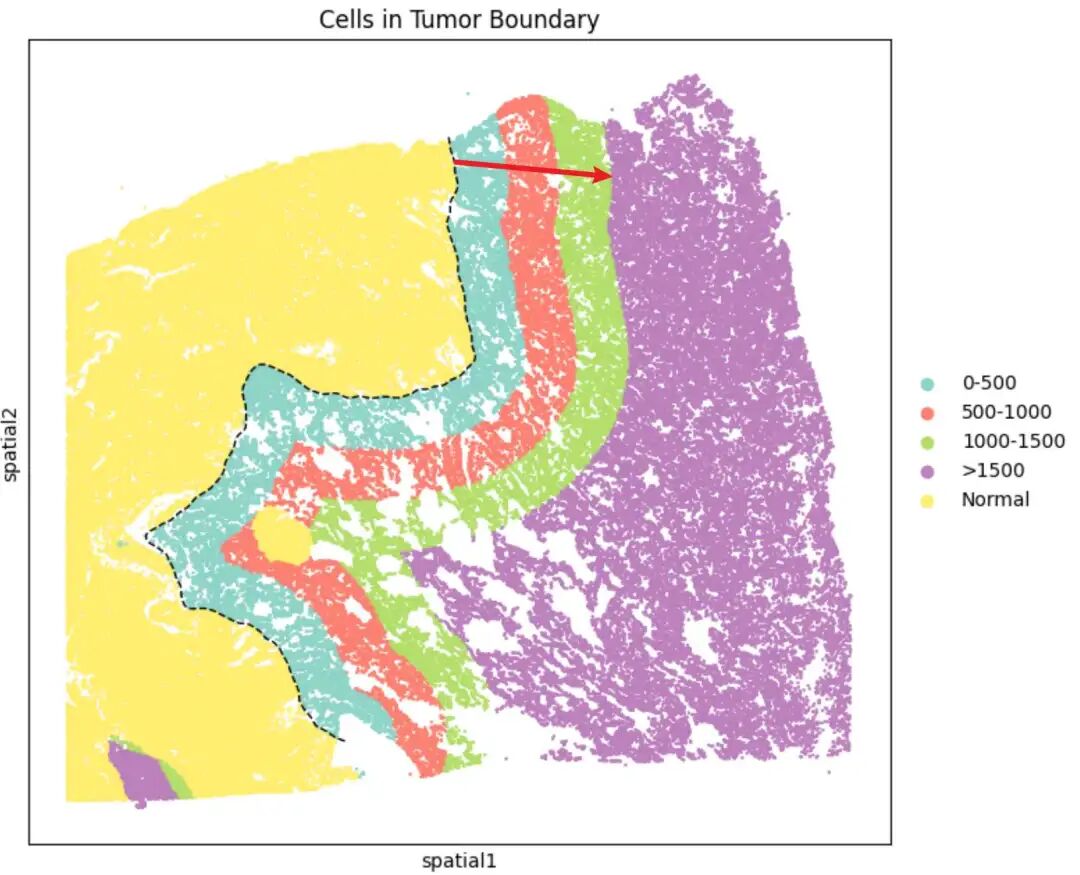
2. 以肿瘤边界线为中心,按一定梯度向内或向外扩展出多条带浸润带
上面一步我们计算出了所有细胞到边界线的距离后,只需要根据一定的梯度距离,就可以将所有的细胞分到不同的浸润带内,只要给同一浸润带内的细胞打上标签,这样的话就能够画出多个连续的浸润带。
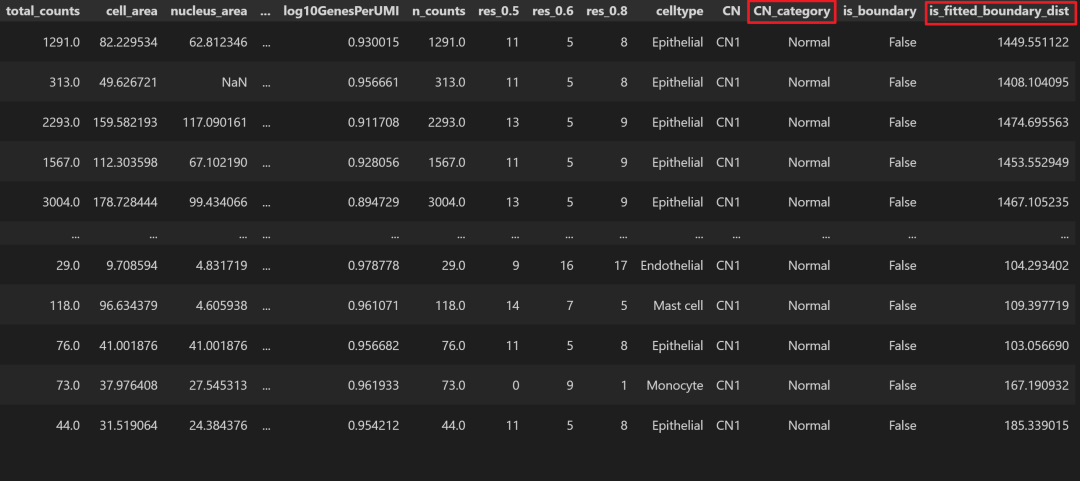
3. 根据边界线,指定向肿瘤核心的距离为正值,向边界外的距离值为负值
这个需求其实是可以投机取巧的,首先,我们还是第一步一样,计算每个细胞到边界线的距离,然后我们也知道每个细胞的类别,到底是肿瘤细胞还是正常细胞,同时也知道哪些细胞是在边界上的细胞,这样的话我们只要将除了边界上的细胞外,将正常细胞到边界线的距离值加上负号即可。
完整代码实现
定义calculate_boundary函数实现
import os
import numpy as np
import pandas as pd
import scanpy as sc
import squidpy as sq
import seaborn as sns
import matplotlib.pyplot as plt
from scipy.spatial import cKDTree
from gene_set_enrichment import gsea
from scipy.ndimage import gaussian_filter
from sklearn.neighbors import NearestNeighbors, KNeighborsClassifier
import matplotlib.colors as mcolors
def calculate_boundary(adata, obs_col, cat_a, cat_b, n_neighbors, cat_focus, smooth_sigma=2, min_vertex_count=200, max_dist_threshold=300):
# 1. 获取坐标和标签
coords = adata.obsm['spatial']
labels = adata.obs[obs_col].values
# 2. 构建空间邻居图 (KNN)
nbrs = NearestNeighbors(n_neighbors=n_neighbors).fit(coords)
distances, indices = nbrs.kneighbors(coords)
boundary_mask = np.zeros(len(adata), dtype=bool)
# 3. 遍历每个细胞,检查其邻居
for i in range(len(adata)):
current_label = labels[i]
# 只关心 Tumor 或 Normal 细胞
if current_label not in [cat_a, cat_b]:
continue
# 获取邻居的标签
neighbor_indices = indices[i]
neighbor_labels = labels[neighbor_indices]
# 检查邻居中是否存在"对立"的类别
if current_label == cat_a and cat_b in neighbor_labels:
boundary_mask[i] = True
elif current_label == cat_b and cat_a in neighbor_labels:
boundary_mask[i] = True
# 将结果存回 adata
adata.obs['is_boundary'] = boundary_mask
print(f"识别出 {sum(boundary_mask)} 个边界细胞")
# 4. 训练KNN分类器
X = adata.obsm['spatial']
y = (adata.obs[obs_col] == cat_focus).astype(int).values
clf = KNeighborsClassifier(n_neighbors=n_neighbors)
clf.fit(X, y)
# 5. 创建网格
padding_percent = 0.1
x_min, x_max = X[:, 0].min(), X[:, 0].max()
y_min, y_max = X[:, 1].min(), X[:, 1].max()
x_pad = (x_max - x_min) * padding_percent
y_pad = (y_max - y_min) * padding_percent
grid_resolution = 500
xx, yy = np.meshgrid(np.linspace(x_min - x_pad, x_max + x_pad, grid_resolution),
np.linspace(y_min - y_pad, y_max + y_pad, grid_resolution))
grid_points = np.c_[xx.ravel(), yy.ravel()]
# 计算网格上每个点,距离最近的真实细胞有多远
nbrs_geometry = NearestNeighbors(n_neighbors=1).fit(X)
dists, _ = nbrs_geometry.kneighbors(grid_points)
dists = dists.reshape(xx.shape)
# 6. 预测概率 + 高斯模糊
Z = clf.predict_proba(grid_points)[:, 1]
Z = Z.reshape(xx.shape)
Z_smooth = gaussian_filter(Z, sigma=smooth_sigma)
# 如果网格点距离真实细胞太远,将其概率设为 NaN,这样 contour 就不会在那里画线了
Z_smooth[dists > max_dist_threshold] = np.nan
# 绘制背景散点
dot_size = 1
fig, ax = plt.subplots(figsize=(6, 6))
is_boundary = adata.obs['is_boundary'].astype(bool).values
mask_other_normal = (y == 0) & (~is_boundary)
ax.scatter(X[mask_other_normal, 0], X[mask_other_normal, 1], c='#F0A830', s=dot_size, alpha=0.8, linewidth=0)
mask_core_normal = (y == 1) & (~is_boundary)
ax.scatter(X[mask_core_normal, 0], X[mask_core_normal, 1], c='#7F1CD3', s=dot_size, alpha=0.8, linewidth=0)
mask_boundary = is_boundary
ax.scatter(X[mask_boundary, 0], X[mask_boundary, 1], c='red', s=dot_size, label='Identified Boundary Cells', alpha=0.8)
# 7. 提取轮廓, 这里的 contour 会自动处理 NaN 值(断开线条)
contours = ax.contour(xx, yy, Z_smooth, levels=[0.5], alpha=0)
# 8. 筛选并画线, 用于收集所有有效边界线上的坐标点
valid_boundary_coords = []
if len(contours.allsegs) > 0:
segments = contours.allsegs[0]
for coords in segments:
if len(coords) > min_vertex_count:
ax.plot(coords[:, 0], coords[:, 1], color='black', linewidth=2, linestyle='-', label='Fitted Curve')
valid_boundary_coords.append(coords)
ax.set_title(f"Boundary: Masked Extrapolation")
# 将拟合的线条坐标存入adata.uns, valid_boundary_coords 是一个列表,里面可能有几段线(如果是断开的)
adata.uns['fitted_boundary_coords'] = valid_boundary_coords
# Y轴坐标原点调整到左上角
ylim = ax.get_ylim()
if ylim[0] < ylim[1]:
ax.invert_yaxis()
# 计算每个细胞到拟合边界的距离并存入 adata.obs
col_name = 'is_fitted_boundary_dist'
if len(valid_boundary_coords) > 0:
# 1. 将所有有效线段的点拼接成一个大的坐标数组 (N_points, 2)
all_boundary_points = np.vstack(valid_boundary_coords)
# 2. 构建 KDTree 以便快速查找
tree = cKDTree(all_boundary_points)
# 3. 查询每个细胞(X)到最近边界点的距离
# k=1 表示找这1个最近的邻居
dist_values, _ = tree.query(X, k=1)
# 4. 存入 adata
adata.obs[col_name] = dist_values
print(f"Success: 已计算每个细胞到拟合线的距离,并存入 adata.obs['{col_name}']")
print(f" - 最小距离: {dist_values.min():.2f}")
print(f" - 最大距离: {dist_values.max():.2f}")
else:
# 如果没有找到符合条件的边界线,填充 NaN
print("Warning: 未检测到符合条件的边界线,距离列填充为 NaN。")
adata.obs[col_name] = np.nan
return adata
本文参与 腾讯云自媒体同步曝光计划,分享自微信公众号。
原始发表:2025-12-17,如有侵权请联系 cloudcommunity@tencent.com 删除
评论
登录后参与评论
推荐阅读
目录

