给Seurat默认的气泡图开个美颜!
画图课程如火如荼的内测ing~~~大家的学习热情高涨,我们也在随时收集大家喜欢的图。今天的图是直播课学生许愿的:
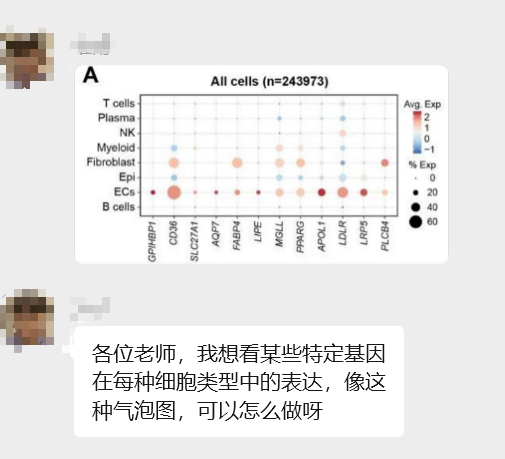
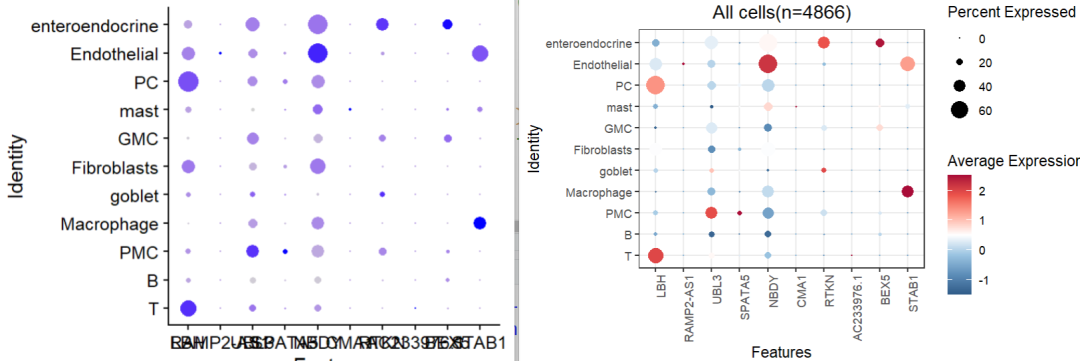
在单细胞数据分析的江湖里,Seurat无疑是绝对的王者。但是呢,标准流程只是起点,美丽的图表任重道远~默认的dotplot图长这样:
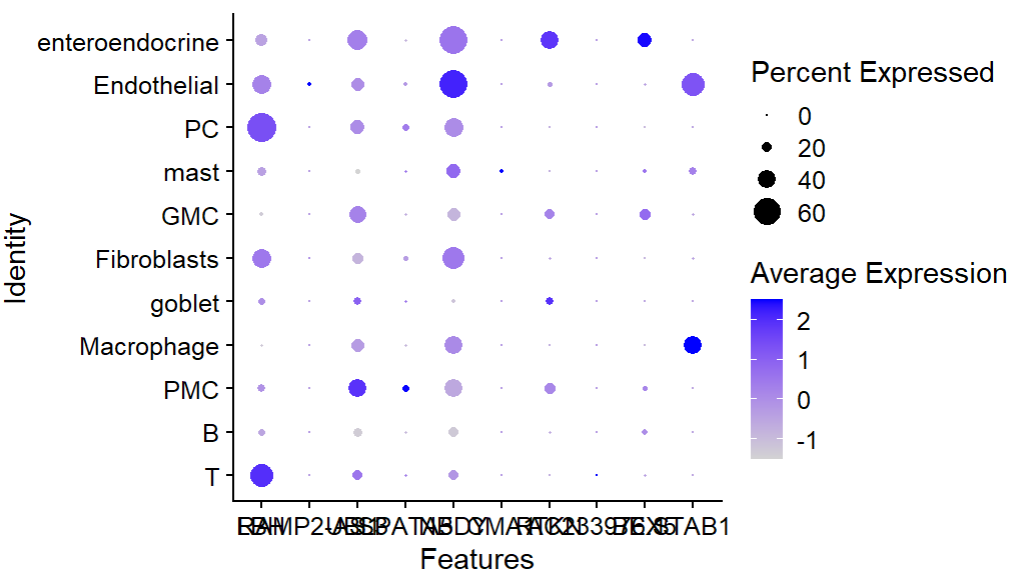
默认的蓝紫色渐变、灰色的背景、还有那挤作一团的X轴标签,太基础啦。ps:加一个函数RotatedAxis()可以旋转45°,避免重叠。
在这张图的基础上,基于ggplot2和paletteer做一些个性化调整,给他变个身!
先上我们调试好的代码和效果预览:
rm(list = ls())
library(Seurat)
library(ggplot2)
library(paletteer)
# 加载已经注释好的Seurat对象
load("seu.obj.Rdata")
# 查看细胞类型,如果没有注释这里会显示样本名称或者是0、1、2...这样的编号
levels(seu.obj)
# 设置随机种子以保证结果可复现
set.seed(12281)
# 从Seurat对象中随机抽取10个基因用于演示
genes = rownames(seu.obj)[sample(seq_len(nrow(seu.obj)),10)]
# 画图
DotPlot(seu.obj, features = genes) +
theme_bw() +
theme(
axis.text.x = element_text(angle = 90, hjust = 1), # X轴标签旋转90度并对齐
plot.title = element_text(hjust = 0.5) # 标题居中
) +
scale_color_paletteer_c(
"ggthemes::Red-Blue-White Diverging", # paletteer包中的配色
direction = -1# 反转颜色方向
) +
ggtitle(paste0("All cells(n=", ncol(seu.obj), ")")) # 居中标题
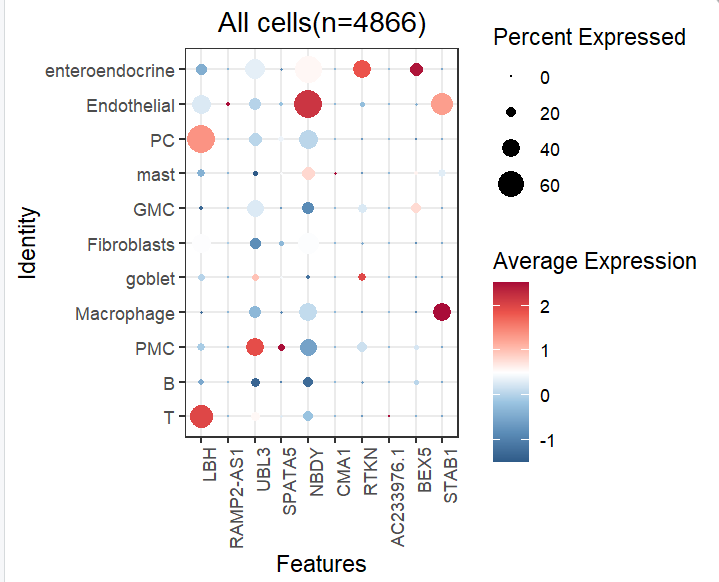
因为对ggplot2比较熟悉,这些代码是我手搓的。不过时代在进步,用ai写代码的能力也在突飞猛进啊。我们的画图课程也渗透了AI的戏份,降低些许学习成本。
本文参与 腾讯云自媒体同步曝光计划,分享自微信公众号。
原始发表:2026-01-31,如有侵权请联系 cloudcommunity@tencent.com 删除
评论
登录后参与评论
推荐阅读


