Python字符串拼接的十种方式
Python字符串拼接的十种方式
参考链接: 如何检查字符串在Python中是否为有效关键字?
Python字符串拼接的十种方式
字符串是所有编程语言中都有的基本变量的类型,程序员基本每天都在和字符串打交道。
本篇文章我们一起细数Python中的各种字符串拼接方法。每种字符串拼接方式的使用场景各不相同,我们可以在开发过程中灵活运用。
一、用逗号,拼接
str_a = 'python'
print('hello', str_a, '!')
运行结果:
hello python !
用逗号拼接的结果中,相邻的两个字符串之间会有空格。
二、空格自动拼接
str_b = 'It is summer ' 'of 2019!'
print(str_b)
运行结果:
It is summer of 2019!
空格自动拼接后,两个字符串之间的空格不会保留,两个字符串会直接连在一起,不能通过多个变量名来拼接。
三、多行字符串自动拼接
str_c = 'Love makes ' \
'man grow up ' \
'or sink down!'
print(str_c)
运行结果:
Love makes man grow up or sink down!
多行字符串之间会有反斜杠\作为连接,自动拼接后,多行字符串会直接连在一起,中间没有空格,不能通过多个变量名来拼接。
所以我们在编程中,一行写不完的字符串可以换行,最后的结果是一样的。
四、通过加号+拼接
str_d = 'string'
str_e = 'demo'
print(str_d + str_e)
str_e += str_d
print(str_e)
运行结果:
stringdemo
demostring
加号拼接的结果也是两个字符串直接拼接在一起,中间没有空格。
五、通过乘法*进行拼接
str_f = 'a-' * 10
print(str_f)
运行结果:
a-a-a-a-a-a-a-a-a-a-
Python中一个字符串与整数相乘的结果为字符串拼接整数次。
六、字符串格式化操作符%拼接
str_g = 'aaaaaaaaaaaa%saaaaaaaaa' % 'A'
print(str_g)
str_h = 'aaaaaaaaaaaa%06daaaaaaaaa' % 10
print(str_h)
str_i = 'aaaaaaaaaaaa%.03faaaaaaaaa' % 0.77
print(str_i)
运行结果:
aaaaaaaaaaaaAaaaaaaaaa
aaaaaaaaaaaa000010aaaaaaaaa
aaaaaaaaaaaa0.770aaaaaaaaa
%s,%d,%f的作用是占位作用,然后在字符串后面跟一个%,再在后面写拼到占位位置的内容。
%s:将一个字符串拼接到前面的字符串中
%d:将一个整型数字转换成字符串拼接到前面的字符串中,可以设置整数的位数,前面补0
%f:将一个浮点型数字转换成字符串拼接到前面的字符串中,可以设置小数点后的位数,后面补0
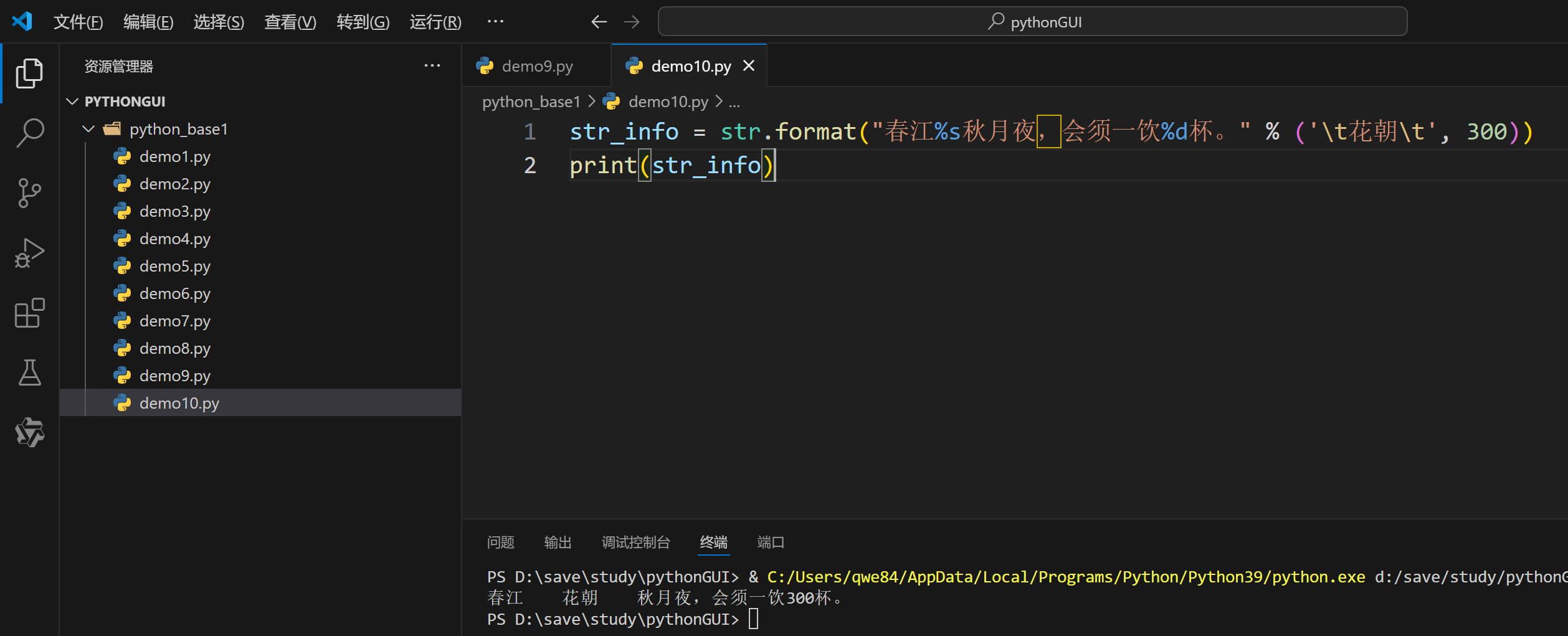
七、通过str.format()方法拼接
str_j = 'python {}! format {}!'.format(666, 999)
print(str_j)
str_k = '生如夏花之{a},死如秋叶之{b}!'.format(b='静美', a='绚烂')
print(str_k)
运行结果:
python 666! format 999!
生如夏花之绚烂,死如秋叶之静美!
format()方法可以说是最通用(基本是万能的)的拼接方法了,不管后面拼接的数据是字符串还是数字,甚至元组、列表、字典、集合等数据类型,format统统都可以拼接到字符串中。
在要拼接的字符串中使用大括号{}来给拼接内容占位,后面按顺序依次传入对应的内容即可,也可以给每个占位的{}起一个变量名,然后通过关键字参数传递给format().
八、通过str.join()方法拼接
list_l = ['生', '如', '夏', '花', '之', '绚', '烂', ',', '死', '如', '秋', '叶', '之', '静', '美', '!']
str_l = ''.join(list_l)
print(str_l)
tuple_m = ('生', '如', '夏', '花', '之', '绚', '烂', ',', '死', '如', '秋', '叶', '之', '静', '美', '!')
str_m = '-'.join(tuple_m)
print(str_m)
运行结果:
生如夏花之绚烂,死如秋叶之静美!
生-如-夏-花-之-绚-烂-,-死-如-秋-叶-之-静-美-!
str.join()最常用来将一个列表内的字符串拼接成一个大的字符串,列表中的每个元素都需要是字符串类型。
前面的str是拼接时用于连接的字符串,列表的每两个元素之间使用str来连接。
九、通过string模块中的Template对象拼接
from string import Template
t = Template('${s1} ${s2}!')
str_n = t.safe_substitute(s1='Hello', s2='Python')
print(str_n)
运行结果:
Hello Python!
Template的实现方式是首先通过Template初始化一个字符串对象t,在创建对象t时,在字符串中使用 ${变量名} 的方式来给拼接内容占位。
也可以省略{},直接 $变量名,只是没有{}时变量名后面一定要有空格,且可读性不如有{}的好。
通过调用t对象的substitute或safe_subsititute方法,将拼接内容通过关键字参数的方式依次传递进去,实现在指定的位置拼接字符串。
这种方法与上面的format()方法一样,不管传入的拼接内容是什么数据类型,统统都可以拼接。
十、通过F-strings拼接
o = 6666666666
p = 7777777777
str_o = f'Python {o} hello {p} !'
print(str_o)
运行结果:
Python 6666666666 hello 7777777777 !
在Python3.6版本中,提出了一种新型字符串格式化机制,被称为“字符串插值”或者F-strings,F-strings提供了一种明确且方便的方式将python表达式嵌入到字符串中来进行格式化。而且F-strings的运行速度很快,比%和format()这两种格式化方法都快得多。
上面的代码中直接在f后面跟字符串,然后在字符串中用{}传入拼接内容。
厉害的是,在F-strings中我们可以执行函数:
def add_string(str):
return str.upper()
str = 'a k q j 10'
str_p = f'顺子! {add_string(str)}'
print(str_p)
运行结果:
顺子! A K Q J 10
将函数传入{}中,最后拼接到字符串中的内容是函数执行的结果!
现在我们已经细数了十种Python字符串的拼接方式,希望您可以在使用中融会贯通,灵活运用。
本文系转载,前往查看
如有侵权,请联系 cloudcommunity@tencent.com 删除。
本文系转载,前往查看
如有侵权,请联系 cloudcommunity@tencent.com 删除。
腾讯云开发者

扫码关注腾讯云开发者
领取腾讯云代金券
Copyright © 2013 - 2025 Tencent Cloud. All Rights Reserved. 腾讯云 版权所有
深圳市腾讯计算机系统有限公司 ICP备案/许可证号:粤B2-20090059 深公网安备号 44030502008569
腾讯云计算(北京)有限责任公司 京ICP证150476号 | 京ICP备11018762号 | 京公网安备号11010802020287
Copyright © 2013 - 2025 Tencent Cloud.
All Rights Reserved. 腾讯云 版权所有