🤩 DGSEA | GSEA做完了不要停,再继续比较一下有意义的通路吧!~
🤩 DGSEA | GSEA做完了不要停,再继续比较一下有意义的通路吧!~
1写在前面
GSEA大家都会用了,但GSEA也有它自己的缺点,就是不能比较两个基因集或通路的富集情况。🤒
今天介绍一个Differential Gene Set Enrichment Analysis (DGSEA),可以量化两个基因集的相对富集程度。😉
2用到的包
rm(list = ls())
# devtools::install_github('JamesJoly/DGSEA')
library(DGSEA)
library(tidyverse)
library(RCurl)
library(GSA)
3示例数据
这里我们准备了一个RNA sequencing的数据,包含31个乳腺癌细胞系,分别接受1%和20%的氧供。🥰
数据有点太大了,这里我们就只选取其中2个细胞系吧(MCF10A和MCF12A)。😘
原数据来自这篇paper:👇
Ye,I.C. et al. (2018) Molecular Portrait of Hypoxia in Breast Cancer: A Prognostic Signature and Novel HIF-Regulated Genes. Mol. Cancer Res. MCR, 16, 1889–1901.
dat <- read.csv("./DGSEA_example.csv",row.names = 1)
DT::datatable(dat)
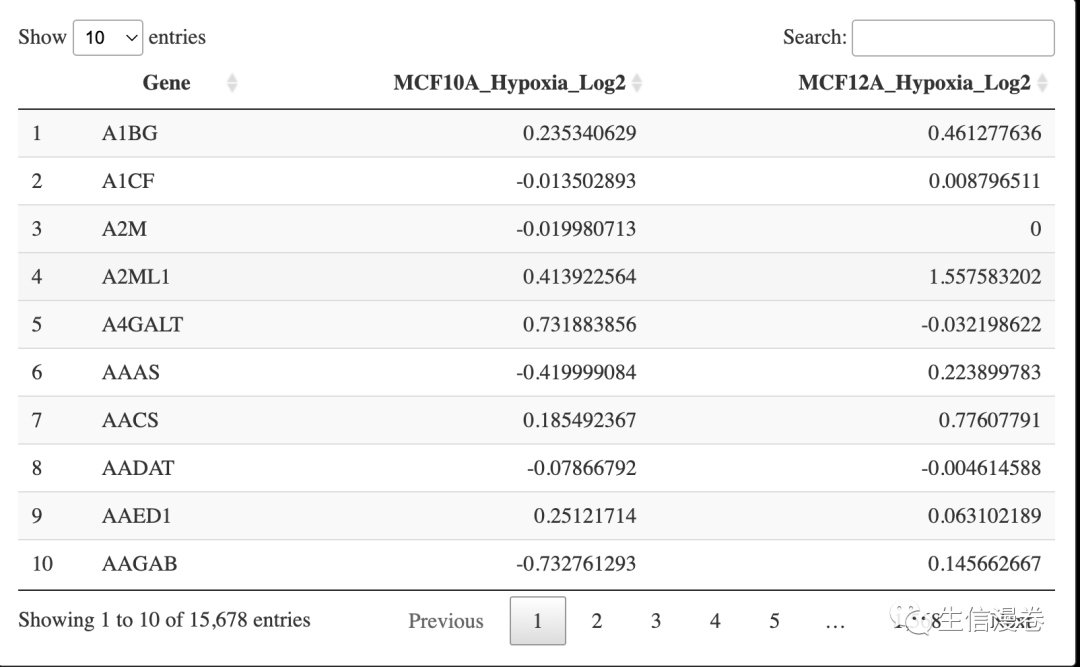
4整理数据
4.1 提取数据
MCF10A <- dat[,1:2]
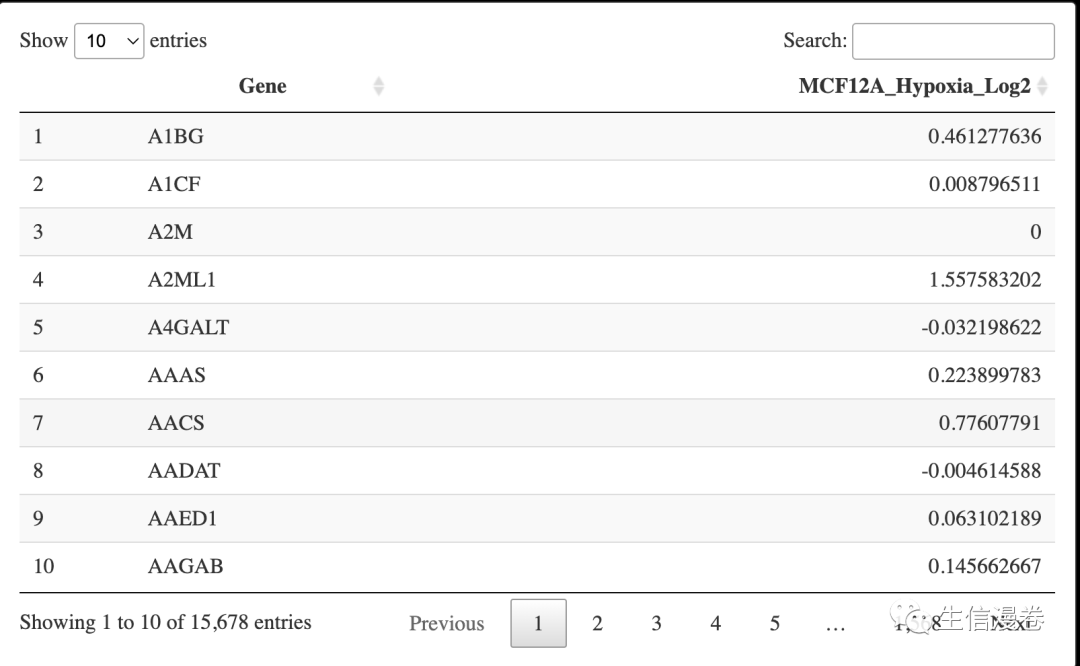
MCF12A <- dat[,-2]
4.2 MCF10A数据
DT::datatable(MCF10A)
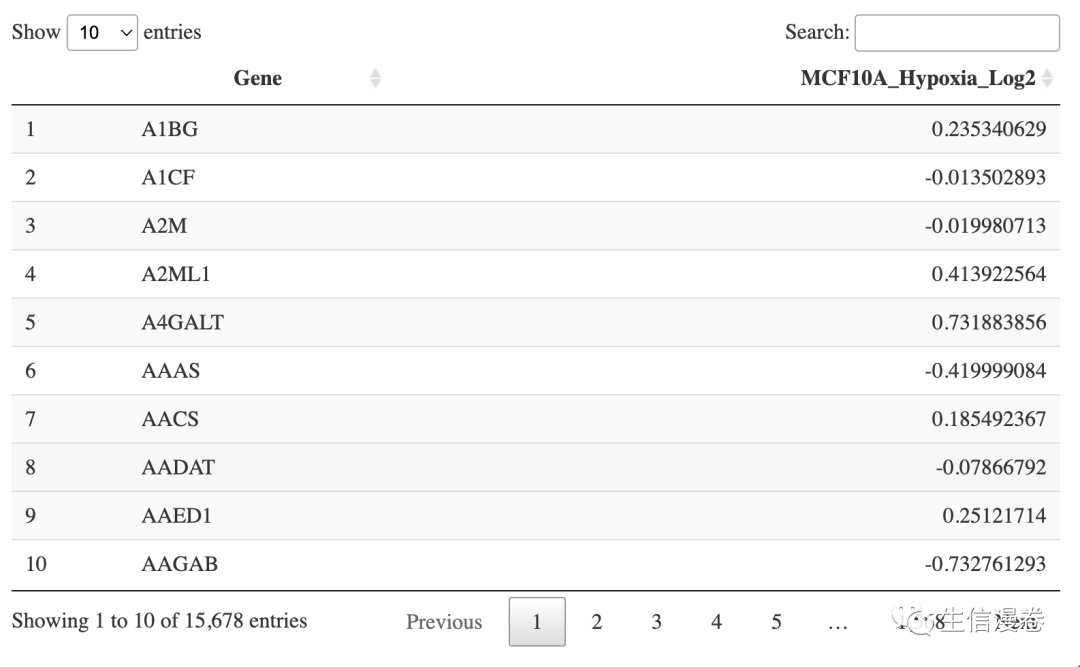
4.3 MCF12A数据
DT::datatable(MCF12A)
5读入gmt文件
这次我们比较一下KEGG的代谢通路,看一下这里的69个代谢通路吧。🤪
kegg.pathways <- GSA.read.gmt("./KEGG_metabolic_pathways.gmt")
kegg.pathways$geneset.names
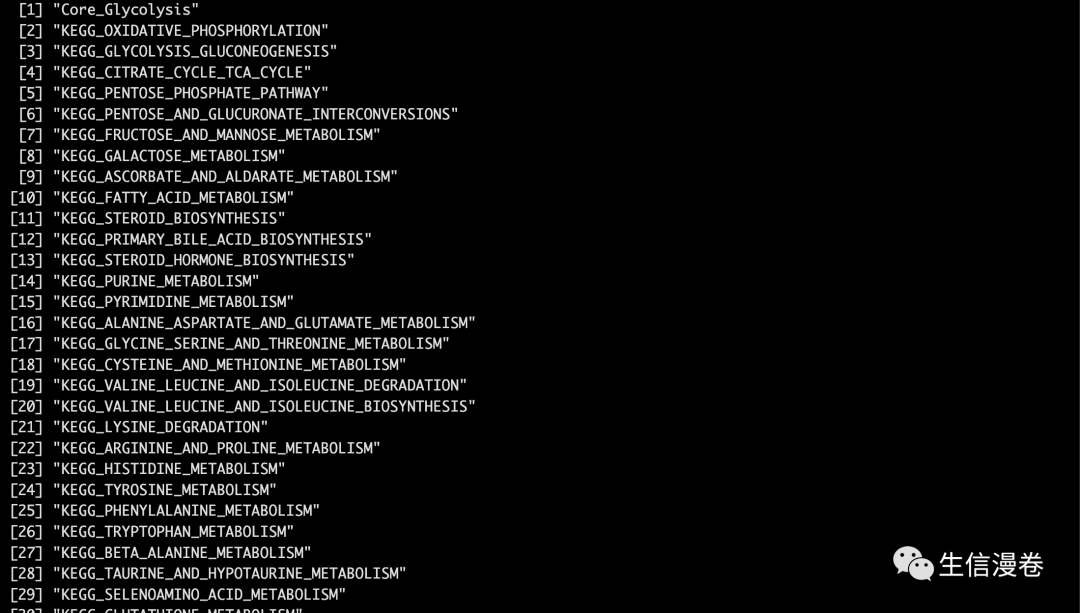
6靶向DGSEA
如果我们有非常明确的需要比较的两个通路,可以使用这种方法,进行定量分析。🤖
这里我们分别在2个不同的细胞系中比较一下这2个代谢通路吧:👇
- "Core_Glycolysis";
- "KEGG_OXIDATIVE_PHOSPHORYLATION";
6.1 开始DGSEA
1️⃣ MCF10A细胞系:👇
set.names <- kegg.pathways$geneset.names
targeted.MCF10A <- dgsea_targeted(MCF10A, kegg.pathways,
set.names[1], set.names[2])
2️⃣ MCF12A细胞系:👇
targeted.MCF12A <- dgsea_targeted(MCF12A, kegg.pathways,
set.names[1], set.names[2])
6.2 可视化
这里我们画一下经典的GSEA图吧,不过这里比较的是两个通路的富集情况啦。🤩
1️⃣ MCF10A细胞系:👇
MCF10A.mtn.plot <- make_mountain_plots(targeted.MCF10A, set.names[1], set.names[2])
MCF10A.mtn.plot
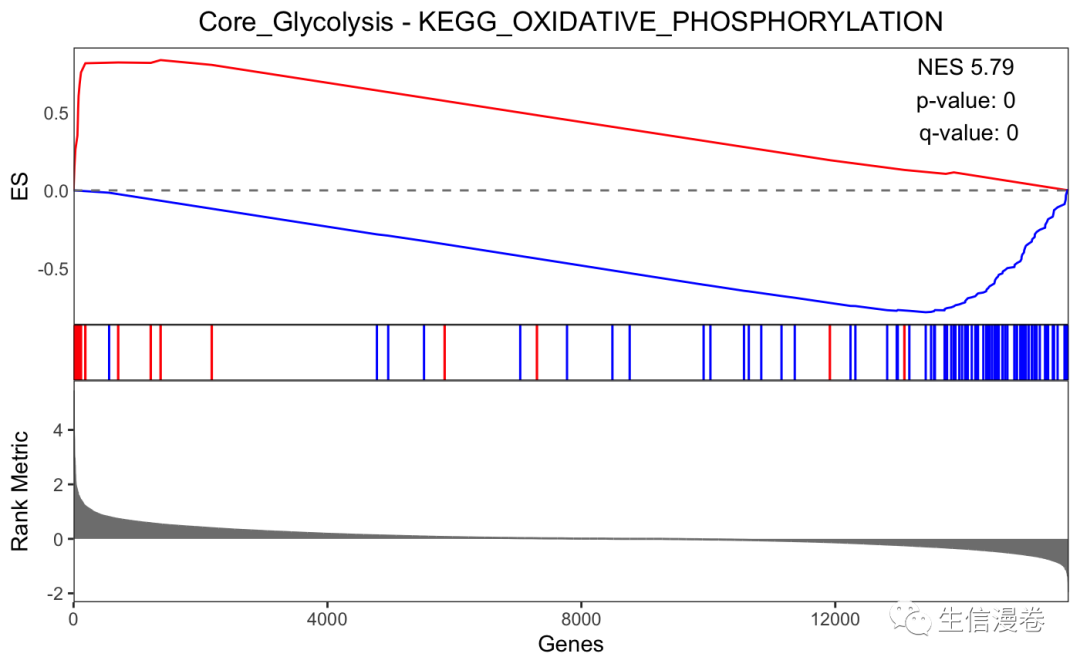
2️⃣ MCF12A细胞系:👇
MCF12A.mtn.plot <- make_mountain_plots(targeted.MCF12A, set.names[1], set.names[2])
MCF12A.mtn.plot
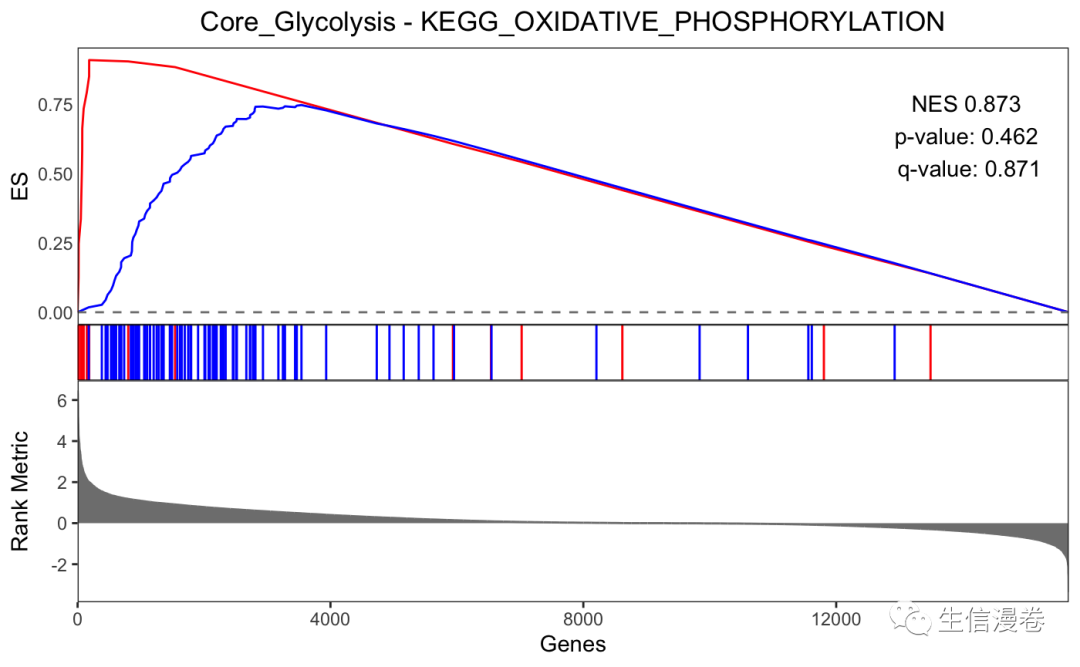
7非靶向DGSEA
有的时候我们也没有明确的需要比较的通路,这个时候可以做非靶向的DGSEA。😂
大家拿到DGSEA的结果后,就可以通过filter选取你需要的通路。🤠
untargeted.MCF10A <- dgsea_untargeted(MCF10A, kegg.pathways)
DT::datatable(untargeted.MCF10A$DGSEA.Results)
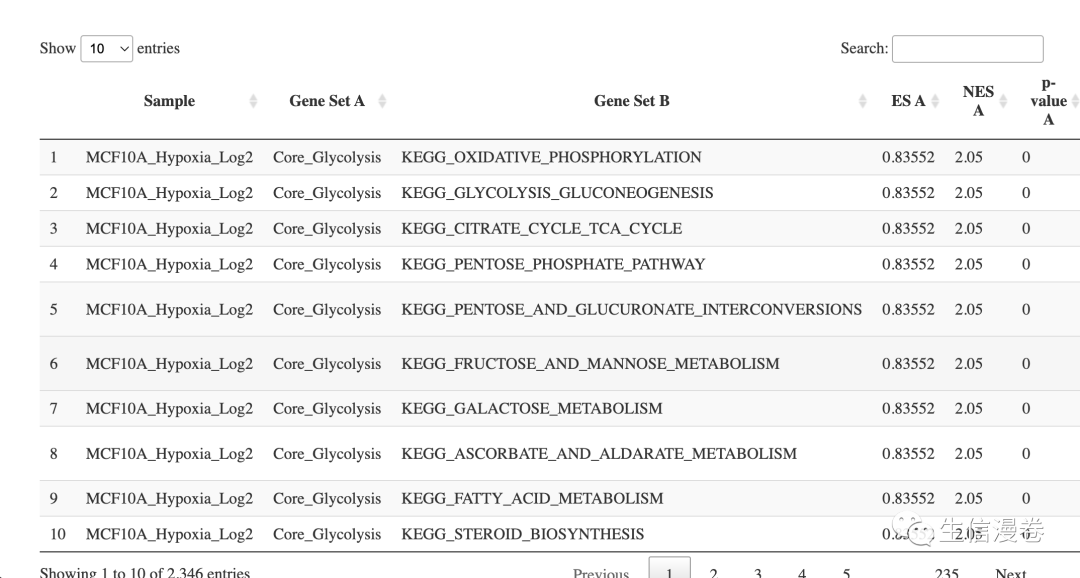
我们再来计算另一个细胞系的吧。😚
untargeted.MCF12A <- dgsea_untargeted(MCF12A, kegg.pathways)
DT::datatable(untargeted.MCF12A$DGSEA.Results)
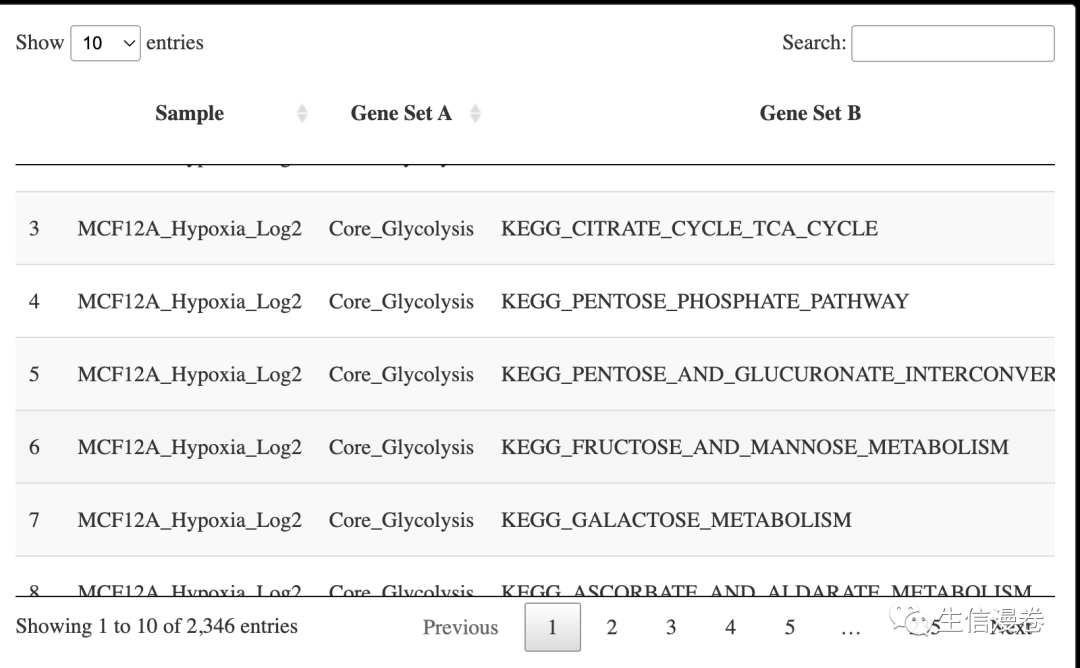
8如何引用
📍
Joly JH, Lowry WE, Graham NA. Differential Gene Set Enrichment Analysis: a statistical approach to quantify the relative enrichment of two gene sets. Bioinformatics. 2021;36(21):5247-5254. doi:10.1093/bioinformatics/btaa658
最后祝大家新的一年顺顺利利!~
