🤩 WGCNA | 值得你深入学习的生信分析方法!~(网状分析-第二步-网络构建与模块识别)
🤩 WGCNA | 值得你深入学习的生信分析方法!~(网状分析-第二步-网络构建与模块识别)
生信漫卷
发布于 2023-02-24 14:29:29
发布于 2023-02-24 14:29:29
文章被收录于专栏:R语言及实用科研软件
1写在前面
上期我们完成了WGCNA输入数据的清洗,然后进行了样本的聚类与异常值的剔除,总体来说是非常简单的。😘 这期我们继续完成WGCNA分析的第二步,网络构建和模块识别。🤒
2用到的包
rm(list = ls())
library(WGCNA)
library(tidyverse)
3示例数据
load("FemaleLiver-01-dataInput.RData")
4软阈值
4.1 topology analysis
首先我们要进行soft thresholding power β的计算。🤒
powers <- c(c(1:10), seq(from = 12, to=20, by=2))
sft <- pickSoftThreshold(datExpr, powerVector = powers, verbose = 5)
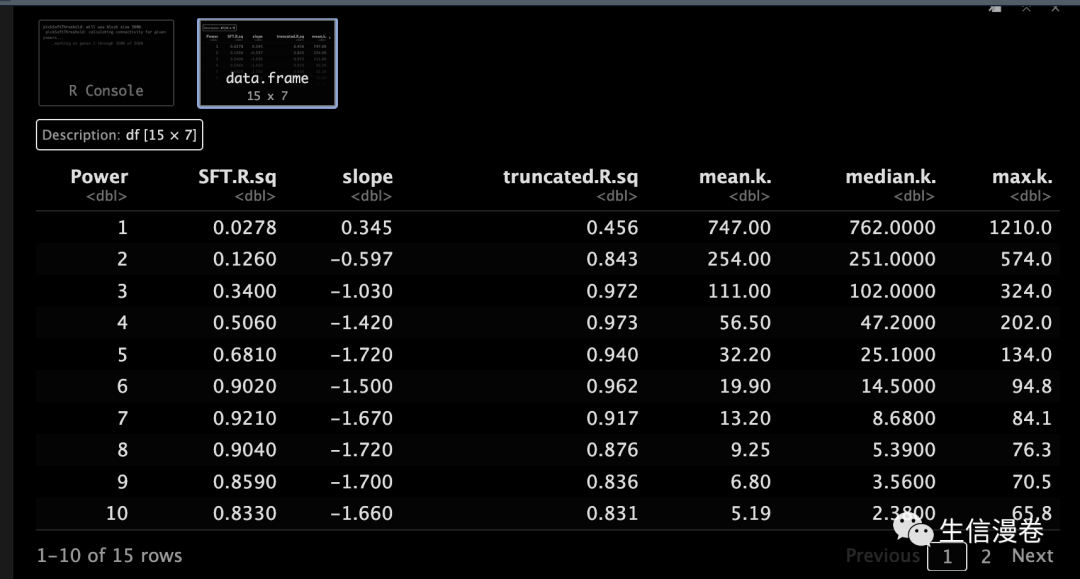
4.2 可视化
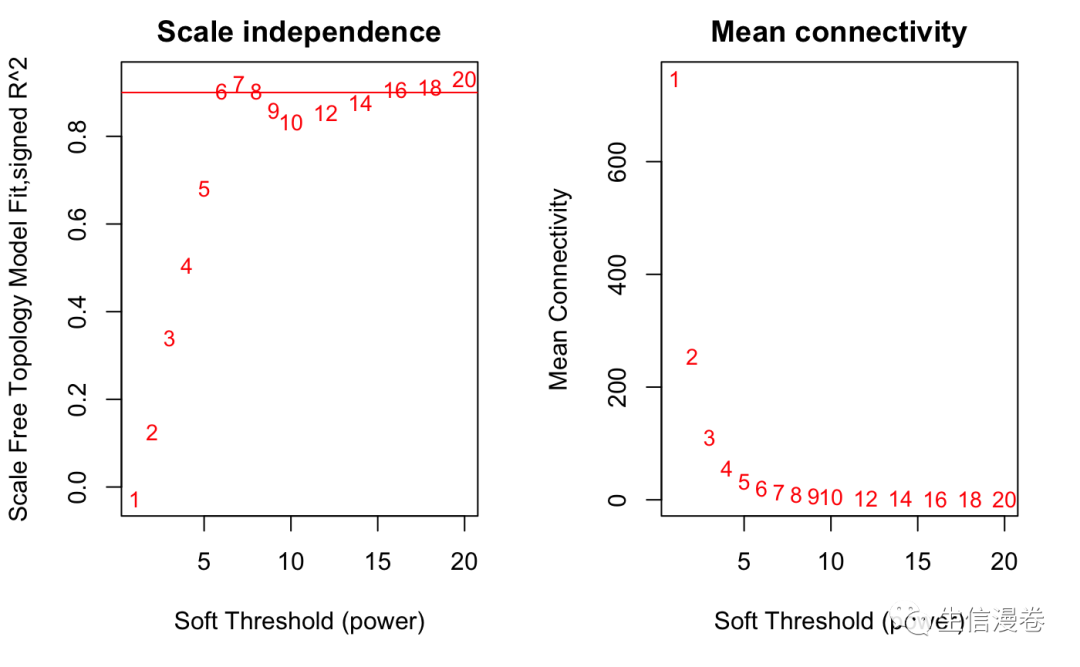
这个时候我们可以看到最佳的soft thresholding power β为6,后面我们会用到。😚
sizeGrWindow(9, 5)
par(mfrow = c(1,2))
cex1 = 0.9;
plot(sft$fitIndices[,1], -sign(sft$fitIndices[,3])*sft$fitIndices[,2],
xlab="Soft Threshold (power)",
ylab="Scale Free Topology Model Fit,signed R^2",
type="n",
main = paste("Scale independence"))
text(sft$fitIndices[,1], -sign(sft$fitIndices[,3])*sft$fitIndices[,2],
labels=powers,cex=cex1,col="red")
abline(h=0.90,col="red")
plot(sft$fitIndices[,1], sft$fitIndices[,5],
xlab="Soft Threshold (power)",
ylab="Mean Connectivity",
type="n",
main = paste("Mean connectivity"))
text(sft$fitIndices[,1], sft$fitIndices[,5], labels=powers, cex=cex1,col="red")
5构建网络与模块识别(一步法)
这里我们只需要一个函数就可以完成网络构建和模块识别,即blockwiseModules,不过参数比较多,大家需要去好好理解,这里不做太多介绍了,大家看帮助文档吧。🧐
5.1 构建网络
这里会有一个maxBlockSize的参数,大家可以根据自己的电脑配置进行选择,16 GB内存最大值为20000,32 GB内存最大值为30000。🤨
net <- blockwiseModules(datExpr, power = 6,
TOMType = "unsigned",
minModuleSize = 30,
reassignThreshold = 0, mergeCutHeight = 0.25,
numericLabels = T, pamRespectsDendro = F,
saveTOMs = T,
saveTOMFileBase = "femaleMouseTOM",
verbose = 3)
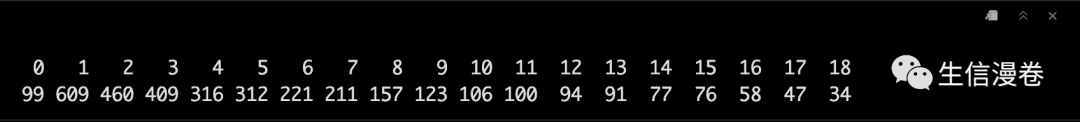
5.2 查看模块数
table(net$colors)
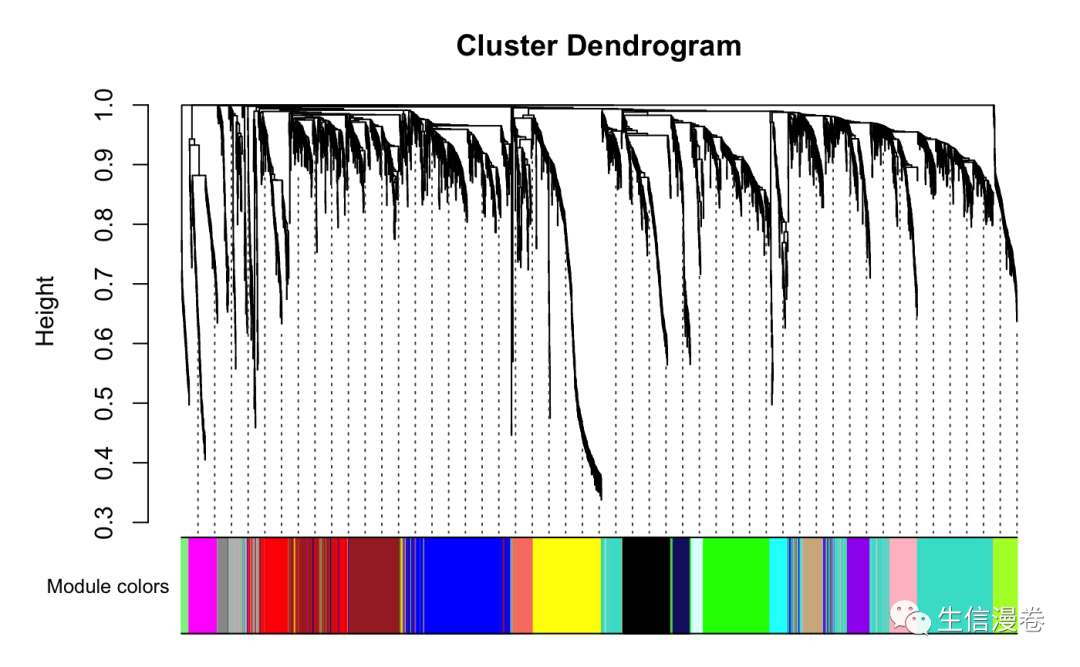
5.3 可视化
sizeGrWindow(12,9)
mergedColors <- labels2colors(net$colors)
plotDendroAndColors(net$dendrograms[[1]], mergedColors[net$blockGenes[[1]]],
"Module colors", dendroLabels = F, hang = 0.03, addGuide = T,
guideHang = 0.05)
5.4 save一下data
moduleLabels <- net$colors
moduleColors <- labels2colors(net$colors)
MEs <- net$MEs
geneTree <- net$dendrograms[[1]]
save(MEs, moduleLabels, moduleColors, geneTree,
file = "FemaleLiver-02-networkConstruction-auto.RData")
6构建网络与模块识别(分步法)
6.1 共表达相似性与邻接距离计算
这里我们把之前算好的power应用一下。😚
softPower <- 6
adjacency <- adjacency(datExpr, power = softPower)
6.2 计算TOM
这里我们需要把邻接矩阵转换为Topological Overlap Matrix(TOM矩阵),最大限度地减少噪音和假相关性。🧐
TOM <- TOMsimilarity(adjacency);
dissTOM <- 1-TOM

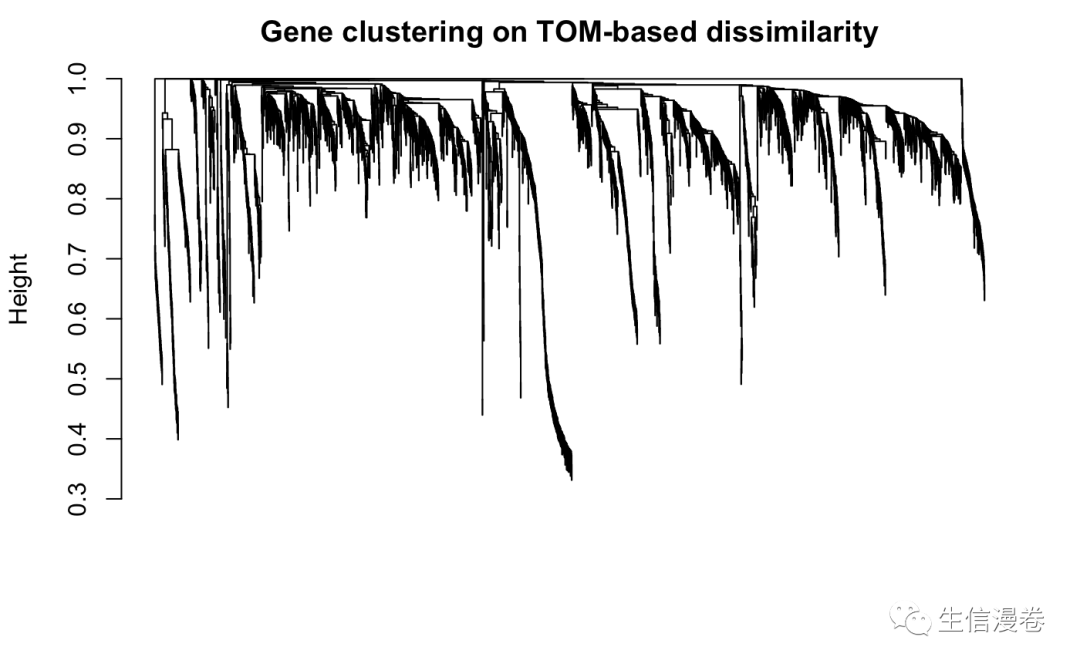
6.3 基因聚类并可视化
geneTree <- hclust(as.dist(dissTOM), method = "average")
plot(geneTree,
xlab="", sub="",
main = "Gene clustering on TOM-based dissimilarity",
labels = F, hang = 0.04)
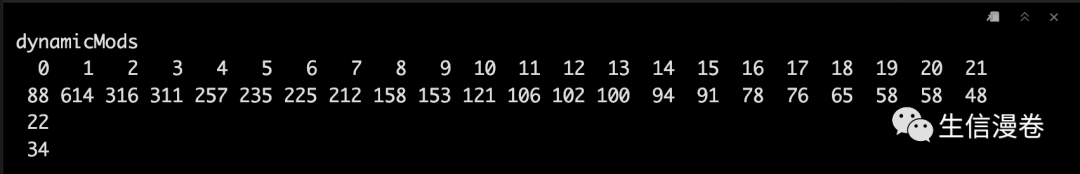
6.4 识别模块
我们一般喜欢基因多一点的模块,这里将最小的模块设置为30。😏
补充一下 !这里0模块是指未能归类到任意模块的基因。😷
minModuleSize <- 30
dynamicMods <- cutreeDynamic(dendro = geneTree, distM = dissTOM,
deepSplit = 2, pamRespectsDendro = F,
minClusterSize = minModuleSize)
table(dynamicMods)
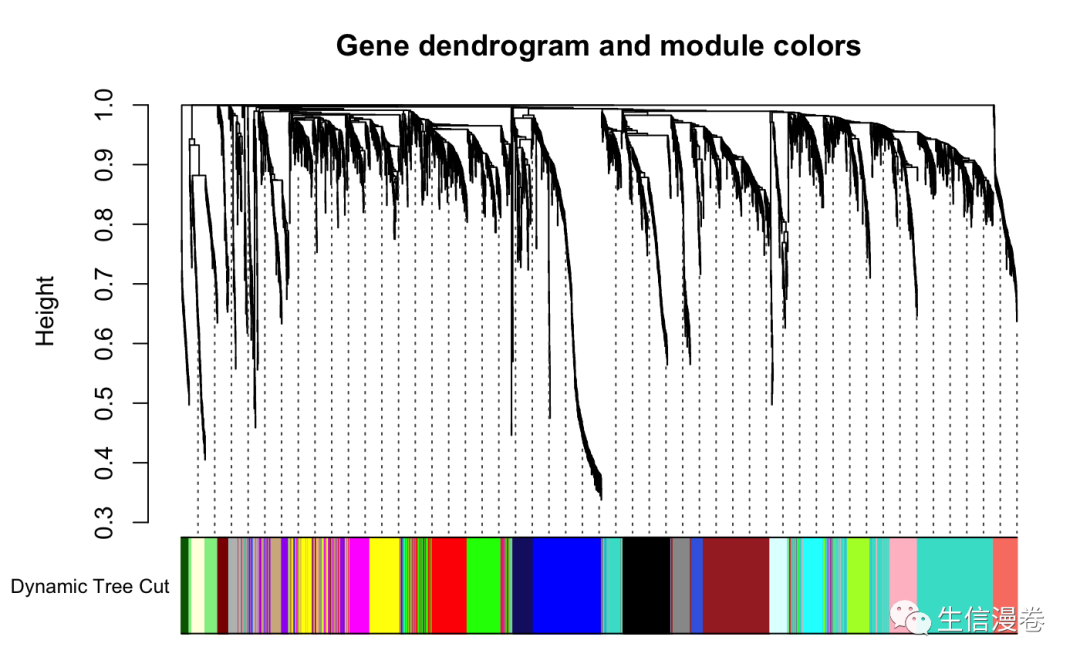
6.5 数字转为颜色并可视化
上面的模块为数字,我们需要把它转成颜色进行可视化。🤨
dynamicColors <- labels2colors(dynamicMods)
table(dynamicColors)
sizeGrWindow(8,6)
plotDendroAndColors(geneTree, dynamicColors,
"Dynamic Tree Cut",
dendroLabels = F, hang = 0.03,
addGuide = T, guideHang = 0.05,
main = "Gene dendrogram and module colors")
6.6 合并表达相似的模块
MEList <- moduleEigengenes (datExpr, colors = dynamicColors)
MEs <- MEList$eigengenes
MEDiss = 1-cor(MEs);
METree <- hclust(as.dist(MEDiss), method = "average")
6.7 合并模块的可视化
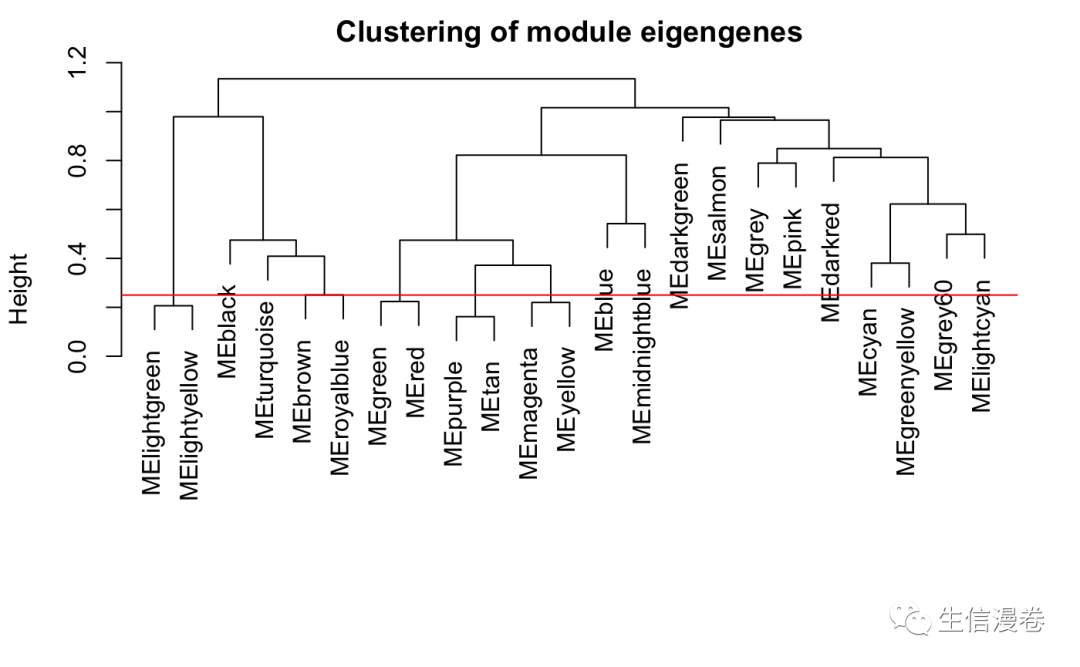
这里我们将相关性在0.75以上的模块合并在一起,当然你也可以按照你的要求来调整。🫶
sizeGrWindow(7,6)
plot(METree, main = "Clustering of module eigengenes",xlab = "", sub = "")
MEDissThres = 0.25
abline(h=MEDissThres, col = "red")
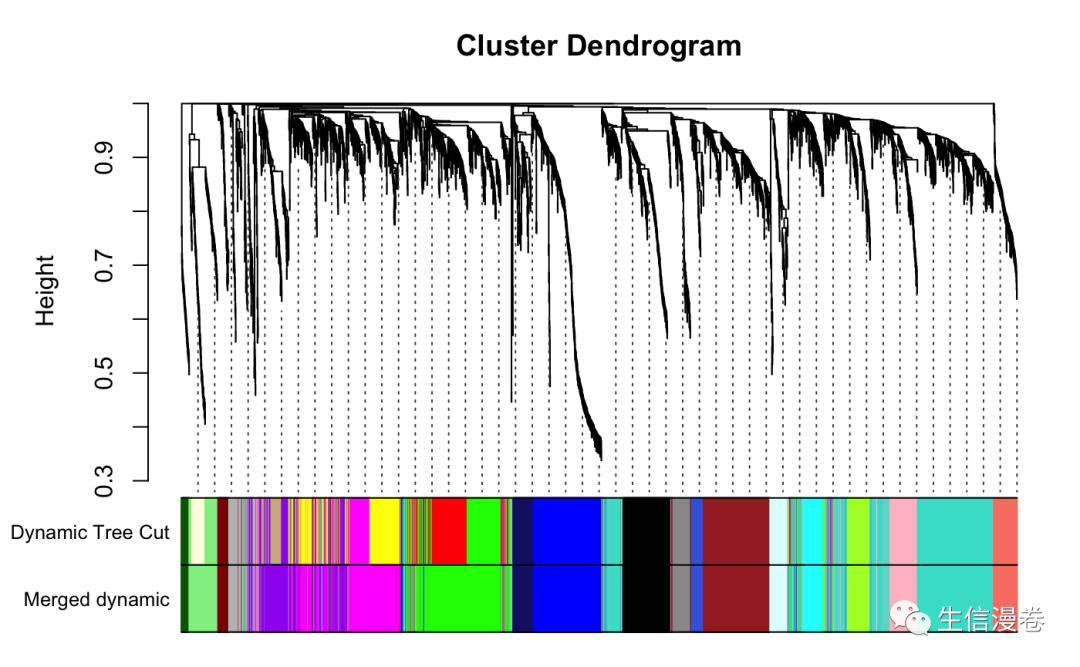
merge <- mergeCloseModules(datExpr, dynamicColors, cutHeight = MEDissThres, verbose = 3)
mergedColors <- merge$colors
mergedMEs <- merge$newMEs
sizeGrWindow(12,9)
plotDendroAndColors(geneTree, cbind(dynamicColors, mergedColors), c("Dynamic Tree Cut", "Merged dynamic"), dendroLabels = F, hang = 0.03, addGuide = T, guideHang = 0.05)
moduleColors <- mergedColors
colorOrder <- c("grey", standardColors(50))
moduleLabels <- match(moduleColors, colorOrder)-1
MEs <- mergedMEs
7save一下
我们save一下这个data吧,后面会再用到。😉
save(MEs, moduleLabels, moduleColors, geneTree,
file = "FemaleLiver-02-networkConstruction-stepByStep.RData")
8如何引用
📍
Langfelder, P., Horvath, S. WGCNA: an R package for weighted correlation network analysis. BMC Bioinformatics 9, 559 (2008). https://doi.org/10.1186/1471-2105-9-559
最后祝大家早日不卷!~
本文参与 腾讯云自媒体同步曝光计划,分享自微信公众号。
原始发表:2023-02-07,如有侵权请联系 cloudcommunity@tencent.com 删除
评论
登录后参与评论
推荐阅读
目录
