10X空间转录组平台vs国产空间平台(华大 & 百迈客)
原创作者,Evil Genius
4月9日,宜祈福,求个福,渡过难关,相亲相爱~~~
这一篇我们简单来比较一下10X空间转录组与国产空间转录组分析上的区别。
第一点、精度带来的分析区别
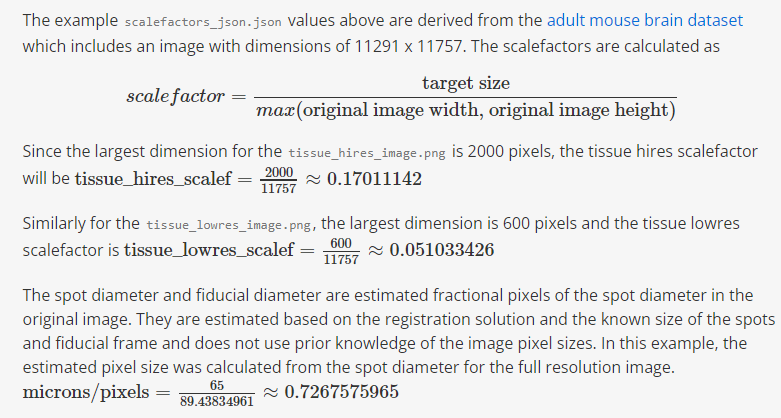
我们都知道10X空间转录组的精度为55um,这个精度是我们无法调整的,55um大多数情况下包含多个细胞,针对10X空间转录组的分析也开发了很多注释方法,这些都是固定的,我们知道即可,其中标准分析的结果里面有一个scalefactors_json.json,我们需要了解一下
tissue_hires_scalef: A scaling factor that converts pixel positions in the original, full-resolution image to pixel positions in tissue_hires_image.png. tissue_lowres_scalef: A scaling factor that converts pixel positions in the original, full-resolution image to pixel positions in tissue_lowres_image.png. fiducial_diameter_fullres: The number of pixels that span the diameter of a fiducial spot in the original, full-resolution image. spot_diameter_fullres: The number of pixels that span the diameter of a theoretical 65µm spot in the original, full-resolution image.
cat scalefactors_json.json
{"tissue_hires_scalef": 0.17011142,
"tissue_lowres_scalef": 0.051033426",
"fiducial_diameter_fullres": 144.4773339857,
"spot_diameter_fullres": 89.43834961021503}这个地方官方解释点的直径为65um,与我们通常宣传的55um有些出入,官方目前还没有给出很好的解释。
还有一个文件也需要我们关注一下,tissue_positions.csv:
This text file contains a table with rows that correspond to spots. From Space Ranger v2.0 onwards this file, which was previously named tissue_positions_list.csv, is renamed and includes a header column. Excluding the header column, the file has 4,992 rows for Visium slides with a 6.5 mm Capture Area and 14,336 rows for Visium slides with 11 mm Capture Area which is the number of spots in the spatial array. Columns correspond to the following fields: barcode: The sequence of the barcode associated to the spot. in_tissue: Binary, indicating if the spot falls inside (1) or outside (0) of tissue. array_row: For Visium slide (6.5 mm Capture Area), the row coordinate of the spot in the array from 0 to 77. The array has 78 rows. For Visium slides (11 mm Capture Area), the row coordinate of the spot range from 0 to 127 as the array has 128 rows. array_col: The column coordinate of the spot in the array. In order to express the "orange crate" arrangement of the spots, for Visium slides (6.5 mm Capture Area) this column index uses even numbers from 0 to 126 for even rows, and odd numbers from 1 to 127 for odd rows with each row (even or odd) resulting in 64 spots. For Visium slides (11 mm Capture Area) this column index uses even numbers from 0 to 222 for even rows, and odd numbers from 1 to 223 for odd rows with each row (even or odd) resulting in 111 spots. pxl_row_in_fullres: The row pixel coordinate of the center of the spot in the full resolution image. pxl_col_in_fullres: The column pixel coordinate of the center of the spot in the full resolution image.
$ cd /home/jdoe/runs/sample345/outs/spatial
$ column -s, -t < tissue_positions.csv | head -n 10
barcode in_tissue array_row array_col pxl_row_in_fullres pxl_col_in_fullres
GTCACTTCCTTCTAGA-1 0 0 0 1832 11971
CACGGTCTCCTTACGA-1 0 0 2 1832 11833
ATAGCTGCGGATAAGA-1 0 0 4 1832 11695
GTCAGTATGTCCGGCG-1 0 0 6 1832 11556
ATGTACCAGTTACTCG-1 0 0 8 1831 11418
ACGCTCAGTGCACCGT-1 0 0 10 1831 11280
TCACTAACGTATAGTT-1 0 0 12 1831 11142
CGGTTAGGCCTGGACG-1 0 0 14 1831 11004
GATATCACCAGCATGG-1 0 0 16 1831 10866从计算上来看,点之间的距离的pxl为138,那么就是100um的距离对应138个像素,上述的点的直径覆盖了89.43834961021503个像素,那么确实是对应的65um,这个地方还需要更多的解释,期待官方回复。
我们再来看国产国产平台的空间数据,国产平台都说自己是亚细胞级精度,但是亚细胞级精度是无法直接用于分析的,所以策略就是合并成一个super spot,很多时候合并后的直径比10X平台还要大。
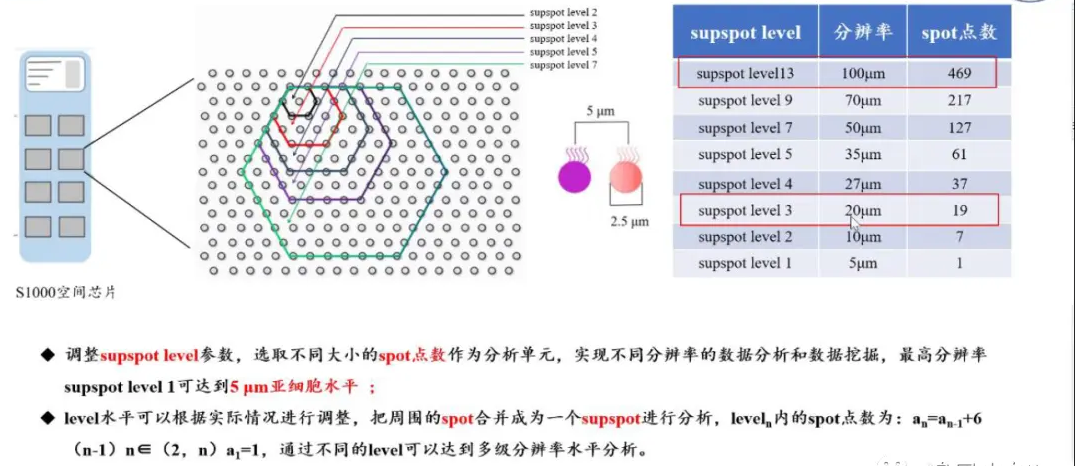
这里引用一篇用华大平台的文章作为辅助
Furthermore, to validate the spatial relationship at nearly single-cell resolution, we analyzed the mouse E13.5 FL ST data based on Stereo-seq, which is a sequencing-based spatially resolved transcriptomic technology with subcellular resolution.We defined 20 bins as a spot (10–15 μm in diameter), which may include 1–3 cell(s), and then annotated the spots (including intra-spotsStereo-seq, interspotsStereo-seq and other distant spotsStereo-seq) referring to the aforementioned pipeline.
也就是说bin20对应的直径为10-15um,也就是55um对应的是bin100左右,白迈客就是水平7左右,这个bin是可以调节的,大家可以根据自己的样本特性,要求公司或者自己对数据进行调节,比如自己的样本细胞大多数直径在20um,那么可以调节bin为40,这样理论上一个spot含有一个细胞,这个时候每个spot的检出基因数应该与单细胞的水平差不多,大家有数据的不妨试试,但实际上还是有很多问题。大家也可以把数据调整成一致对应看看基因检出数的差异。
第二点,软件集成度
不得不说大多数软件还是针对10X平台开发,集成度较好,国产平台目前没有很好地团队配合开发针对国产平台的软件,都是嫁接针对10X平台分析的软件,所以这方面欠缺一点。其中最主要的一个体现就是图片与数据的展示性问题,10X的平台很容易进行以下展示,底片和数据结合在一起,包括聚类结果,基因展示等等:
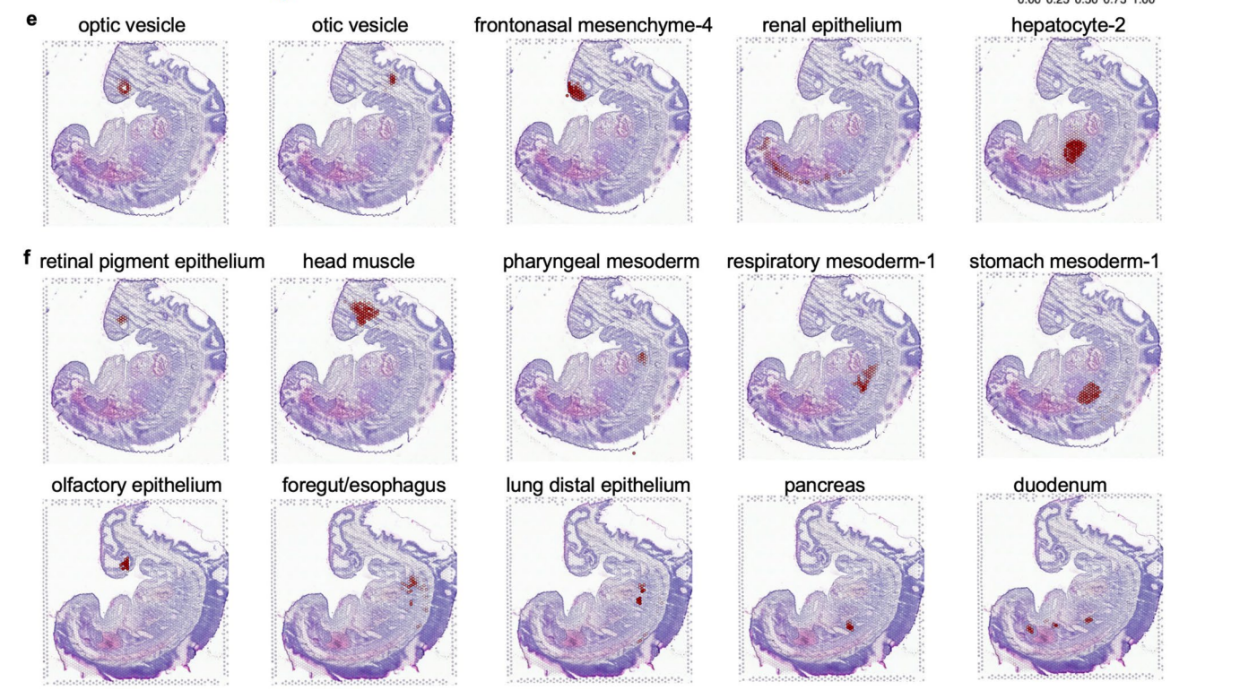
但是国产平台很难做到这一点,只能分开展示:
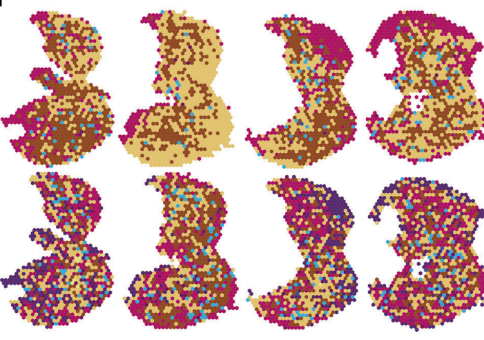
熟悉空间检测原理的同学也应该知道,即使调整到单细胞的精度,也不能保证每个spot就是含有一个细胞,那么在空间单细胞精度的分析条件下,就会有以下的分析优劣势:
优势:1、很多spot为单细胞精度,针对目标细胞的生态位细胞类型的富集有很强的应用型,但是可能与单细胞数据分析有出入,因为基因检出数也要达到单细胞的水准才可以。 2、如果数据可以,那么很多单细胞的分析方法直接可以运用到空间分析上(例如marker注释,通讯,CNV),相当于单细胞数据有了空间位置信息,只需要做空间转录组就可以了。 3、高精度在研究发育上有独特的优势
劣势:1、精度不好调节,因为细胞的大小存在很大的差异 2、我们的组织是一个严密的组织,细胞之间的空间位置很复杂,远不是调整成单细胞的精度就完全可以做到表征细胞空间信息,文章也证明了这一点,细胞更多呈现的是一种比例的变化。

3、bin一旦可以调节,那么就会存在不同的bin分析条件下结果会存在很大的出入,具体采用哪一个,需要人为判断,当然公司会按照一个固定的bin出一个报告,但仍需要自我判断。 4、精度越大,对实验的要求越高,“污染”的程度比10X要大。 5、肿瘤研究存在挑战
如果说国产平台在相同的直径下基因检出数不低于10X平台,那么国产平台将会有很大的应用,生活很好,有你更好
原创声明:本文系作者授权腾讯云开发者社区发表,未经许可,不得转载。
如有侵权,请联系 cloudcommunity@tencent.com 删除。
原创声明:本文系作者授权腾讯云开发者社区发表,未经许可,不得转载。
如有侵权,请联系 cloudcommunity@tencent.com 删除。