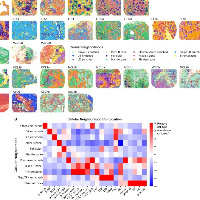
10X空间转录组通讯信号流的运用(COMMOT)
原创作者,Evil Genius
最近收到了很多粉丝的提问,其中对于空间通讯信号的用法需要在加强一点。
其实单独运用COMMOT做通讯信号流的做法并不完美,而实应该和空间轨迹向量场结合起来才更具有意义。
所以这里我们需要做到的是,
1、细胞的空间分布情况 2、细胞的空间分布趋势 3、细胞之间的共定位情况 4、细胞的信号流向
前三点其实分享了很多了,关于空间轨迹向量场也都分享过,这个地方我提醒大家一点,空间轨迹向量场表征的是细胞或者通路的聚集方向,信号流是通讯的方向,两者之间的结合才更具有意义
COMMOT软件特点 a package that infers CCC by simultaneously considering numerous ligand–receptor pairs for either spatial transcriptomics data or spatially annotated scRNA-seq data equipped with spatial distances between cells estimated from paired spatial imaging data; summarizes and compares directions of spatial signaling; identifies downstream effects of CCC on gene expressions using ensemble of trees models; and provides visualization utilities for the various analyses.
软件总览----COMMOT
配体和受体通常在有限的空间范围内与多种复合物相互作用。考虑到这一点,作者提出了具有三个重要特征的collective optimal transport:首先,the use of non-probability mass distributions to control the marginals of the transport plan to maintain comparability between species(需要一点数学背景知识);其次,对CCC实施空间距离约束,以避免连接空间上相距较远的细胞;最后,将多种配体分布结合到多中受体分布以解释多种相互作用。
这里我以一个空间肝癌样本为例,分享一下COMMOT的用法。
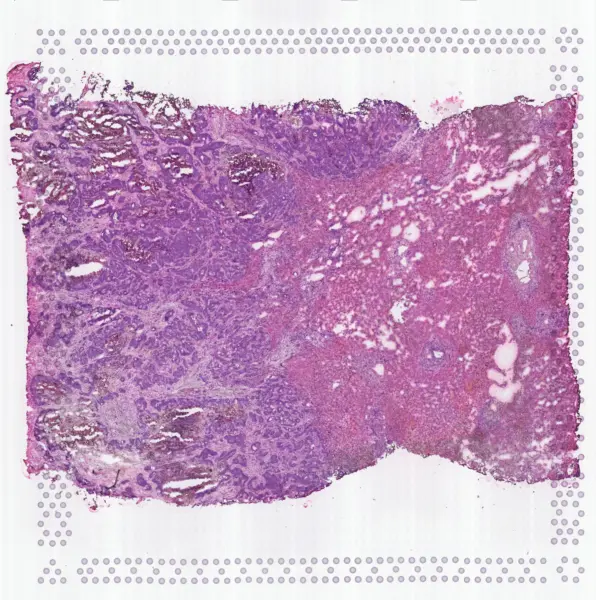
第一步看形态学划分,这个看HE图片应该都能看出来。
数据的简单处理
import os
import gc
import ot
import pickle
import anndata
import scanpy as sc
import pandas as pd
import numpy as np
from scipy import sparse
from scipy.stats import spearmanr, pearsonr
from scipy.spatial import distance_matrix
import matplotlib.pyplot as plt
import commot as ct
####读取空间数据
adata = sc.read_visium(sp_input,library_id = 'liver1')
adata.var_names_make_unique()
sc.pp.normalize_total(adata, inplace=True)
sc.pp.log1p(adata)
adata_dis500 = adata.copy()
sc.pp.highly_variable_genes(adata, min_mean=0.0125, max_mean=3, min_disp=0.5)
adata = adata[:, adata.var.highly_variable]
sc.tl.pca(adata, svd_solver='arpack')
sc.pp.neighbors(adata, n_neighbors=10, n_pcs=40)
sc.tl.umap(adata)
sc.tl.leiden(adata, resolution=0.4)这个地方我们看一下聚类和形态学的匹配度,癌区和正常区域有明显的划分
sc.pl.spatial(adata, color='leiden')
构建通讯网络
species = 'human'
df_cellchat = ct.pp.ligand_receptor_database(species=species, signaling_type='Secreted Signaling', database='CellChat')
df_cellchat_filtered = ct.pp.filter_lr_database(df_cellchat, adata_dis500, min_cell_pct=0.05)ct.pp.filter_lr_database这个地方有一个参数需要注意,min_cell_pct,空间的通讯信号方向性我们需要知道主导的信号,这个值适当大一点。
看一下信息:
df_cellchat_filtered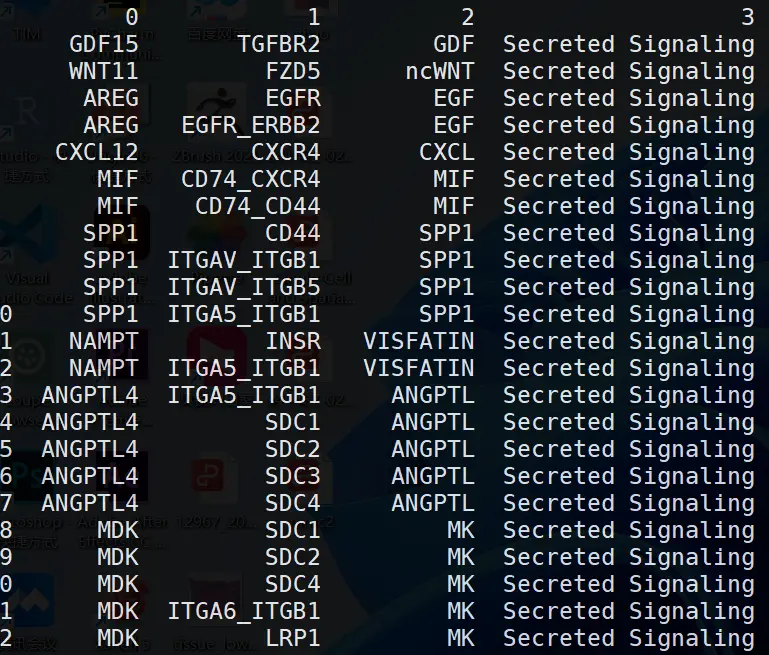
每列依次为配体、受体、信号通路,配受体类型,我们在计算的时候要把所有的信号都进行计算,但是时间比较长,以其中EGF通讯为例。
df_cellchat_filtered = df_cellchat_filtered.iloc[2:4,]
pathway = 'EGF'
ct.tl.communication_direction(adata_dis500, database_name='cellchat', pathway_name=pathway, k=5)
ct.pl.plot_cell_communication(adata_dis500, database_name='cellchat', pathway_name=pathway, plot_method='grid', background_legend=True,scale=0.00003, ndsize=8, grid_density=0.4, summary='sender', background='image', clustering='leiden', cmap='Alphabet',normalize_v = True, normalize_v_quantile=0.995)
ct.pl.plot_cell_communication(adata_dis500, database_name='cellchat', pathway_name=pathway, plot_method='grid', background_legend=True,scale=0.000005, ndsize=8, grid_density=0.4, summary='sender', background='image', clustering='leiden', cmap='Alphabet',normalize_v = True, normalize_v_quantile=0.995)
ct.pl.plot_cell_communication(adata_dis500, database_name='cellchat', pathway_name=pathway, plot_method='stream', background_legend=True,scale=0.00001, ndsize=8, grid_density=0.4, summary='sender', background='image', clustering='leiden', cmap='Alphabet',normalize_v = True, normalize_v_quantile=0.995)
可见信号流的方向是肿瘤指向正常区域。这个时候需要结合向量场的内容看细胞和通路的走向,参考文章空间轨迹向量场
如果我们需要把所有的内容全部输出,就需要进行脚本封装,实现COMMOT的全功能分析,封装好的代码放在这里。
import argparse
parse=argparse.ArgumentParser(description='COMMOT')
parse.add_argument('--sp_input',help='the input file of matrix for spatial',type=str,required = True)
parse.add_argument('--sample',help='the sample name',type=str,required = True)
parse.add_argument('--species',help='the species',type=str,required = True,choices = ['mouse','human'])
parse.add_argument('--outdir',help='the outdir',type=str,required = True)
argv=parse.parse_args()
sp_input=args.sp_input
sample = args.sample
species = args.species
outdir = args.outdir
import os
import gc
import ot
import pickle
import anndata
import scanpy as sc
import pandas as pd
import numpy as np
from scipy import sparse
from scipy.stats import spearmanr, pearsonr
from scipy.spatial import distance_matrix
import matplotlib.pyplot as plt
import commot as ct数据处理
adata = sc.read_visium(sp_input,library_id = sample)
adata.var_names_make_unique()
sc.pp.normalize_total(adata, inplace=True)
sc.pp.log1p(adata)
adata_dis500 = adata.copy()
sc.pp.highly_variable_genes(adata, min_mean=0.0125, max_mean=3, min_disp=0.5)
adata = adata[:, adata.var.highly_variable]
sc.tl.pca(adata, svd_solver='arpack')
sc.pp.neighbors(adata, n_neighbors=10, n_pcs=40)
sc.tl.umap(adata)
sc.tl.leiden(adata, resolution=0.4)
df_cellchat = ct.pp.ligand_receptor_database(species=species, signaling_type='Secreted Signaling', database='CellChat')
df_cellchat_filtered = ct.pp.filter_lr_database(df_cellchat, adata_dis500, min_cell_pct=0.05)
ct.tl.spatial_communication(adata_dis500,
database_name='cellchat', df_ligrec=df_cellchat_filtered, dis_thr=500, heteromeric=True, pathway_sum=True)
for pathway in list(set(df_cellchat_filtered.iloc[:,2])):
ct.tl.communication_direction(adata_dis500, database_name='cellchat', pathway_name=pathway, k=5)
ct.pl.plot_cell_communication(adata_dis500, database_name='cellchat', pathway_name=pathway, plot_method='grid', background_legend=True,scale=0.00001, ndsize=8, grid_density=0.4, summary='sender', background='image', clustering='leiden', cmap='Alphabet',normalize_v = True, normalize_v_quantile=0.995)
plt.savefig(outdir + '/' + sample + 'signal.arrow.grid.spatial.pdf',bbox_inches = 'tight')
ct.pl.plot_cell_communication(adata_dis500, database_name='cellchat', pathway_name=pathway, plot_method='stream', background_legend=True,scale=0.00001, ndsize=8, grid_density=0.4, summary='sender', background='image', clustering='leiden', cmap='Alphabet',normalize_v = True, normalize_v_quantile=0.995)
plt.savefig(outdir + '/' + sample + 'signal.arrow.stream.spatial.pdf',bbox_inches = 'tight')
生活很好, 有你更好
原创声明:本文系作者授权腾讯云开发者社区发表,未经许可,不得转载。
如有侵权,请联系 cloudcommunity@tencent.com 删除。
原创声明:本文系作者授权腾讯云开发者社区发表,未经许可,不得转载。
如有侵权,请联系 cloudcommunity@tencent.com 删除。