转座子分类软件deepTE使用中遇到的报错
deepTE的github主页
https://github.com/LiLabAtVT/DeepTE
单独新建一个虚拟环境
conda create -n DeepTE
安装3.6的python
conda activate DeepTE
conda install python=3.6 -y
然后安装指定的依赖python模块,这里python模块使用pip安装
pip install biopython==1.72-i https://mirrors.aliyun.com/pypi/simple/
pip install keras==2.2.4 -i https://mirrors.aliyun.com/pypi/simple/
pip install tensorflow==1.14.0 -i https://mirrors.aliyun.com/pypi/simple/
pip install sklearn -i https://mirrors.aliyun.com/pypi/simple/
然后使用conda安装hmmer
conda install hmmer -y
https://de.cyverse.org/dl/d/89D2FE7A-41BA-4F64-80E2-B9C26D49E99F/Plants_model.tar.gz
把这个文件下载下来再解压出来
运行命令
python /home/myan/biotools/DeepTE/DeepTE.py -i /home/myan/my_data/raw_data/longan/reference/longan-families_unknown.fa -sp P -d working.dir -o output_dir02 -m_dir /home/myan/biotools/DeepTE/Plants_model
会有报错
File "/home/myan/anaconda3/envs/DeepTE/lib/python3.6/site-packages/keras/engine/saving.py", line 224, in _deserialize_model
model_config = json.loads(model_config.decode('utf-8'))
AttributeError: 'str' object has no attribute 'decode'
我这里的办法是把saving.py这个文件里的所有.decode('utf-8')都给删掉
sed -i "s/.decode('utf-8')//g" /home/myan/anaconda3/envs/DeepTE/lib/python3.6/site-packages/keras/engine/saving.py
接下来还有报错
File "/home/myan/anaconda3/envs/DeepTE/lib/python3.6/site-packages/keras/engine/saving.py", line 229, in _deserialize_model
original_keras_version = model_weights_group['keras_version'].decode('utf8')
AttributeError: 'str' object has no attribute 'decode'
再把 把saving.py这个文件里的所有.decode('utf8')都给删掉
sed -i "s/.decode('utf8')//g" /home/myan/anaconda3/envs/DeepTE/lib/python3.6/site-packages/keras/engine/saving.py
这次再运行如下命令
python /home/myan/biotools/DeepTE/DeepTE.py -i /home/myan/my_data/raw_data/ABC/reference/ABC-families_unknown.fa -sp P -d working.dir -o output_dir02 -m_dir /home/myan/biotools/DeepTE/Plants_model
就没有报错了,
但是会有一堆提示信息
整个运行过程还挺快的,2000多条序列,90多秒就运行完了
输出文件夹 output_dir02必须提前新建好 working.dir这个文件夹会自己创建 output_dir02有两个文件
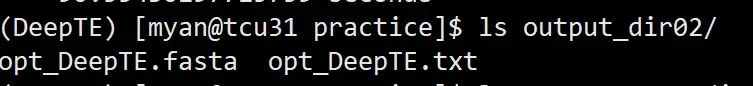
image.png
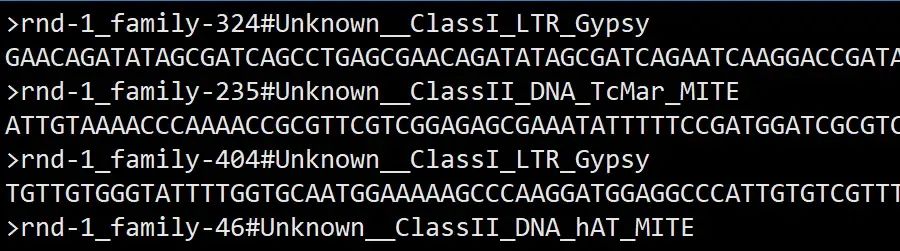
image.png
会在原fasta文件的基础上添加分类信息
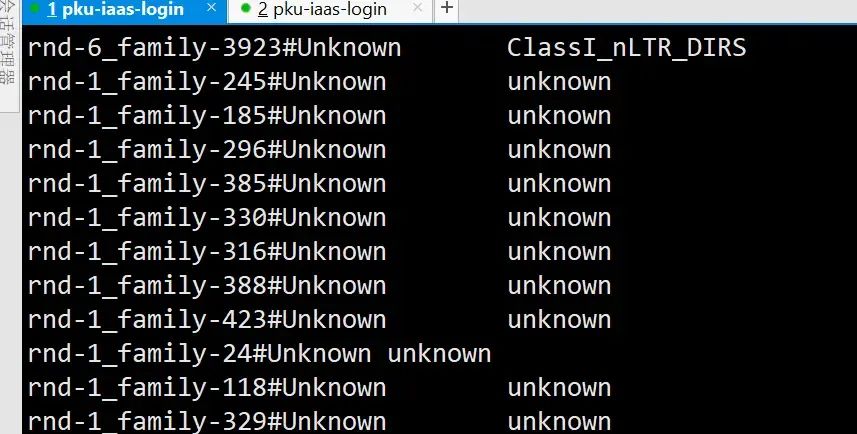
image.png
这个两列,第一列是原fasta的id 第二列是新的分类信息
推文记录的是自己的学习笔记,内容可能会存在错误,请大家批判着看,欢迎大家指出其中的错误
本文参与 腾讯云自媒体同步曝光计划,分享自微信公众号。
原始发表:2023-06-01,如有侵权请联系 cloudcommunity@tencent.com 删除
评论
登录后参与评论
推荐阅读
