解决方案 | 多个plink文件合并的方法
解决方案 | 多个plink文件合并的方法
邓飞
发布于 2023-10-27 16:09:31
发布于 2023-10-27 16:09:31
代码可运行
运行总次数:0
代码可运行
大家好,我是邓飞,这里总结一下多个plink文件合并的问题。
合并有两种应用场景:
- 1,样本一样,位点不一样,不如同样的样本,第一号染色体的数据,第二号染色体的数据合并。
- 2,位点一样,样本不一样,比如同样的芯片数据(map数据一样),第一批的数据,第二批的数据。
所以,这里也分为两种方法总结一下。
1. 样本一样,位点不一样
典型的情况:现在有4条染色体的数据,每个染色体一套plink文件,如何合并在一起。
比如数据:
dat_chr_1.map dat_chr_2.map dat_chr_3.map dat_chr_4.map
dat_chr_1.ped dat_chr_2.ped dat_chr_3.ped dat_chr_4.ped
这里使用--merge-list,对多个文件进行合并。
首先,我们先生成一个txt文件,把需要合并的ped和map数据的名称放进去,ped在前面,map在后面。
下面的文件名为:p12.txt,分为两列内容,第一列为ped的名称,第二列为map的名称,每一行都是一对plink文件。
dat_chr_1.ped dat_chr_1.map
dat_chr_2.ped dat_chr_2.map
dat_chr_3.ped dat_chr_3.map
dat_chr_4.ped dat_chr_4.map
代码如下:
plink --merge-list p12.txt --recode --out hebing
日志如下行:
$ plink --merge-list p12.txt --recode --out hebing
PLINK v1.90b6.21 64-bit (19 Oct 2020) www.cog-genomics.org/plink/1.9/
(C) 2005-2020 Shaun Purcell, Christopher Chang GNU General Public License v3
Logging to hebing.log.
Options in effect:
--merge-list p12.txt
--out hebing
--recode
15236 MB RAM detected; reserving 7618 MB for main workspace.
Performing single-pass merge (165 people, 426095 variants).
Merged fileset written to hebing.bed + hebing.bim + hebing.fam .
426095 variants loaded from .bim file.
165 people (80 males, 85 females) loaded from .fam.
112 phenotype values loaded from .fam.
Using 1 thread (no multithreaded calculations invoked).
Before main variant filters, 112 founders and 53 nonfounders present.
Calculating allele frequencies... done.
Total genotyping rate is 0.997722.
426095 variants and 165 people pass filters and QC.
Among remaining phenotypes, 56 are cases and 56 are controls. (53 phenotypes
are missing.)
--recode ped to hebing.ped + hebing.map ... done.
结果文件:
map数据之和,是合并后的map数据。
$ wc -l *map
119487 dat_chr_1.map
119502 dat_chr_2.map
98971 dat_chr_3.map
88135 dat_chr_4.map
426095 hebing.map
852190 total
ped数据不变:
$ wc -l *ped
165 dat_chr_1.ped
165 dat_chr_2.ped
165 dat_chr_3.ped
165 dat_chr_4.ped
165 hebing.ped
825 total
2. 位点一样,样本不一样
同样使用上面的方法。用--merge-list,然后定义名称的文件去进行合并。
这里用两个plink文件,sample1和sample2,多个文件操作方法是一样的。
sample1.map sample1.ped sample2.map sample2.ped
生成p12.txt文件:
sample1.ped sample1.map
sample2.ped sample2.map
运行命令合并:
plink --merge-list p12.txt --recode --out hebing2
日志如下:
$ plink --merge-list p12.txt --recode --out hebing2
PLINK v1.90b6.21 64-bit (19 Oct 2020) www.cog-genomics.org/plink/1.9/
(C) 2005-2020 Shaun Purcell, Christopher Chang GNU General Public License v3
Logging to hebing2.log.
Options in effect:
--merge-list p12.txt
--out hebing2
--recode
15236 MB RAM detected; reserving 7618 MB for main workspace.
Performing single-pass merge (25 people, 1457897 variants).
Merged fileset written to hebing2.bed + hebing2.bim + hebing2.fam .
1457897 variants loaded from .bim file.
25 people (13 males, 12 females) loaded from .fam.
17 phenotype values loaded from .fam.
Using 1 thread (no multithreaded calculations invoked).
Before main variant filters, 17 founders and 8 nonfounders present.
Calculating allele frequencies... done.
Total genotyping rate is 0.996107.
1457897 variants and 25 people pass filters and QC.
Among remaining phenotypes, 10 are cases and 7 are controls. (8 phenotypes are
missing.)
--recode ped to hebing2.ped + hebing2.map ... done.
Warning: 2 het. haploid genotypes present (see hebing2.hh ); many commands
treat these as missing.
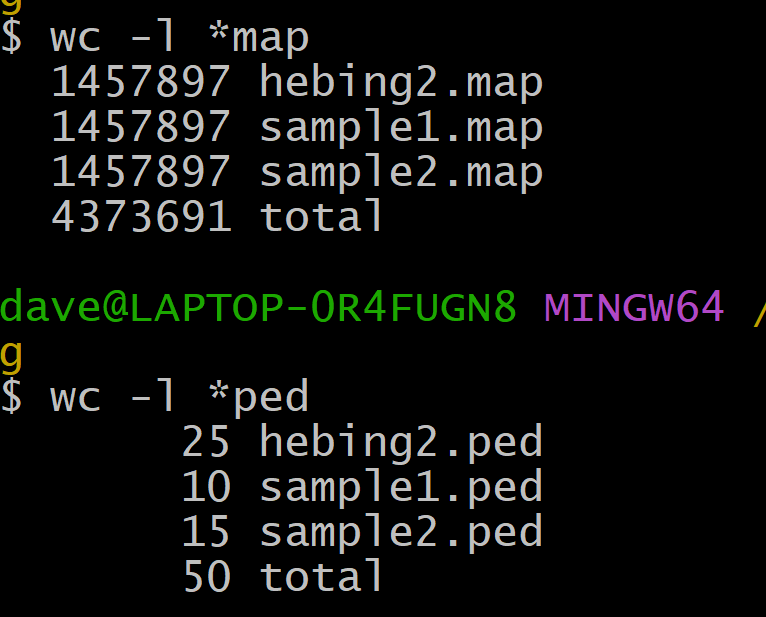
「结果如下:」
map数据完全一样,ped数据相加。
本文参与 腾讯云自媒体同步曝光计划,分享自微信公众号。
原始发表:2023-10-26,如有侵权请联系 cloudcommunity@tencent.com 删除
评论
登录后参与评论
推荐阅读
目录

