历史上那些经典的RNA-Seq数据比对软件
我们知道,mRNA 因为可变剪切丢失了内含子,因此不能像 DNA 数据那样简单地比对到基因组上。

如上图所示,mRNA reads 比对到基因组可能出现 3 种情况:
- read 完全比对到 1 个 exon 内(红色)
- read 跨越 2 个 exon(蓝色)
- read 跨越 2 个以上的 exon(紫色)
过去的 10 多年,已经开发了多种比对工具来应对快速增长的 RNA-Seq 测序数据,本文就来盘点一下其中几个比较经典的工具。
2009 TopHat
TopHat 是一款经典的 RNA-Seq 数据比对软件,能够精确地将测序 reads 比对到基因组上。其利用 Bowtie 进行快速比对,并考虑了剪接事件,提高了对剪接变异的检测灵敏度。曾经 Tophat + Cufflinks 作为转录组数据分析的标准流程,不知帮多少人完成了毕业论文,可以说为转录组学的发展立下了汗马功劳。但江山代有人才出,随着新软件工具的出现,经典也逐渐落幕,现在已经是不推荐使用了。
2013 TopHat2
4年之后,TopHat 迎来了升级版,采用了更先进的比对算法,提供更高的速度和准确性。支持多线程运行,同时考虑了单端和双端测序数据,有效识别复杂基因结构。
2013 STAR
STAR,最大的特点就是快,而且准确。它采用独特的分段式比对策略,优化了对基因组的多处比对。具有高效的多线程支持,适用于大规模测序数据。STAR 的开发得到了美国国立卫生研究院国家人类基因组研究所的支持。
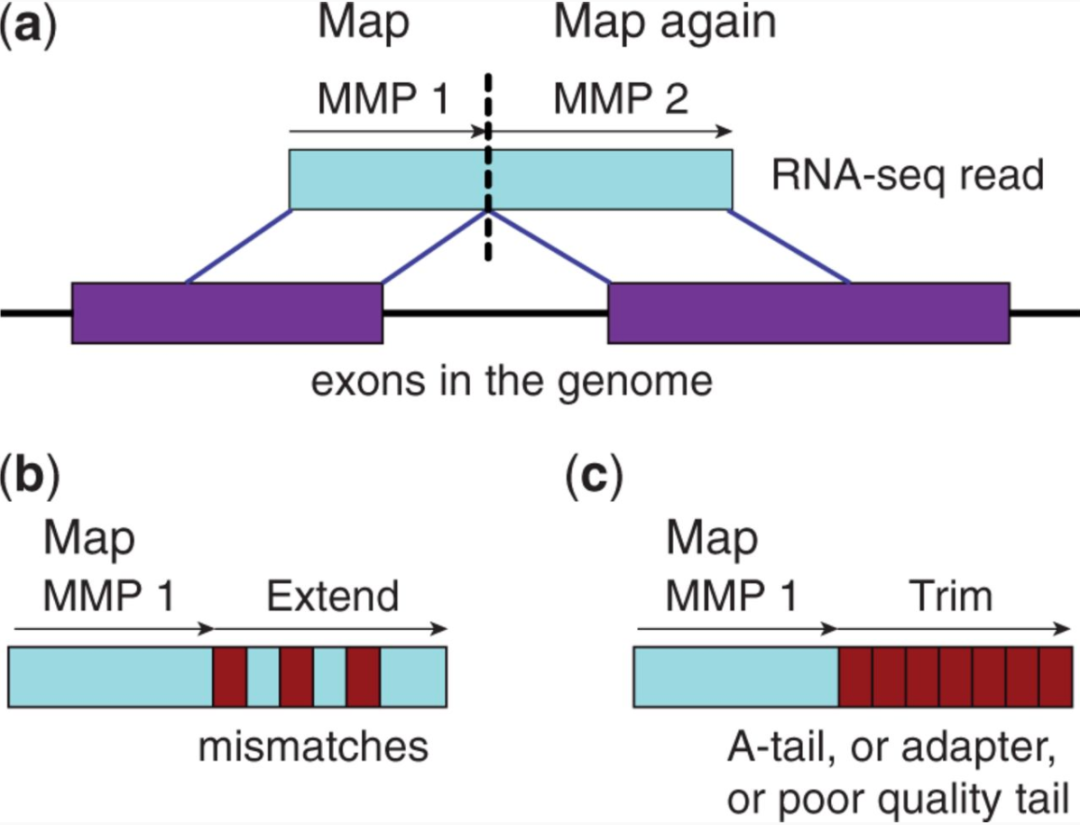
2015 HISAT
Tophat 作者团队在 2015 年开发了 HISAT,一种基于 Hierarchical Graph FM Index 的 RNA-Seq 比对工具,能够迅速、准确地处理大规模测序数据。具备较低的内存占用和高效的多线程处理能力。在 2016年,作者团队在 Nature Protocal 发表了新的转录组最佳实践流程:Hisat + StringTie + Ballown 组合。其中 Hisat 用于替换 Tophat,StringTie 用于替换 Cufflinks,R包 Bollown 用于差异表达等分析。我们 Galaxy 网站就集成了这一经典流程,详情见之前写的文章:云上生信,未来已来 | 转录组标准分析流程重磅上线!
2019 HISAT2
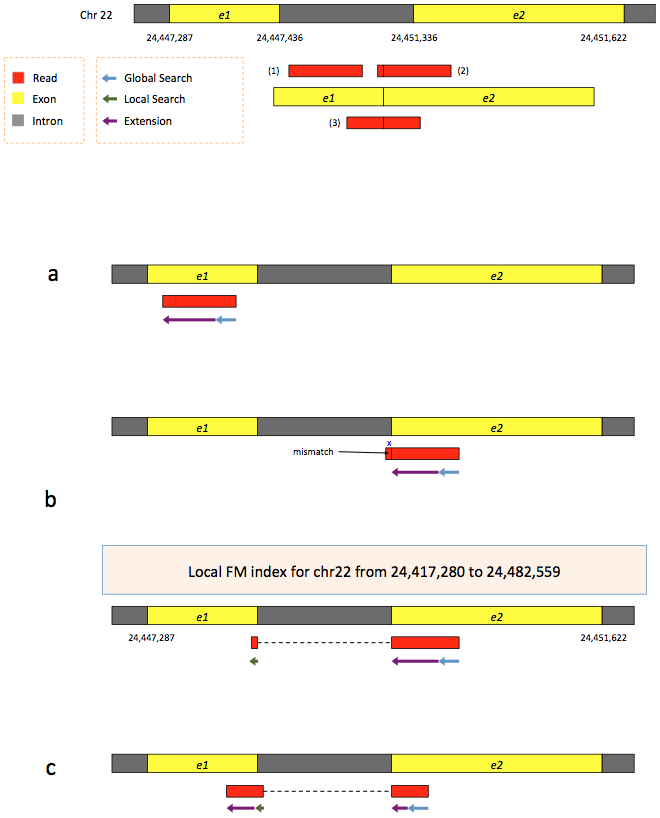
为了进一步优化和加速剪接读段比对,开发了 HISAT 的升级版 HISAT2。它使用分层图FM(HGFM)索引,代表整个基因组和最终的变体,以及重叠的局部索引(每个跨越~57 kb),共同覆盖基因组及其变体。这允许使用全局索引找到基因组中潜在读段比对的初始种子位置,并使用相应的局部索引快速细化这些比对。HISAT / HISAT2中的分层图FM索引见下图:
最后小结
目前最为流行的 RNA-Seq 数据比对软件是 HISAT2 和 STAR,它们可以说是大浪淘沙后的优胜者。特别值得一提的是,STAR 的升级版 STARsolo 在单细胞测序数据的比对和质控方面得到了越来越广泛的应用,感兴趣的小伙伴可以参考论文:STARsolo: accurate, fast and versatile mapping/quantification of single-cell and single-nucleus RNA-seq。

