Nature单细胞富集分析条形图复现
今天给大家复现一篇高分文章中对单细胞功能富集结果进行展示的条形图,此图无论是配色还是美观程度都还可以!
文章原图如下:

颜值是不是还可以!
首先拿到单细胞不同亚群的功能富集结果,此次,我们使用SeuratData包中的pbmcsca。
差异分析
使用FindAllMarkers函数对每个亚群进行差异分析,参数使用默认参数,如果自己做其他项目可以调整一些参数
rm(list=ls())
library(Seurat)
library(SeuratData)
library(clusterProfiler)
library(org.Hs.eg.db)
obj <- LoadData("pbmcsca")
obj
head(obj@meta.data)
# 使用原有的细胞注释
Idents(obj) <- "CellType"
# 得到每个细胞亚群的高表达基因
obj.markers <- FindAllMarkers(obj, only.pos = TRUE)
head(obj.markers)
p_val avg_log2FC pct.1 pct.2 p_val_adj cluster gene
CST7 0 Inf 0.635 0.145 0 Cytotoxic T cell CST7
GZMA 0 Inf 0.491 0.125 0 Cytotoxic T cell GZMA
PYHIN1 0 Inf 0.259 0.090 0 Cytotoxic T cell PYHIN1
SAMD3 0 Inf 0.198 0.055 0 Cytotoxic T cell SAMD3
ARL4C 0 Inf 0.549 0.305 0 Cytotoxic T cell ARL4C
C12orf75 0 Inf 0.351 0.112 0 Cytotoxic T cell C12orf75
功能富集
使用clusterProfiler进行KEGG Pathway通路富集分析,KEGG通路中基因为基因 ENTREZID,需要进行转换;当然如果是GO,直接设置keytype类型就可以了。
# 基因转成ENTREZID
Symbol <- mapIds(get("org.Hs.eg.db"), keys = obj.markers$gene, keytype = "SYMBOL", column="ENTREZID")
ids <- bitr(obj.markers$gene,"SYMBOL","ENTREZID", "org.Hs.eg.db")
# 合并ENTREZID到obj.markers中
data <- merge(obj.markers, ids, by.x="gene", by.y="SYMBOL")
head(data)
gene p_val avg_log2FC pct.1 pct.2 p_val_adj cluster ENTREZID
1 AAK1 1.191686e-262 Inf 0.385 0.192 4.015267e-258 CD4+ T cell 22848
2 AATF 1.298094e-03 Inf 0.103 0.091 1.000000e+00 Cytotoxic T cell 26574
3 ABCA8 2.613325e-34 0.6758008 0.565 0.079 8.805336e-30 Unassigned 10351
4 ABCE1 9.240937e-35 317.6269921 0.110 0.067 3.113641e-30 CD4+ T cell 6059
5 ABHD14B 7.371315e-69 106.9935161 0.173 0.099 2.483691e-64 CD4+ T cell 84836
6 ABHD17A 1.396019e-129 282.6004204 0.258 0.145 4.703745e-125 Cytotoxic T cell 81926
功能富集:compareCluster函数
gcSample <- split(data$ENTREZID, data$cluster)
gcSample
xx <- compareCluster(gcSample, fun="enrichGO", keytype="SYMBOL", OrgDb="org.Hs.eg.db", ont="BP", pvalueCutoff=1, qvalueCutoff=1)
xx <- compareCluster(gcSample, fun="enrichKEGG", organism="hsa", pvalueCutoff=1, qvalueCutoff=1)
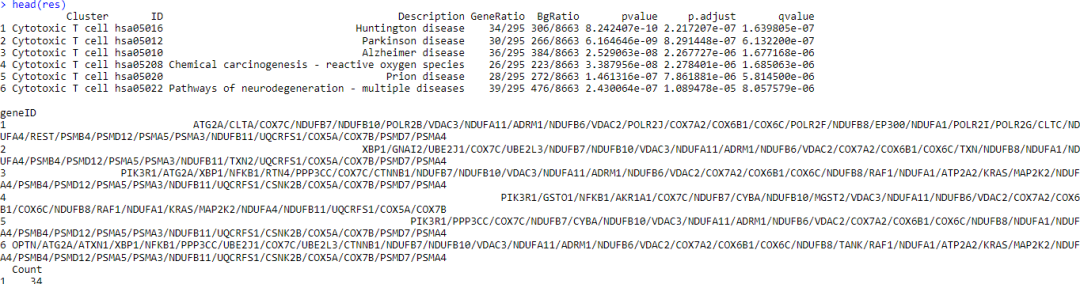
res <- xx@compareClusterResult
## 将富集结果中的 ENTREZID 重新转为 SYMBOL
for (i in 1:dim(res)[1]) {
arr = unlist(strsplit(as.character(res[i,"geneID"]), split="/"))
gene_names = paste(unique(names(Symbol[Symbol %in% arr])), collapse="/")
res[i,"geneID"] = gene_names
}
head(res)
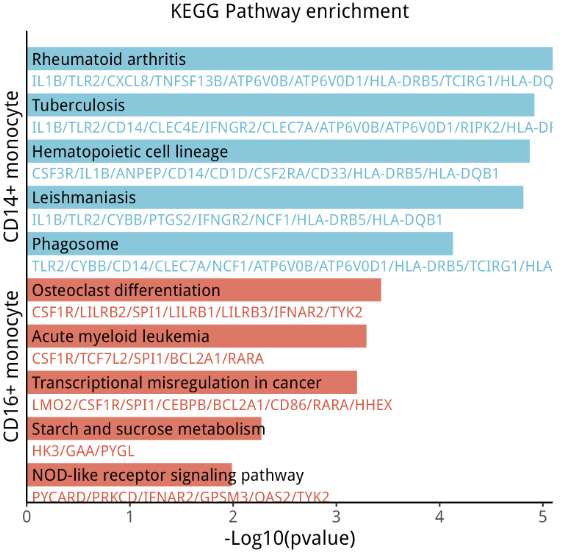
绘图
此次绘图使用ggplot,由于亚群数比较多,这里选择两个亚群的 pvalue top5通路进行展示
## 通路筛选
enrich <- res %>%
group_by(Cluster) %>%
top_n(n = 5, wt = -pvalue) %>%
filter(Cluster %in% c("CD14+ monocyte", "CD16+ monocyte"))
dt <- enrich
dt <- dt[order(dt$Cluster), ]
dt$Description <- factor(dt$Description, levels = dt$Description)
colnames(dt)
自定义主题:
# 先自定义主题:
mytheme <- theme(
axis.title = element_text(size = 13),
axis.text = element_text(size = 11),
axis.text.y = element_blank(), # 在自定义主题中去掉 y 轴通路标签:
axis.ticks.length.y = unit(0,"cm"),
plot.title = element_text(size = 13, hjust = 0.5, face = "bold"),
legend.title = element_text(size = 13),
legend.text = element_text(size = 11),
plot.margin = margin(t = 5.5, r = 10, l = 5.5, b = 5.5)
)
绘图,颜色当然可以跟文献中的一样,有很多种方法把文章中的颜色抠出来,后面专门介绍~
p <- ggplot(data = dt, aes(x = -log10(pvalue), y = rev(Description), fill = Cluster)) +
scale_fill_manual(values =c('#6bb9d2', '#d55640')) +
geom_bar(stat = "identity", width = 0.5, alpha = 0.8) +
scale_x_continuous(expand = c(0,0)) + # 调整柱子底部与y轴紧贴
labs(x = "-Log10(pvalue)", y = "CD16+ monocyte CD14+ monocyte", title = "KEGG Pathway enrichment") +
# x = 0.61 用数值向量控制文本标签起始位置
geom_text(size=3.8, aes(x = 0.05, label = Description), hjust = 0) + # hjust = 0,左对齐
geom_text(size=3, aes(x = 0.05, label = geneID), hjust = 0, vjust = 2.5, color=rep(c('#6bb9d2', '#d55640'),each=5)) + # hjust = 0,左对齐
theme_classic() +
mytheme +
NoLegend()
p
# 保存,这里的保存宽和高进行了调整,可以使得结果比较美观
ggsave(filename = "test.png", width = 5.2, height = 5.1, plot = p)
结果如下:
文献信息如下:
https://www.nature.com/articles/s41586-023-06783-1
CHIT1-positive microglia drive motor neuron ageing in the primate spinal cord
Nature. 2023 Dec;624(7992):611-620. doi: 10.1038/s41586-023-06783-1. Epub 2023 Oct 31
下期见~
本文参与 腾讯云自媒体同步曝光计划,分享自微信公众号。
原始发表:2024-03-16,如有侵权请联系 cloudcommunity@tencent.com 删除
评论
登录后参与评论
推荐阅读
目录
