PRGdb:植物R基因数据库
PRGdb:植物R基因数据库简要介绍
背景介绍
全世界近40%的作物产量因病虫害而损失,植物育种家和研究人员在寻找与植物抗病机制有关的基因方面付出了巨大的努力。植物通过进化发展出了识别潜在病原体和捕食者的能力,并激活防御机制来对抗它们。这些机制的激活是基于 病原体识别基因(PRGs) 编码的特定受体。但是通常植物与病原体相互作用的知识是通过对单个基因的研究获取的,而疾病反应是由高度连接的基因网络以及各种过程和途径之间共同调节的。
尽管在PRGs中存在不同的结构,富含亮氨酸重复序列(LRR)结构域具有普遍性。该结构域存在于模式识别受体(PRRs)中作为跨膜蛋白,PRRs识别外部信号并导致第一层诱导防御(也称为pmp触发免疫或PTI)。这些可以分为两大类:RLP,只包含LRR和跨膜结构域(TM);和RLKs,含有激酶(KIN)结构域。此外LRR结构域存在于NLR蛋白中,NLR蛋白还含有核苷酸结合位点(NBS)结构域。这些受体在细胞内引起更强大的免疫(效应触发免疫或ETI),也可以进一步分为两大类:TNL,它还含有一个toll -白细胞介素1受体(TIR)结构域;和CNL,携带一个额外的线圈(CC)结构域。同时也有受体具有其他结构域,而不是LRR结构域。
到目前为止在植物-病原体相互作用领域已经进行了大量的转录组研究,将转录组学建立为一个合适的平台来阐明这种相互作用的分子机制的复杂性。尽管组学和生信领域取得了进展,但数据探索性分析仍然是一项繁琐的任务,使用生物信息学工具研究PRGs对科学界的很大一部分仍然具有挑战性。数据库PRGdb就是为填补这一空白开发的,可以为研究植物抗病过程中涉及PRGs的基因提供参考。

PRGdb数据库
PRGdb简要介绍
新版本的植物抗性基因数据库(PRGdb;http://prgdb.org/prgdb4/)为了跟上可用知识和数据数量的增长(蛋白质组测序、克隆基因、公开分析数据等)进行了扩展。数据库网站更新了预测工具,增加了更多的数据和新的部分。新的部分包含植物抗性转录组实验,提供额外的易于访问的实验信息。作为PRGdb背后植物抗性基因的自动标注和预测工具,DRAGO3的准确性和灵敏度都得到了提高,预测结果更加的可靠。PRGdb从182个测序的蛋白质组中获得199个参考抗性基因和586.652个推定抗性基因。
与之前的版本相比,PRGdb 4.0将参考抗性基因的数量从153个增加到199个,推测抗性基因的数量从176个蛋白质组的177K增加到182个蛋白质组的586K。整合了公共转录组数据,这些转录组数据来自专注于五种农业兴趣物种的植物-病原体相互作用的研究。PRGdb的4.0版本中PRGs的种类已经扩展到7种,包括LYK、LYP和LECRK受体。注释工具,抗病分析和基因Orthology (DRAGO)也都得到了改进,可以提供更准确和敏感的注释任何给定的DNA或氨基酸(AA)序列。
PRGdb 4.0的创建可以为全球植物科学界和育种者提供参考,帮助进一步研究有助于对抗病原体的植物抗性机制。感兴趣可在http://prgdb.org/prgdb4/ 上浏览。
PRGdb数据库界面
新版本主要改进
建立了新的参考PRGs种类和蛋白质组
检索新的克隆耐药基因,搜索的目标扩展到LRR受体之外,涵盖了LYK、LYP和LECRK蛋白的克隆基因。使用公共预测工具InterProScan、Pfam、CDD、Smart和Prosite确认其结构域组成,建立了新的参考PRGs。新版本中共有51个蛋白被纳入参考PRGs。
建立了HMM
从PRGdb 4.0收录的7类耐药基因(CNL、TNL、RLK、RLP、LYK、LYP和LECRK)中分离出内参基因AA序列。使用MEGA X为每个类别构建多序列比对(MSA)。使用MSA构建隐马尔可夫模型(HMMs)。每个结构域的HMMs分别由每个类别的MSA构建,除了LYSM结构域(LYK和LYP蛋白也被组合在一起,为LYSM和LECM产生额外的HMMs)和LECM (LECRK内的两个亚组也构建了额外的HMMs:一个包含结构域legumeLECM结构域和另一个包含球型LECM结构域)。
使用InterProScan(激活SMART, Pfam, CDD和Prosite工具)定位MSA内的抗性结构域,并使用MEGA软件将MSA可视化并确定HMMs起源结构域。使用hmmsearch(hmmer工具;http://hmmer.org/)对比最初的FASTA文件,以测试它们是否确实对抗性域预测有用。
新的DRAGO3功能
基于BLoSuM62矩阵计算不同命中的对齐分数。对HMMs进行了更新(除了CNL和TNL类),并添加了三个新的蛋白质类:LYK, LYP和LECRK,以及所有其他非规范结构域组合。定义了DRAGO3的最小得分阈值,并分别使用COILS 2.2和TMHMM 2.0软件对CC域和TM域进行了相同的预测。
使用DRAGO2和DRAGO3分析了拟南芥蛋白质组的Araport 11注释,然后使用InterProScan分析获得的推定抗性基因,并根据两个标准与DRAGO结果进行比较:包含DRAGO和InterProScan预测相同的蛋白质;考虑DRAGO预测相同或更好的蛋白质。
从每个抗性等级的MSA中提取最保守的区域构建209个HMMs。根据以下标准对这些HMMs进行进一步过滤:属于不相关区域的HMMs(即TM域,它是用TMHMM工具分析而不是用DRAGO3 HMMs);不能识别用于构建它们的蛋白质的HMMs;将新HMMs与DRAGO2 HMMs进行比较,并保留性能更高的HMMs。
最终总共保留了109HMMs,几乎是之前DRAGO2的两倍。
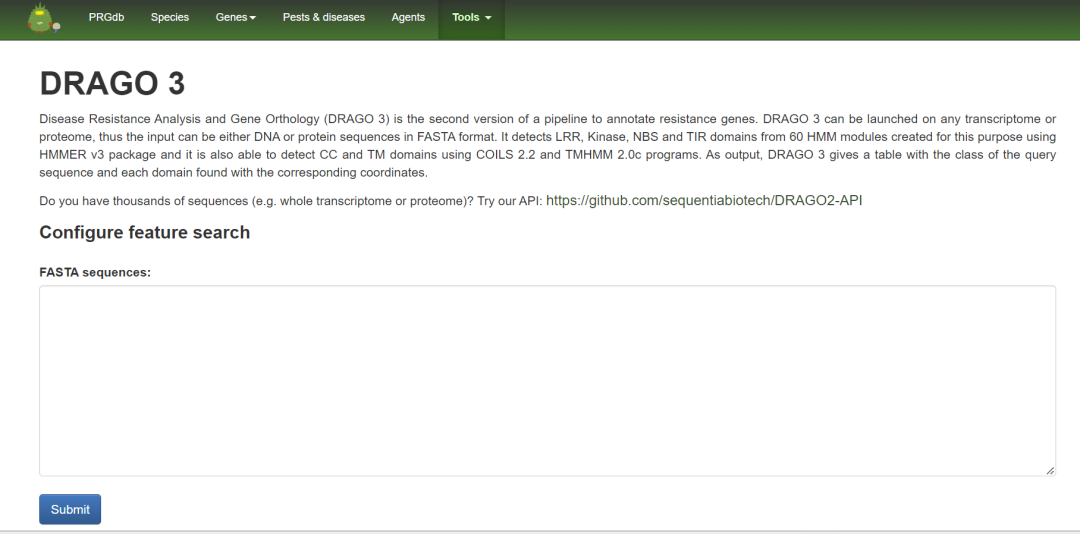
DRAGO3界面
公开RNA-seq实验数据
进行公开可用的RNA-seq实验,研究植物与五种农业作物的相互作用:番茄、水稻、小麦、葡萄和拟南芥。在这些研究中,不同的植物物种受到各种病原体的挑战,如细菌、真菌、昆虫和病毒。水稻、小麦、葡萄和拟南芥的DEGs列表从著作中检索。对于番茄,原始测序数据(fastq文件)从NCBI的SRA储存库下载(https://www.ncbi.nlm.nih.gov/sra) 使用SRA-toolkit (http://ncbi.github.io/sra-tools/),使用web-based A.I.R. RNAseq分析包在同一生信管道上进行分析(https://transcriptomics.sequentiabiotech.com/) 来探索和比较不同的研究。
除这之外新的数据库还进行了新注释,随着三个新的抗性类别LYK, LECRK和LYP的加入,PRGdb 4.0包括了植物中七个典型的抗性蛋白类别,并且能够预测超出这些已建立的类别的结构域组合。PRGdb 4.0共收录了199个参考抗性基因。利用DRAGO3分析182个植物蛋白质组及其在prgdb4.0中纳入的推定抗性基因,RLK和RLP仍然是最丰富的类别,抗性类LECRK、LYP和LYK含量最少。
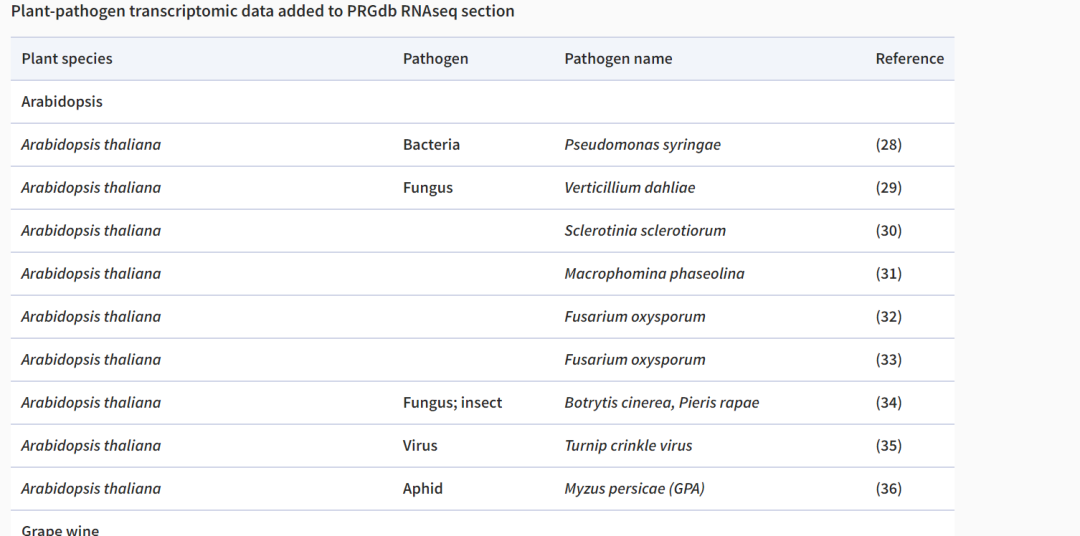
新增RNA-seq数据
经过植物-病原体转录组分析获得35个RNA-seq研究,获得了所有这些基因的差异表达基因(DEG)列表,并将这些数据纳入到PRGdb4.0的新部分中。
这部分很容易通过专门为五个物种提供表达分析的页面访问(图A)。在主页上显示了特定物种的可用实验(图B)。选择其中一个将把用户带到DEG矩阵(图C)。用户将能够探索各种研究的差异表达分析的结果。该信息显示为上调和下调基因的DEG矩阵,报告基因ID, log2倍变化,用于比较每个研究的兴趣和基因功能注释。用户可以下载csv格式的数据,可以按基因ID、日志FC和功能描述对数据进行排序。可为感兴趣的基因提供实验视角。
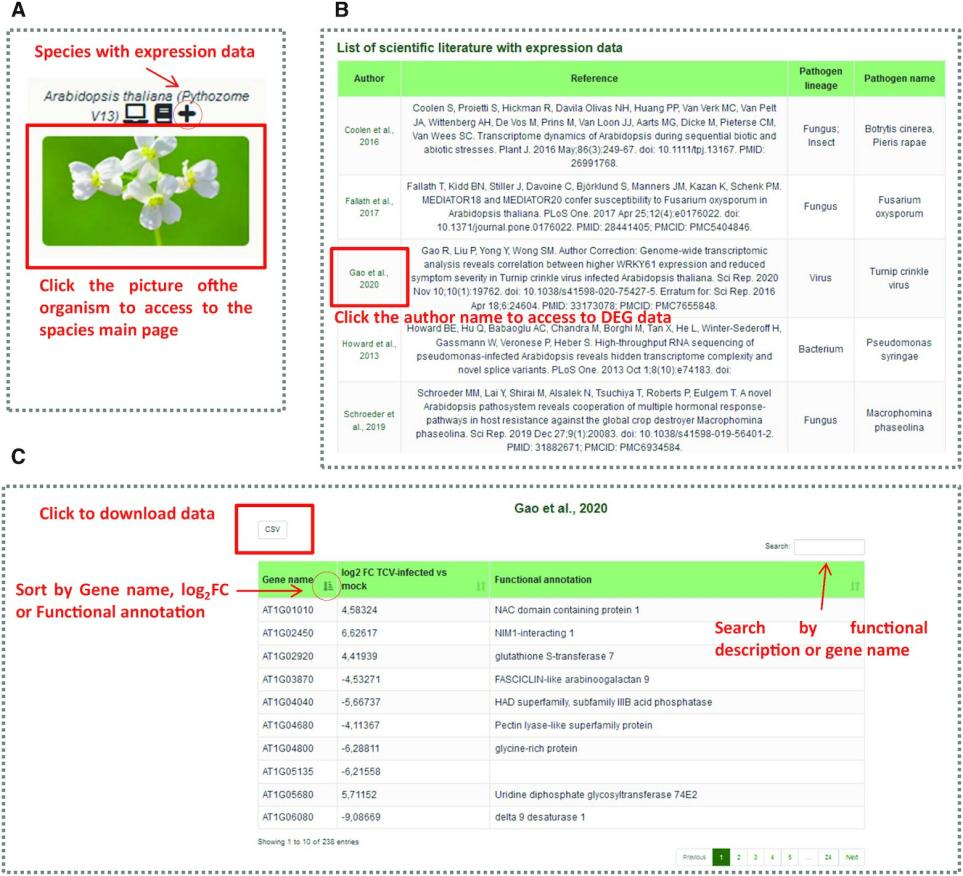
PRGdb4.0新部分
新版本的PRGdb 4.0增加了植物科学和育种界的效用。可以用于查询许多植物和藻类的植物抗性基因,分析抗性基因的序列以预测抗性基因,并研究特定植物病原体条件下基因的表达。新的蛋白质组的加入将使不可能的物种研究成为可能。随着新的参考PRGs和基因组公开化,PRGdb还会进行多组学数据整合,成为植物研究人员解决植物与病原体相互作用中的关键挑战的参考数据库。
感兴趣可阅读相关参考文献:
Calle García, J., Guadagno, A., Paytuvi-Gallart, A., Saera-Vila, A., Amoroso, C. G., D'Esposito, D., Andolfo, G., Aiese Cigliano, R., Sanseverino, W., & Ercolano, M. R. (2022). PRGdb 4.0: an updated database dedicated to genes involved in plant disease resistance process. Nucleic acids research, 50(D1), D1483–D1490.
Tan, Y. C., Kumar, A. U., Wong, Y. P., & Ling, A. P. K. (2022). Bioinformatics approaches and applications in plant biotechnology. Journal, genetic engineering & biotechnology, 20(1), 106.