难道铁死亡数据库记录的基因都是人类未知基因吗
最近看到了2023的一个数据库挖掘文章《Identification of ferroptosis-related genes in the progress of NASH》,里面有一个韦恩图展现了作者自己的转录组定量流程拿到的表达量矩阵的全部基因去跟铁死亡数据库记录的基因做交集,但是居然交集很少,如下所示:

居然交集很少
可以看到,铁死亡数据库记录的基因有接近40%都不在人类已经基因里面,非常的诡异。
人类这个物种的基因数量解析
只需要打开任意就近的GTF(Gene Transfer Format)文件即可,它是一种广泛使用的基因组注释文件格式,它详细描述了基因组中的各种基因特征。在GTF文件中,每一行代表一个基因组特征,并且通常会包含该特征的类型(gene_type)、位置、分数、链、相位以及属性等信息。属性字段(通常在第九列)可以包含多种不同的标签,其中就包括了gene_type,它描述了基因的具体类型。我们简单的统计如下所示:
grep -w gene /home/jmzeng/rna/human/pipeline/gencode.v39.annotation.gtf|cut -d";" -f 2|sort|uniq -c|sort -k1,1n
1056 gene_type "TEC"
1879 gene_type "miRNA"
1901 gene_type "snRNA"
2212 gene_type "misc_RNA"
2605 gene_type "unprocessed_pseudogene"
10157 gene_type "processed_pseudogene"
17755 gene_type "lncRNA"
19982 gene_type "protein_coding"
可以看到虽然这个gtf文件有6万多个基因,但是里面的蛋白质编码基因才2万个,下面是对上面的基因类型分类和数量的解释,这里是每种类型基因的简要解释:
- **miRNA (1879)**:微小RNA,是一种小的非编码RNA分子,通常参与RNA沉默和基因调控。
- **snRNA (1901)**:小核RNA,是组成核糖核蛋白复合体(如剪接体)的一部分,主要参与RNA的剪接过程。
- **misc_RNA (2212)**:其他类型的RNA,这个分类可能包括tRNA、rRNA以外的其他类型的小RNA分子。
- **unprocessed_pseudogene (2605)**:未处理的伪基因,这些是基因的一个非功能拷贝,它们曾经可能是功能性基因,但由于突变等原因,现在不能编码蛋白质。
- **processed_pseudogene (10157)**:经过处理的伪基因,它们是通过基因复制事件产生的,并且经历了与母基因相似的剪接过程,但通常不编码蛋白质。
- **lncRNA (17755)**:长链非编码RNA,这类RNA分子长度通常超过200个核苷酸,不编码蛋白质,但在细胞中扮演调控角色,如染色质重塑、基因表达调控等。
- **protein_coding (19982)**:编码蛋白质的基因,这是最常见的基因类型,它们含有编码序列,可以被翻译成蛋白质,执行细胞中的各种功能。
gtf文件有6万多个基因,但是在每次转录组测序数据分析的时候,绝大部分基因都是在所有的样品都是0,所以实际情况下每次转录组测序其实得到的所有的样品的涉及到的基因就是3万个左右,这个就是上面的韦恩图的左边部分!现在我们看看韦恩图的右边部分
铁死亡数据库记录的基因解析
目前最常用的铁死亡数据库包括:
- FerrDb:这是一个专门致力于铁死亡调节因子和铁死亡与疾病关联的数据库
- FerrDb V2:这是FerrDb数据库的更新版本,FerrDb V2收录了从3288篇论文中搜集到的1001个铁死亡调节因子(264个驱动基因、238个抑制基因、9个基因标志物、110个未分类基因、201个激活剂,179个抑制剂)和143个铁死亡--疾病相互关系。与FerrDb V1相比,覆盖的论文数量增加了3倍,铁死亡调节因子的数量增加了1.75倍,铁死亡--疾病相互关系的数量增加了约50%。环状RNA和假基因也被首次收录进来,非编码RNA的占比由7.3%增加到了13.6%。
在数据库官网可以下载到对应的文件:http://www.zhounan.org/ferrdb/current/operations/download.html
如果直接看最新版即可:
233 ferroptosis_disease.txt
370 ferroptosis_driver.txt
299 ferroptosis_inducer.txt
247 ferroptosis_inhibitor.txt
12 ferroptosis_marker.txt
117 ferroptosis_unclassified.txt
不知道为什么如果是下载ferroptosis_early_preview_upto20221231,可以看到数量要多一点:
292 disease.csv
630 driver.csv
576 inducer.csv
583 inhibitor.csv
105 marker.csv
729 suppressor.csv
126 unclassified.csv
我们试试看读取这个:
fs2 = lapply( list.files('ferroptosis_early_preview_upto20221231',full.names = T) ,
rio::import) #批量读取
lapply(fs2, dim)
lapply(fs2, head)
lapply(fs2, function(x){
length(unique(x[,2]))
})
lapply(fs2, function(x){
head(colnames(x))
})
可以很明显的看到有基因名字但是driver.csv以及 marker.csv ,suppressor.csv,还有unclassified.csv :
file coln
1 disease.csv RCD;Effect;Exp_organism;PMID;Remark;Latest_update
2 driver.csv RCD;Symbol_or_reported_abbr;HGNC_ID;ENSG_stable;Dependence;Evidence
3 inducer.csv RCD;Molecule;Name;PubChem_CID;PMID;Remark
4 inhibitor.csv RCD;Molecule;Name;PubChem_CID;PMID;Remark
5 marker.csv RCD;Symbol;HGNC_ID;ENSG_stable;Dependence;Evidence
6 suppressor.csv RCD;Symbol;HGNC_ID;ENSG_stable;Dependence;Evidence
7 unclassified.csv RCD;Symbol;HGNC_ID;ENSG_stable;Dependence;Evidence
基因的数量也很难跟原文一致,如果我们仔细看上面的文章《Identification of ferroptosis-related genes in the progress of NASH》,其实它使用的早期版本,可以看到:
FRGs were downloaded from FerrDb, an artificial ferroptosis database for managing and identifying ferroptosis-related markers, regulatory factors, and disorders
我们来尝试一下取交集
首先,我们需要一个表达量矩阵,这里使用最流行的airway数据集,如下所示:
library(airway,quietly = T)
data(airway)
rawcount <- assay(airway)
colnames(rawcount)
# 查看表达谱
rawcount[1:4,1:4]
dim(rawcount)
# 过滤在至少在75%的样本中都有表达的基因
keep <- rowSums(rawcount>0) >= floor(0.75*ncol(rawcount))
table(keep)
filter_count <- rawcount[keep,]
filter_count[1:4,1:4]
dim(filter_count)
可以很明显的看到,仅仅是最宽松的过滤,也会让表达量矩阵的6万多个基因变成2万左右:
> filter_count[1:4,1:4]
SRR1039508 SRR1039509 SRR1039512 SRR1039513
ENSG00000000003 679 448 873 408
ENSG00000000419 467 515 621 365
ENSG00000000457 260 211 263 164
ENSG00000000460 60 55 40 35
> dim(filter_count)
[1] 21930 8
> dim(rawcount)
[1] 63677 8
我们就拿上面的表达量矩阵,去跟前面的铁死亡基因数据库交集:
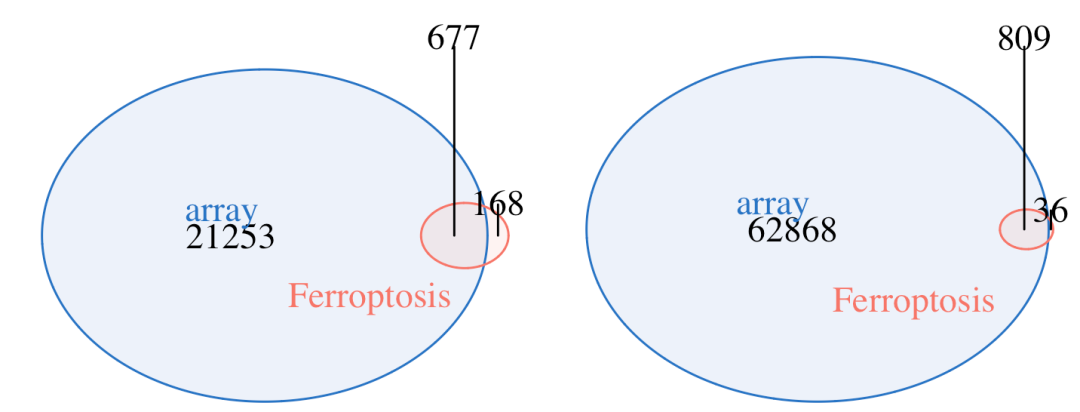
死亡基因数据库交集
很明显可以看到, 理论上铁死亡数据库记录的基因应该是会全部的在我们人类的已知的基因注释信息里面,哪怕是我们的过滤了三分之二后,仍然是可以有很好的交集。不应该是上面的文章《Identification of ferroptosis-related genes in the progress of NASH》那样的。