Sentieon 软件快速入门指南
原创Sentieon软件为完整的纯软件基因变异检测二级分析方案,其分析流程完全忠于BWA、GATK、MuTect2、STAR、Minimap2、Fgbio、picard等金标准的数学模型。在匹配开源流程分析结果的前提下,大幅提升WGS、WES、Panel、UMI、ctDNA、RNA等测序数据的分析效率和检出精度,并匹配目前全部第二代、三代测序平台,对短读长NGS、长读长longread测序数据进行SNP/INDEL/SV分析。最高支持20万人全基因组联合变异检测分析。
Sentieon为纯CPU计算加速软件,完全适配主流CPU计算架构:Intel、AMD、海光等X86架构CPU,华为鲲鹏、阿里倚天等ARM架构CPU。可灵活部署在实验室单机工作站、HPC集群、超算中心和云计算中心,保持同一套流程下不同规模数据计算结果的一致性。

Sentieon软件团队拥有丰富的软件开发及算法优化工程经验,致力于解决生物数据分析中的速度与准确度瓶颈,为来自于分子诊断、药物研发、临床医疗、人群队列、动植物等多个领域的合作伙伴提供高效精准的软件解决方案,共同推动基因技术的发展。 截至2023年底,Sentieon已经在全球范围内为1300+用户提供服务,被世界一级影响因子刊物如NEJM、Cell、Nature等广泛引用,引用次数近千篇。此外,Sentieon连续数年摘得了Precision FDA、Dream Challenges等多个权威评比的桂冠,在业内获得广泛认可。
一、运行环境
要开始使用Sentieon®软件,您需要以下条件:
1.1 硬件要求:
具有以下配置的Linux服务器:
•运行以下或更高版本的Linux发行版之一:RedHat/CentOS 6.5、Debian 7.7、OpenSUSE-13.2或Ubuntu-14.04。
•对于小型面板或全外显子组测序,需16GB内存;对于全基因组测序,需64GB内存。
•(推荐)最好使用高速SSD驱动器,以获得理想的I/O性能,实现最大CPU利用率。
1.2 软件要求:
•需要Python 2.6.x、Python 2.7.x或python3.x。您可以通过以下命令检查Python版本:
python --version1.3 软件安装包:
•(以v202308.03版本为例):
X86 CPU版本
ARM CPU版本
•使用以下命令解压,其中VERSION为您使用的版本,例如202308.03:
tar xvzf sentieon-genomics-VERSION.tar.gz1.4 许可证要求:
请参考“第四章节:许可证设置”了解如何设置许可证的详细信息。可能需要IT支持。
1.5 系统环境要求:
•如果Python 2.6.x、Python 2.7.x或python3.x不是默认Python版本,您可以设置以下环境变量:
export SENTIEON_PYTHON=Python_location•如果使用本地主机许可证文件,请设置以下环境变量,其中LICENSE_DIR为许可证文件所在目录,LICENSE_FILE.lic为许可证文件名:
export SENTIEON_LICENSE=LICSRVR_HOST:LICSRVR_PORT•如果用户使用许可证服务器,请设置以下环境变量,其中LICSRVR_HOST和LICSRVR_PORT分别为许可证服务器的主机名和端口。详情请参阅下一节。
export SENTIEON_LICENSE=LICSRVR_HOST:LICSRVR_PORT•为方便起见,请按如下方式设置二进制路径,其中PATH_TO_SENTIEON_BINARY_DIRECTORY为Sentieon®二进制文件的安装目录:
export SENTIEON_INSTALL_DIR=PATH_TO_SENTIEON_BINARY_DIRECTORY•使用NFS存储时,为提高性能,请将SENTIEON_TMPDIR环境变量设置为指向本地快速临时存储:
export SENTIEON_TMPDIR=/tmp二、首次运行作业
我们提供了一个快速入门演示项目,其中包含示例脚本和数据,以帮助您快速测试安装并诊断潜在问题。
quikstart演示项目:https://insvast-download.oss-cn-shanghai.aliyuncs.com/Sentieon/release/sentieon_quickstart.tar.gz
快速入门包包含单个染色体的数据,包括样本的序列数据和参考材料。作业脚本使用Sentieon DNAscope流程处理一组双端Illumina fastq文件:
•BWA: 将读段比对到参考基因组。
•Metrics和LocusCollector: 收集读段统计信息。
•Dedup: 去除重复读段。
•变异检测: DNAscope变异检测。
注意: DNAscope仅推荐用于二倍体生物的样本。对于其他样本,请使用DNAseq。
2.1 运行快速入门包
要开始使用,请将下载的快速入门包复制到新目录,并通过运行以下命令解压:
tar xzvf sentieon_quickstart.tar.gz包中包含以下内容:
•sentieon_quickstart.sh: 驱动整个流程的示例shell脚本。
•reference: 包含人类基因组参考文件和已知SNP位点数据库文件的目录。
•models: 包含DNAscope模型文件的目录。
•FASTQ文件: 样本序列文件。
在运行脚本之前,您需要确保正确设置了上述环境变量,包括许可证和目录路径。
然后使用您喜欢的编辑器编辑sentieon_quickstart.sh中的用户设置。
# 更新Sentieon软件包的位置
SENTIEON_INSTALL_DIR=/home/release/sentieon-genomics-202308.03
# 更新临时快速存储的位置并取消注释
#SENTIEON_TMPDIR=/tmp
# 在实际情况下分配有意义的名称很重要。
# 特别重要的是要为不同的读组分配不同的名称。
sample="sample_name"
group="read_group_name"
platform="ILLUMINA"
# 其他设置
nt=16 #计算中使用的线程数
# 数据是否使用无PCR文库制备
PCRFREE=true注意: 在用户设置shell脚本sentieon_quickstart.sh中:
•在实际情况下分配有意义的名称很重要。
•特别重要的是要为不同的读组分配不同的名称。
要获取CPU核心数,用户可以运行nproc,如下所示:
nproc要更好地理解sentieon_quickstart.sh脚本的其余部分,请阅读每个部分的注释以及手册中的相应章节。
现在,只需运行sentieon_quickstart.sh来启动脚本,并观察结果展开。在典型的Linux服务器上,整个运行大约需要3-5分钟。实际时间取决于计算环境。
sh sentieon_quickstart.sh &三、 Sentieon模块描述
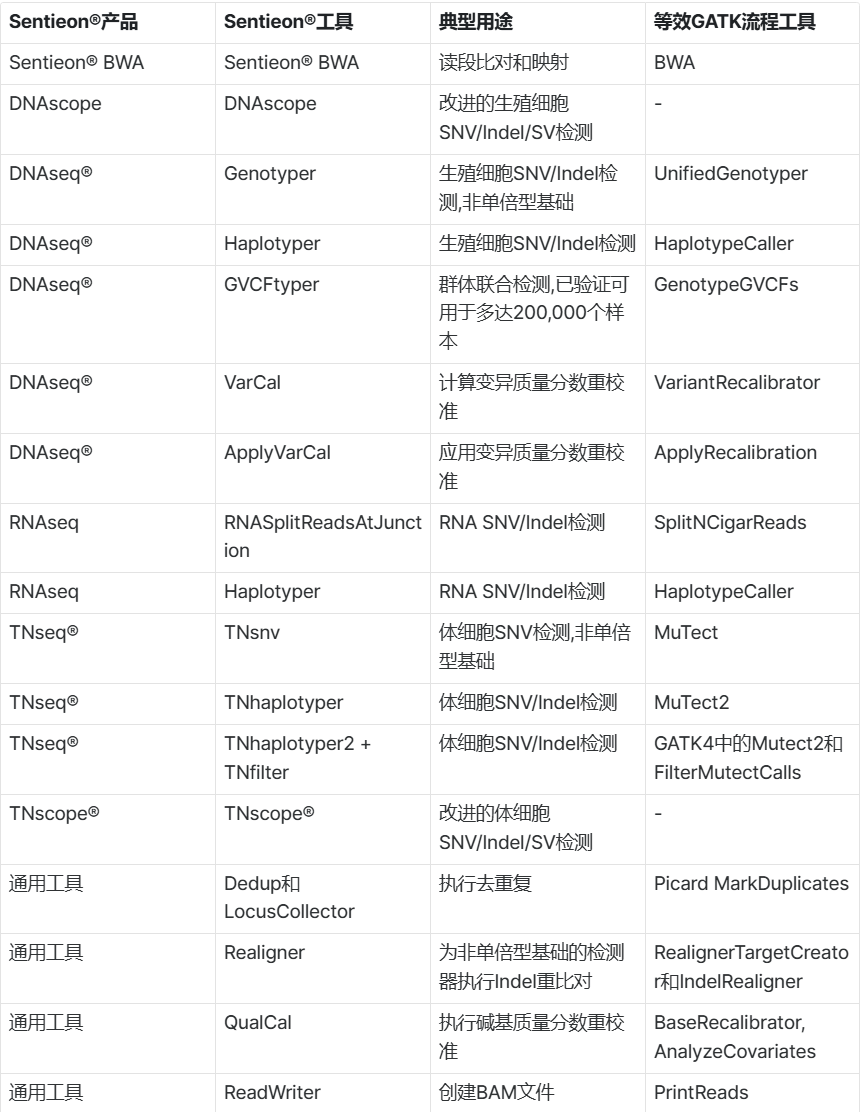
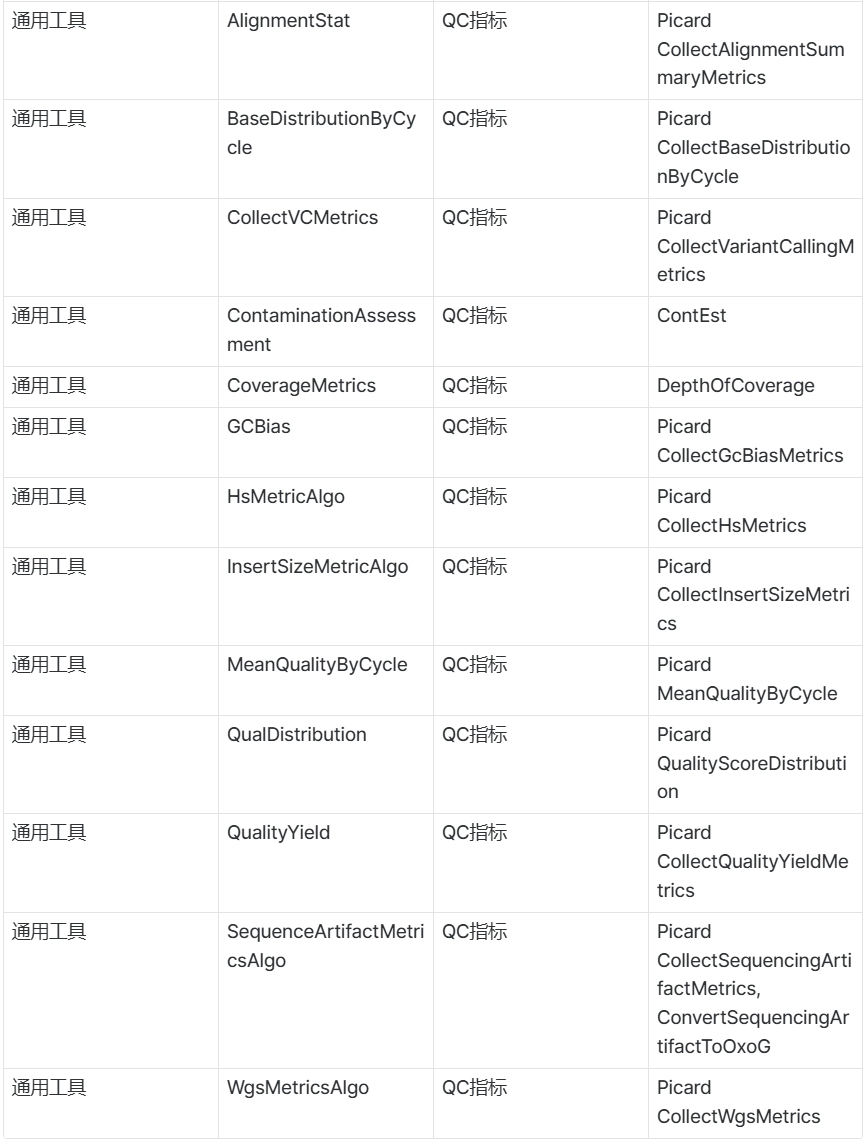
下表展示了不同的Sentieon软件模块及其用途。同时也标注了哪些工具实现了与现有GATK流程工具相同的功能。
3.1 Sentieon模块列表
四、设置许可证
Sentieon®是商用许可类型,用户需要正确设置许可证才能运行软件。
我们提供两种类型的许可证:
•单机评估许可证:此许可证用于在单台机器上评估Sentieon®软件。它允许新用户快速开始使用软件,而无需IT部门的帮助。要使用此许可证,计划运行Sentieon®软件的计算机需要外部Internet访问。
•集群许可证:此许可证用于集群环境。使用此许可证,一个轻量级的浮动许可证服务器进程在集群中的一个节点上运行,通过TCP向所有其他与许可证服务器有网络连接的节点提供许可证。此许可证服务器在集群外围的一个特殊非计算节点上运行,该节点可以通过HTTPS不受限制地访问外部世界,并通过监听集群内需要开放的特定TCP端口向集群中的其余节点提供许可证。
4.1 设置单机评估许可证
要使用单机评估许可证,计算节点需要能够访问Internet。这允许Sentieon®软件验证许可证。
要使用单机评估许可证,请按照以下步骤操作:
1.将许可证文件复制到计算节点。例如,许可证文件LICENSE_FILE.lic现在位于LICENSE_DIR。
2.按如下方式设置环境变量:
export SENTIEON_LICENSE=LICENSE_DIR/LICENSE_FILE.lic4.2 设置许可证服务器
如下面Sentieon软件授权许可拓扑图,许可证服务器需要满足以下条件:
•许可证服务器应该能够访问Internet以执行许可证验证。
•计算节点应该能够通过主机名LICSRVR_HOST访问许可证服务器。
•运行许可证服务器的机器有一个开放的端口供许可证服务监听,并且计算节点可以访问该端口。这里我们假设可用端口是LICSRVR_PORT。
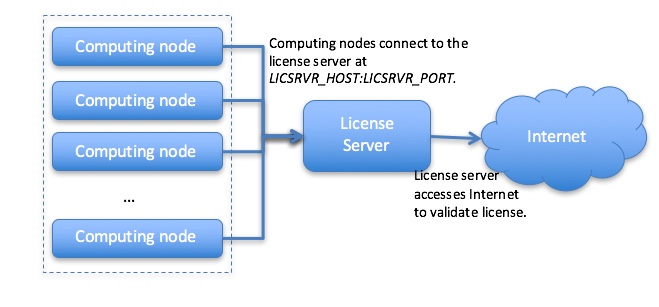
您可能需要IT同事支持来获取LICSRVR_HOST:LICSRVR_PORT,并确认满足上述要求。
注意:如果许可证服务器位于防火墙后面,通过NAT与计算节点分离,则节点可见的许可证服务器主机名/IP可能与其实际主机名/IP不同。如果是这种情况,您需要将许可证服务器绑定到实际IP地址,而计算节点从NAT后的IP地址请求许可证。
按照以下步骤获取许可证文件,设置和测试许可证服务器:
1.将以下信息发送给sentieon以接收许可证文件:
• 指定运行许可证服务的机器的FQDN(Hostname)LICSRVR_HOST。
• 指定的端口LICSRVR_PORT。
2.将收到的许可证文件复制到许可证服务器LICSRVR_HOST。我们假设许可证文件位于LICENSE_PATH/LICENSE_FILE。在许可证服务器上运行以下命令以启动许可证服务器进程:
<SENTIEON_INSTALL_DIR>/bin/sentieon licsrvr --start --log LOG_FILE LICENSE_PATH/LICENSE_FILE3.或者,您可以按照“第五章:设置许可为系统服务”- 将许可证服务器(LICSRVR)作为系统服务运行的说明,将许可证服务器配置并启动为系统守护进程。
4.进入Sentieon®安装目录。在许可证服务器上运行以下命令,确认许可证服务器已启动并正在运行。
<SENTIEON_INSTALL_DIR>/bin/sentieon licclnt ping -s LICSRVR_HOST:LICSRVR_PORT如果命令返回时没有错误消息,则许可证服务器已启动并正在运行。
5.登录到其中一个计算节点,进入Sentieon®安装目录,再次运行上述命令:
<SENTIEON_INSTALL_DIR>/bin/sentieon licclnt ping -s LICSRVR_HOST:LICSRVR_PORT如果命令返回时没有错误消息,则计算节点现在也可以访问许可证服务器。
6.设置以下环境变量,您就可以开始使用了。
export SENTIEON_LICENSE=LICSRVR_HOST:LICSRVR_PORT五、设置许可为系统服务
5.1 使用sysvint将许可证服务器作为系统服务运行
如果您的系统遵循传统的System V init启动脚本,您可以通过运行以下命令作为root用户来设置许可证服务器,使其在系统启动时自动启动:
1.创建并自定义配置文件:配置文件通常为/etc/sysconfig/licsrvr;但在Ubuntu中,配置文件将是/etc/default/licsrvr。以下是配置文件的示例,推荐使用以下设置:
/home/sentieon/release/latest 是最新Sentieon®软件包安装目录的符号链接
/home/sentieon/licsrvr 是运行licsrvr服务的文件夹
/home/sentieon/licsrvr/licsrvr.lic 是Sentieon®许可证文件
licsrvr="/home/sentieon/release/latest/bin/sentieon licsrvr"
licfile="/home/sentieon/licsrvr/licsrvr.lic"
logfile="/home/sentieon/licsrvr/licsrvr.log"2.将许可证服务器启动脚本安装到/etc/init.d目录。启动脚本包含在发布包中。
install -m 0755 $SENTIEON_INSTALL_DIR/doc/licsrvr.sh /etc/init.d/licsrvr3.安装并启用服务。根据您的系统,您将运行不同的命令:
•如果您的系统安装了Linux Standard Base Core Specifications,执行系统init脚本安装脚本:
/usr/lib/lsb/install_initd /etc/init.d/licsrvr•如果您的系统没有安装lsb.conformance包,使用chkconfig命令启用服务:
chkconfig --add licsrvr
chkconfig licsrvr on•对于Ubuntu和Debian系统,如果您没有lsb/install_initd二进制文件且选择不安装lsb-core包,请使用update-rc.d命令安装并启用服务:
update-rc.d licsrvr defaults
update-rc.d licsrvr enable4.您可以使用service命令来启动/停止/重启/检查服务的状态:
service licsrvr {start|stop|restart|status}5.2 使用systemd将许可证服务器作为系统服务运行
您可以使用操作系统的systemd系统和服务功能来设置许可证服务器,使其在系统中自动启动。要做到这一点,请以root用户身份运行以下命令:
1.如果您使用doc文件夹中的licsrvr.service许可证服务器启动脚本,您需要创建脚本所需的必要文件,包括使用sentieon用户名:
•/home/sentieon/release/latest 是最新Sentieon®软件包安装目录的符号链接
•/home/sentieon/licsrvr 是运行licsrvr服务的文件夹
•/home/sentieon/licsrvr/licsrvr.lic 是Sentieon®许可证文件
或者,您可以编辑许可证服务器启动脚本以指向您的特定用户名和/或位置信息。
2.将许可证服务器启动脚本安装到/etc/systemd/system目录:
install -m 0644 $SENTIEON_INSTALL_DIR/doc/licsrvr.service /etc/systemd/system3.运行以下命令以启用许可证服务器在计算机启动时自动启动:
systemctl enable licsrvr.service4.您可以使用systemctl命令手动启动和停止服务:
systemctl start licsrvr.service
systemctl stop licsrvr.service六、常见问题
6.1 Sentieon安装时出现jemalloc error
在安装运行Sentieon过程中可能会出现以下错误:
ERROR: ld.so: object '/usr/lib64/libjemalloc.so.2' from LD_PRELOAD cannot be preloaded: ignored. Failed to contact the license server at 10.10.10.1:8990这个错误与jemalloc有关。Jemalloc是一个内存分配器,针对多线程方案中的高内存分配性能和更少的内存碎片进行了优化。Sentieon建议使用jemalloc来改善Sentieon应用程序中的内存管理和整体性能,尤其是Sentieon bwa-mem。
解决方案:
1.安装jemalloc:
对于不同的操作系统,安装命令如下:
•RHEL/CentOS 8.x:
yum install epel-release
yum install jemalloc默认安装在 /usr/lib64/libjemalloc.so.2
•RHEL/CentOS 7.x:
yum install epel-release
yum install jemalloc默认安装在 /usr/lib64/libjemalloc.so.1
•Ubuntu 20.04:
apt update
apt install libjemalloc2默认安装在 /usr/lib/x86_64-linux-gnu/libjemalloc.so.2
•Ubuntu 18.04:
apt update
apt install libjemalloc1默认安装在 /usr/lib/x86_64-linux-gnu/libjemalloc.so.1
2.对于没有预构建软件包的其他系统,请参考jemalloc GitHub页面 (https://github.com/jemalloc/jemalloc) 以获取有关如何构建和安装jemalloc的更多信息。
3.使用环境变量在运行时加载jemalloc库到Sentieon中:
例如,在CentOS 8.x系统上,在运行Sentieon工具之前,您可以使用以下命令设置环境变量:
export LD_PRELOAD=/usr/lib64/libjemalloc.so.26.2 准备参考文件以供使用
如果您的参考FASTA文件尚未预处理,使得软件无法使用中指定的数据,您需要按照以下步骤对其进行处理:
1.使用BWA生成BWA索引。这将创建".fasta.amb"、".fasta.ann"、".fasta.bwt"、".fasta.pac"和".fasta.sa"文件。
sentieon bwa index reference.fasta2.使用samtools生成FASTA文件索引。这将创建".fasta.fai"文件。
samtools faidx reference.fasta3.使用Picard生成序列字典。这将创建".dict"文件。
java -jar picard.jar CreateSequenceDictionary REFERENCE=reference.fasta \
OUTPUT=reference.dict6.3 准备RefSeq文件以供使用
RefSeq文件用于将CoverageMetrics算法的结果聚合到基因级别。为了使用从UCSC基因组浏览器下载的RefSeq文件,需要按染色体和位点对它们进行排序。要执行排序,您需要执行以下步骤:
1.从文件中删除头部
grep -v "^#" FILE.refSeq > FILE.refSeq.headerless
grep -e "^#" FILE.refSeq > FILE.refSeq.header2.首先使用unix sort对位点进行排序。
sort -k 5 -n FILE.refSeq.headerless > FILE.refSeq.presorted3.使用GATK sortByRef.pl和FASTA索引fai按染色体排序。
perl sortByRef.pl --k 3 FILE.refSeq.presorted FASTA.fai --tmp ~/tmp \
> FILE_sorted_headerless.refSeq4.将头部放回文件。
cat FILE.refSeq.header > FILE_sorted.refSeq
cat FILE_sorted_headerless.refSeq >> FILE_sorted.refSeq6.4 许可证消息:No more license available for Sentieon…
当您请求运行Sentieon®软件的线程数超过您的许可证当前允许的数量时,会产生此消息。这种情况发生是因为您同时运行的命令集体请求的线程数超过了您的许可证支持的核心数。可使用以下命令查看授权剩余的线程数:
sentieon licclnt query -s LICSRVR_HOST:LICSRVR_PORT klibSentieon®命令在等待空闲许可证时将处于空闲状态,但命令不会失败。
6.5 Driver失败并显示错误:Readgroup XX is present in multiple BAM files with different attributes
当您输入两个不同的BAM文件,其中包含具有相同ID但不同属性的读组时,会产生此错误。例如,在TNseq®和TNscope®中,肿瘤和正常样本BAM文件的RG ID都是"1"。
在使用BAM文件之前,您需要编辑它们以使RG ID唯一,例如通过将SM名称添加到RG ID中。您可以查看此问题的解决方法示例。
或者,您可以使用samtools addreplacerg功能来修改输入BAM文件的RG ID并使其唯一:
# 添加新的RG并修改BAM文件中的所有读段
RGtag=$(samtools view -H $INPUT_BAM|grep ^@RG|sed "s|ID:$ORIG_RGID|ID:$NEW_RGID|g")
samtools addreplacerg -r "$RGtag" -o $TMP_BAM $INPUT_BAM
# 重新设置BAM头以删除不再使用的原始RG
samtools view -H $TMP_BAM|grep -v "^@RG.*$ORIGINAL_RGID" \
|samtools reheader - $TMP_BAM > $OUTPUT_BAM
rm $TMP_BAM6.6 Driver报告警告:none of the QualCal tables is applicable to the input BAM files
此警告意味着重校准表输入文件中的信息都不能应用于输入的BAM文件,这可能是由于使用了与BAM文件不对应的重校准表。
当QualCal的输入BAM文件在RG字段中没有正确的字段时,可能会产生此警告。例如,如果RG的PL标签设置为ILLUMINA以外的其他值,就可能发生这种情况;在这种情况下,您需要修改BAM头以包含/修改缺失/不正确的字段,为此您可以使用samtools reheader功能。
6.7 使用从BAM文件创建的FASTQ文件时,BWA使用异常大量的内存
当您使用通过转换已排序的BAM文件创建的FASTQ文件时,可能会发生所有未映射的读段都被分组到FASTQ输入的末尾。在这种情况下,BWA可能在对齐结束时使用异常大量的内存,因为映射质量差或无法映射的读段需要额外的内存。
为了减少异常内存使用,您应该首先重新排序bam文件,以确保未映射的读段不会被分组在一起。您可以使用samtools来做到这一点:
samtools sort -n -@ 32 input.bam | samtools fastq -@ 32 \
-s >(gzip -c > single.fastq.gz) -0 >(gzip -c > unpaired.fastq.gz) \
-1 >(gzip -c > output_1.fastq.gz) -2 >(gzip -c > output_2.fastq.gz) -6.8 KPNS - 已知问题无解决方案
6.8.1 不支持未使用bgzip压缩的gzip压缩vcf文件
普通的gzip文件不允许随机或索引访问其中包含的信息,只有使用bgzip压缩的文件才可索引。
因此,Sentieon®软件不支持gzip压缩的VCF文件作为输入。
要使用这些文件,您需要使用gunzip解压缩它们,然后不压缩使用或使用bgzip重新压缩。
或者,您可以使用util vcfconvert重新压缩和索引文件。
sentieon util vcfconvert INPUT.vcf.gz OUTPUT.vcf.gz6.8.2 不支持gzip压缩的fasta文件
目前,软件不支持gzip压缩的FASTA文件作为输入。您需要在使用之前gunzip这些文件。
6.8.3 FASTQ文件需要采用SANGER质量格式
如果您的FASTQ文件是在1.8之前使用Illumina™测序技术编码的,读取质量分数将不是SANGER格式,这可能会产生意外结果。Sentieon®基因组学软件不会检测到您正在使用不支持的格式。
6.8.4 Driver失败并显示错误:ImportError: No module named argparse
当在Python版本为2.6.x且不存在argparse模块的环境中运行tnhapfilter时,会产生此错误。您需要为您的Python安装安装argparse模块;您可以通过运行pip install argparse或您使用的其他包管理器来完成此操作。
6.9 常见使用问题
以下是常见问题的症状列表以及解决方案。
6.9.1 Driver或Util失败并显示错误:can not open file (xxx) in mode(r), Too many open files
此错误的根本原因是系统中同时打开的文件限制设置得不够高。
您可以通过设置系统ulimit -n来解决此错误。在基于Linux的系统中:
检查系统中最大打开文件数的限制,运行以下命令:
ulimit -n通过以root身份编辑文件/etc/security/limits.conf来设置更高的限制,并添加以下2行:
* soft nofile 16384
* hard nofile 16384如果您的系统运行的是Ubuntu,您还需要将此行添加到您的shell配置文件~/.bashrc中:
ulimit -n 16384您需要注销系统并重新登录才能使更改生效。登录后,通过运行以下命令检查更改是否正确应用:
ulimit -n命令应返回16384。
6.9.2 Driver失败并显示错误:Contig XXX from vcf/bam is not present in the reference, or error Contig XXX has different size in vcf/bam than in the reference
此错误的根本原因是输入的VCF或BAM文件与参考fasta文件不兼容。文件中的contig不存在于参考中,或contig的大小不同。这很可能是由于使用了与不同参考处理的VCF或BAM文件造成的。
6.9.3 Driver报告警告:Contigs in the vcf file XXX do not match any contigs in the reference
此警告的根本原因是输入的VCF文件与参考fasta文件不兼容,文件中的contig不存在于参考中。这很可能是由于使用了来自不同参考的VCF文件造成的。
6.9.4 BWA失败并显示错误:Killed
当BWA从操作系统接收到SIGKILL信号时,会产生此错误。如果系统可用内存不足,SIGKILL可能是由内核的内存不足(OOM)管理器发送的。您可以检查系统上的内核日志以确认SIGKILL信号是否由OOM管理器发送。
要解决此错误,您可以使用bwt_max_mem环境变量减少BWA的内存使用。
原创声明:本文系作者授权腾讯云开发者社区发表,未经许可,不得转载。
如有侵权,请联系 cloudcommunity@tencent.com 删除。
原创声明:本文系作者授权腾讯云开发者社区发表,未经许可,不得转载。
如有侵权,请联系 cloudcommunity@tencent.com 删除。

