课前准备---空间转录组数据分析之分子niche
原创课前准备---空间转录组数据分析之分子niche
原创
追风少年i
发布于 2024-07-17 09:12:48
发布于 2024-07-17 09:12:48
作者,Evil Genius
最近有人问我为什么一直要设置付费呢?而且还费用这么高,这个问题,其实都是血泪史啊。
去年(2023年)刚开始上单细胞的时候,就是不设置任何限制,公开分享,结果是什么呢?遭受一些医生的诋毁,网暴,各种举报,等等等等。
而且不得不说,我们都有劣根性,太容易得到,都不知道珍惜,当初一分享,立马烂大街,不仅把行业搞乱了,自己也没落下什么好名声,不会有人说这是谁谁谁分享的,大家要珍惜这些话的。就像董宇辉说的,怎么可能有人完全理解你的苦,然后因此懂得你的难呢?不会有的。
所以不得不设置付费,而且确实回归了正常,真正有需要的人,付费完了一般会加上微信,多聊聊课题什么的,一切都恢复了平静,都开始正常的科研工作了。而且就算有人买了代码去倒卖,没人指导也没几个人能跑通,更何况个性化分析需要很多设置和注意事项等等,很多内容不是简单的几行代码都可以解决的。就算有黑粉,也是极个别现象了。
至于费用高不高,就看自己有没有需要了,如果是课题组自己测单细胞空间需要分析的,那这费用跟白送没区别。公司做售后的费用大家心里都应该有数。设置付费,挡掉了所有别有用心的人。
这一篇我们要总结分子niche,
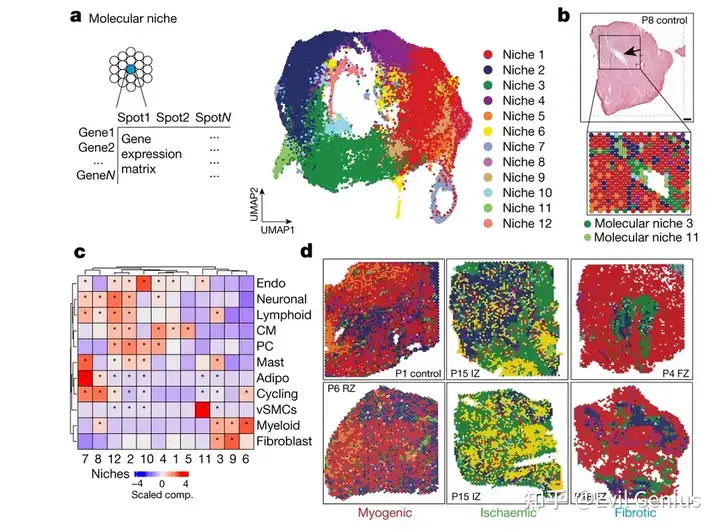
我们同样需要实现的目标如下:
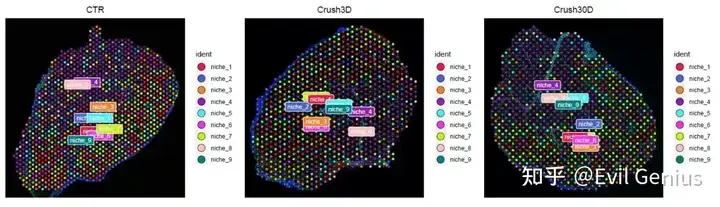
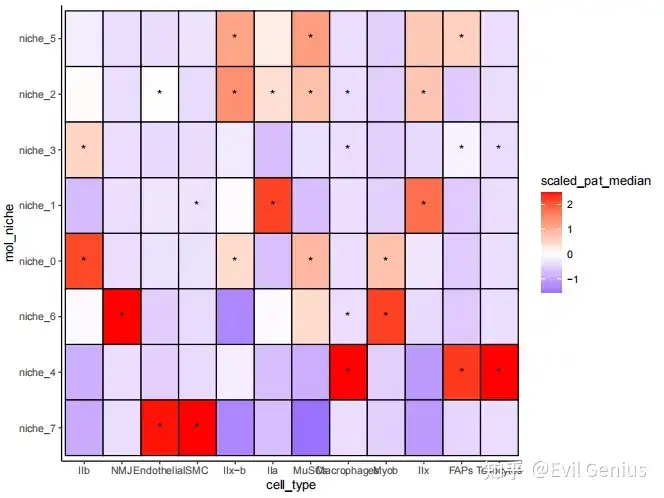
其中的生物学意义,我们课上会讲解。
完整代码如下
#! usr/R
####zhaoyunfei
####20240716
suppressMessages({
library(Seurat)
library(compositions)
library(tidyverse)
library(clustree)
library(uwot)
library(cluster)
library(RColorBrewer)
})
adata = readRDS('Muscle.spatial.rds')
cluster_info <- as.data.frame(adata$seurat_clusters)
colnames(cluster_info) = 'mol_niche'
cluster_info$row_id = rownames(cluster_info)
integrated_compositions <- t(adata@assays$predictions@data)
integrated_compositions = integrated_compositions[,-which(colnames(integrated_compositions) %in% c("max",'MTJ','RegMyon'))]
niche_summary_pat <- integrated_compositions %>% as.data.frame() %>% rownames_to_column("row_id") %>% pivot_longer(-row_id,values_to = "ct_prop", names_to = "cell_type") %>% left_join(cluster_info) %>% mutate(orig.ident = strsplit(row_id, "[..]") %>% map_chr(., ~ .x[1])) %>% group_by(orig.ident, mol_niche, cell_type) %>% summarize(median_ct_prop = median(ct_prop))
niche_summary <- niche_summary_pat %>% ungroup() %>% group_by(mol_niche, cell_type) %>% summarise(patient_median_ct_prop = median(median_ct_prop))
niche_summary$mol_niche = paste('niche',niche_summary$mol_niche,sep = '_')
niche_summary_pat %>% ungroup() %>% group_by(mol_niche, cell_type) %>% summarise(patient_median_ct_prop = median(median_ct_prop))
niche_summary_mat <- niche_summary %>% pivot_wider(values_from = patient_median_ct_prop, names_from = cell_type, values_fill = 0) %>% column_to_rownames("mol_niche") %>% as.matrix()
niche_order <- hclust(dist(niche_summary_mat))
niche_order <- niche_order$labels[niche_order$order]
ct_order <- hclust(dist(t(niche_summary_mat)))
ct_order <- ct_order$labels[ct_order$order]
# Find characteristic cell types of each niche
# We have per patient the proportion of each cell-type in each niche
run_wilcox_up <- function(prop_data) {
prop_data_group <- prop_data[["mol_niche"]] %>% unique() %>% set_names()
map(prop_data_group, function(g) {
test_data <- prop_data %>% mutate(test_group = ifelse(mol_niche == g,"target", "rest")) %>% mutate(test_group = factor(test_group,levels = c("target", "rest")))
wilcox.test(median_ct_prop ~ test_group, data = test_data, alternative = "greater") %>% broom::tidy()
}) %>% enframe("mol_niche") %>% unnest()
}
wilcoxon_res <- niche_summary_pat %>%
ungroup() %>%
group_by(cell_type) %>%
nest() %>%
mutate(wres = map(data, run_wilcox_up)) %>%
dplyr::select(wres) %>%
unnest() %>%
ungroup() %>%
mutate(p_corr = p.adjust(p.value))
wilcoxon_res <- wilcoxon_res %>% mutate(significant = ifelse(p_corr <= 0.15, "*", ""))
wilcoxon_res$mol_niche = paste('niche',wilcoxon_res$mol_niche,sep = '_')
####head(niche_summary_pat)
mean_ct_prop_plt <- niche_summary %>%
left_join(wilcoxon_res, by = c("mol_niche", "cell_type")) %>%
mutate(cell_type = factor(cell_type, levels = ct_order),
mol_niche = factor(mol_niche, levels = niche_order)) %>%
ungroup() %>%
group_by(cell_type) %>%
mutate(scaled_pat_median = (patient_median_ct_prop - mean(patient_median_ct_prop))/sd(patient_median_ct_prop)) %>%
ungroup() %>%
ggplot(aes(x = cell_type, y = mol_niche, fill = scaled_pat_median)) +
geom_tile(color="black",size=0.5) +
geom_text(aes(label = significant)) +
theme(axis.text.x = element_text(angle = 180, hjust = 1, vjust = 0.5, size = 12),
legend.position = "bottom",
plot.margin = unit(c(0, 0, 0, 0), "cm"),
axis.text.y = element_text(size=12)) +
scale_fill_gradient2(high = "red",mid = 'white', low = "blue") + theme_classic()
# Finally describe the proportions of those niches in all the data
cluster_counts <- cluster_info %>%
dplyr::select_at(c("row_id", "mol_niche")) %>%
group_by(mol_niche) %>%
summarise(nspots = length(mol_niche)) %>%
mutate(prop_spots = nspots/sum(nspots))
write_csv(cluster_counts, file = "./mol_niche_prop_summary.csv")
barplts <- cluster_counts %>%
mutate(ct_niche = factor(mol_niche, levels = niche_order)) %>%
ggplot(aes(y = mol_niche, x = prop_spots)) +
geom_bar(stat = "identity") +
theme_classic() + ylab("") +
theme(axis.text.y = element_blank(),
plot.margin = unit(c(0, 0, 0, 0), "cm"),
axis.text.x = element_text(size=12))
niche_summary_plt <- cowplot::plot_grid(mean_ct_prop_plt, barplts, align = "hv", axis = "tb")
pdf('characteristic_mol_niches.pdf',width = 15,height = 5.5)
plot(niche_summary_plt)
dev.off()
adata = adata[,cluster_info$row_id]
Idents(adata) = cluster_info$ct_niche
sample_cols = unique(c(brewer.pal(8,"Dark2"),brewer.pal(9,"Set1"),brewer.pal(8,"Set2"),brewer.pal(12,"Set3")))
defined_cols = c('#e6194b', '#4363d8', '#f58231', '#911eb4', '#46f0f0', '#f032e6', '#bcf60c', '#fabebe', '#008080', '#e6beff', '#9a6324', '#fffac8', '#800000', '#aaffc3', '#808000', '#ffd8b1', '#000075', '#808080', sample_cols)
p = SpatialDimPlot(adata, label = TRUE, label.size = 3)
for (i in 1:length(p)){p[[i]] = p[[i]] + guides(fill=guide_legend(override.aes = list(size=5))) + scale_fill_manual(values = defined_cols) }
pdf('niche.spatial.pdf',width = 15)
print(p)
dev.off()生活很好,有你更好
原创声明:本文系作者授权腾讯云开发者社区发表,未经许可,不得转载。
如有侵权,请联系 cloudcommunity@tencent.com 删除。
原创声明:本文系作者授权腾讯云开发者社区发表,未经许可,不得转载。
如有侵权,请联系 cloudcommunity@tencent.com 删除。
评论
登录后参与评论
推荐阅读
目录

