课前准备----细胞分子邻域矩阵的获得与运用
原创课前准备----细胞分子邻域矩阵的获得与运用
原创
追风少年i
发布于 2024-07-25 15:30:03
发布于 2024-07-25 15:30:03
作者,Evil Genius
最近可能是放假的原因,所以硕博士科研的强度低了一点,开始有的人去玩一玩,或者做一些自己感兴趣的内容,或者报考驾照等等,我当初读研的时候,暑假放20天,不知道大家放多少天,,这段日子,其中就有一些人选择了学习生物信息。
最近有人问我报班学习的事,之前聊过,看自己需求,当然了,培训班都有费用,因为一般培训班上课都至少2个月,那么费用自然几千到1万不等,而且各种培训班良莠不齐,大家要能够识别。
其中有很多人问没法报账,自费太贵怎么办,这让我想起了自己当初考研报培训班的事,我本科学的生物科学,考研想报考工科,但是工科需要学习数学,高数、现代、概率论,本科阶段就学过高数,而且都还给老师了,当然可以选择自己自学,不过肯定非常吃力而且一个人琢磨很容易放弃,当时报班相比于现在算便宜,1000,但是本科的时候大家都没钱,都伸手给家里要钱,当时没办法,大三上半学期结束跑到超市做了2个月收银员,清晰的记得2个月挣了4000出头,花了2000报了英语和数学。
学习技能绝对是值得肯定的,最重要的是有一群和自己一样的人,在一起学习和交流,效果当然比一个人琢磨好得多。
今天我们分享如何获得细胞的分子邻域矩阵。
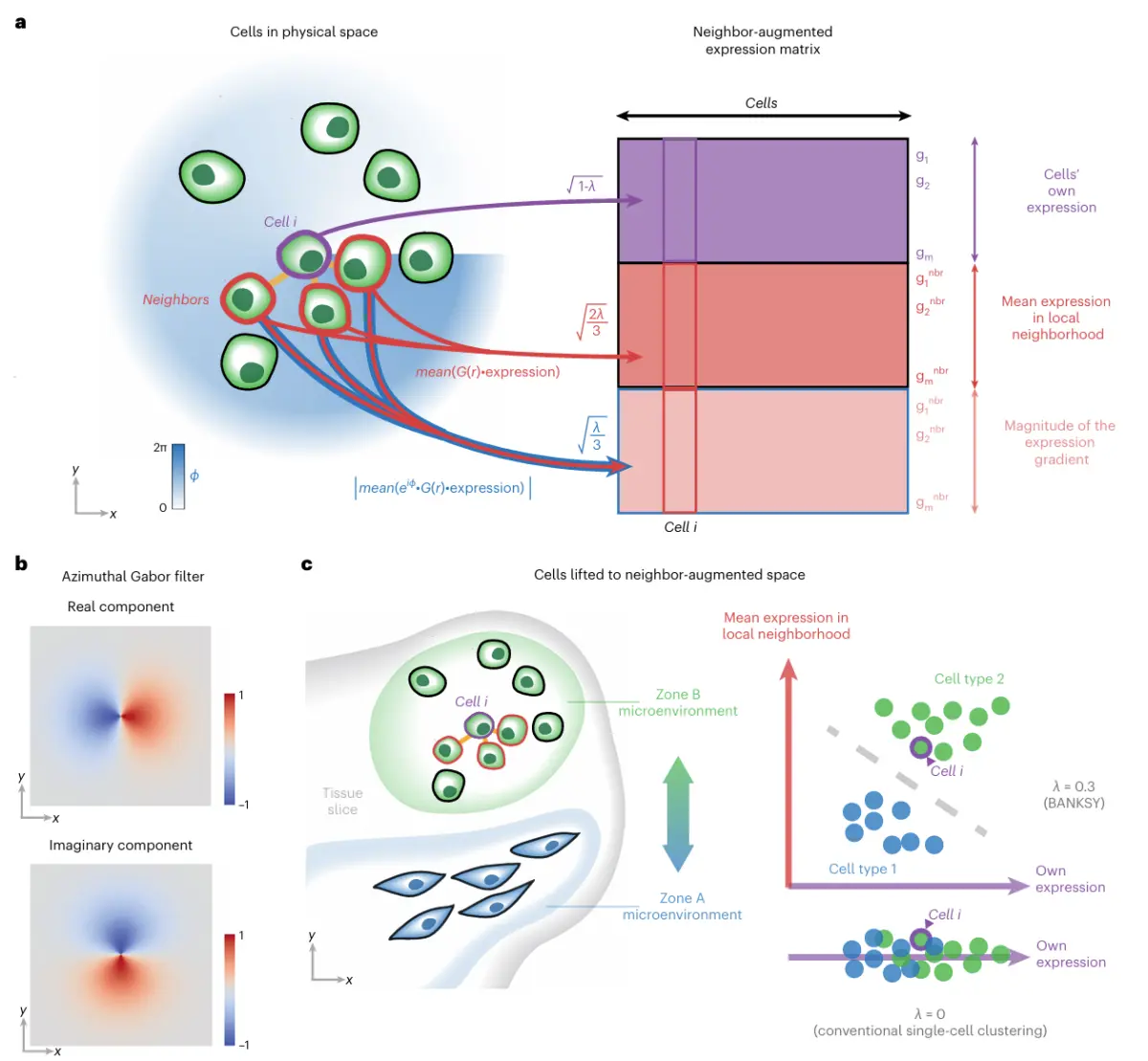
每个细胞/spot都处在一定的空间微环境中,这个微环境包括的分子的表达情况和细胞的分布情况,以10X为例,我们需要知道每个spot周围6个spot的基因表达情况和细胞分布特点。对于高精度平台,例如CODEX,那么我们需要知道每个细胞周围最近的10(20)个细胞的细胞类型和表达特点,进一步为我们提供不同条件下,细胞类型邻域的发生和变化。
当然了,随着分析的深入,自然需要更加精细化的课题设计和数据整理,对大家的要求自然也高一点。
我们的目标是第一步,获取每个spot/细胞的临近spot/细胞坐标信息。
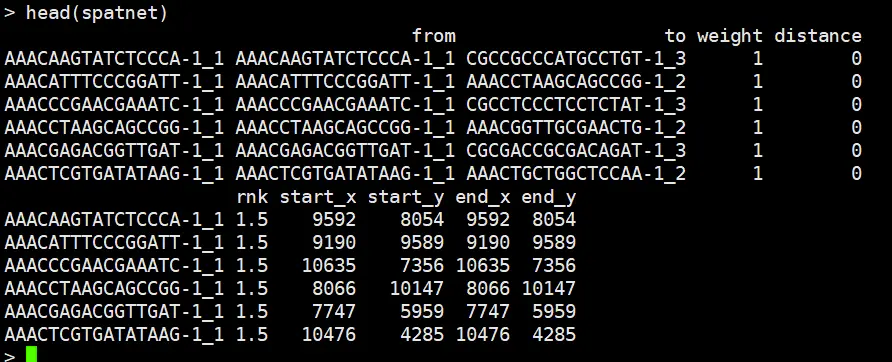
第二步构建细胞的分子邻域矩阵

第三步就是分析分子邻域矩阵的表达特点和差异,这部分随着课题的不同,内容不同,需要大家自己动手分析了。其中最直观的就是邻域通讯的差异。
完整代码如下,适用于各种空间平台。代码有需要的拿走,绝对不能随意分享了。
#! usr/R
####zhaoyunfei
####20240728
library(Seurat)
library(dplyr)
library(dbscan)
library(ggplot2)
library(dplyr)
st.object = readRDS('/home/samples/DB/Spatial/cellniche/Muscle.spatial.rds')
st.object$pixel_x = st.object@images$CTR@coordinates$imagerow
st.object$pixel_y = st.object@images$CTR@coordinates$imagecol
st.object$sample = st.object$orig.ident
####st.object = st.object[VariableFeatures(st.object),]
xys = setNames(st.object@meta.data[, c("pixel_x", "pixel_y", "sample")], c("x", "y", "sample"))
platforms = 'Visium'
spotnames <- rownames(xys)
library(dplyr)
names(spotnames) <- c(1:nrow(xys)) %>% paste0()
et spot distances
sdist = 200
###10X空间转录组临近的6个spot
nNeighbours = 6
maxdist = 200
knn_spatial <- dbscan::kNN(x = xys[, c("x", "y")] %>% as.matrix(), k = nNeighbours)
knn_spatial.norm <- data.frame(from = rep(1:nrow(knn_spatial$id), nNeighbours),
to = as.vector(knn_spatial$id),
weight = 1/(1 + as.vector(knn_spatial$dist)),
distance = as.vector(knn_spatial$dist))
minK = 4
# create network for coordinates
spatnet <- knn_spatial.norm
spatnet$from <- spotnames[spatnet$from]
spatnet$to <- spotnames[spatnet$to]
spatnet <- spatnet %>% group_by(from) %>% mutate(rnk = rank(distance)) %>% ungroup()
spatnet = subset(spatnet, distance <= maxdist | rnk <= minK)
# Add coordinates
spatnet <- cbind(spatnet, setNames(xys[spatnet$from, 1:2], paste0("start_", c("x", "y"))))
spatnet <- cbind(spatnet, setNames(xys[spatnet$to, 1:2], paste0("end_", c("x", "y"))))
####空矩阵
neighbor_mol = matrix(ncol = length(unique(spatnet$from)),nrow = length(rownames(st.object)))
for (i in 1:length(colnames(st.object))){
su = spatnet[which(spatnet$from == colnames(st.object)[i]),]
neighbor_mol[,i] = rowSums(st.object@assays$Spatial@counts[,su$to])
}
colnames(neighbor_mol) = colnames(st.object)
rownames(neighbor_mol) = rownames(st.object)
write.csv(neighbor_mol,file = 'neighbor_mol.csv',quote =F)生活很好,有你更好
原创声明:本文系作者授权腾讯云开发者社区发表,未经许可,不得转载。
如有侵权,请联系 cloudcommunity@tencent.com 删除。
原创声明:本文系作者授权腾讯云开发者社区发表,未经许可,不得转载。
如有侵权,请联系 cloudcommunity@tencent.com 删除。
评论
登录后参与评论
推荐阅读
目录
