Mfuzz/ClusterGVis包时间(规律变化数据即可)序列分析学习和整理
原创Mfuzz/ClusterGVis包时间(规律变化数据即可)序列分析学习和整理
原创
Mfuzz是一个用于时间序列/状态空间/规律变化数据聚类分析的 R 包,适用于生物信息学中的规律变化数据分析。以下是 Mfuzz 包的主要作用:
- 模糊聚类分析:Mfuzz 使用模糊 C 均值(Fuzzy C-Means)算法对数据进行聚类。这种方法允许数据点属于多个聚类,这对于生物学数据中常见的噪声和复杂性特别有用。
- 处理噪声和不确定性:与传统的硬聚类不同,Mfuzz 可以处理数据中的噪声和不确定性,通过为每个数据点分配不同聚类的隶属度,反映出其对多个聚类归属可能性。
- 分析时间序列数据:Mfuzz 特别适合时间序列数据(规律变化数据即可!)分析,如不同时间点或不同实验条件下的基因表达变化。它能够识别出数据中的趋势和模式,帮助研究者理解基因或样本在不同条件下的响应。
- 可视化工具:Mfuzz 提供了可视化功能,可以展示聚类结果、时间序列模式和聚类隶属度,有助于深入分析和解释聚类结果。
- 处理缺失数据:Mfuzz 能够处理数据集中存在的缺失值,使其在实际应用中更加灵活和鲁棒。
ClusterGVis设计用于对这类规律变化的数据结果进行可视化和解释,得到更加精美的图~
分析流程
1、导入
GSE142588:华蟾素/肝癌/不同时间点
rm(list = ls())
library(Mfuzz)
library(limma)
library(clusterProfiler)
library(ggplot2)
library(ggstatsplot)
library(tidyverse)
load("GSE142588.Rdata")2、数据预处理
dat <- log2(edgeR::cpm(exp)+1)
dat[1:4,1:4]
# GSM4232492 GSM4232493 GSM4232494 GSM4232495
# DDX11L1 0.3879564 0.335058 0.3866041 0.3941932
# WASH7P 4.0736755 4.082968 4.0682735 2.7459054
# MIR6859-1 0.2698618 0.335058 0.2062135 0.2105183
# WASH9P 1.2560585 1.104527 1.2213747 1.3023497
dim(dat)
# [1] 19524 16
table(Group)
# con H12 H24
# 4 6 6
identical(rownames(clinical),colnames(exp))
# [1] TRUE
# 修改表达矩阵的列名
colnames(dat) <- Group
colnames(dat)
# [1] "con" "con" "con" "H12" "H12" "H12" "H24" "H24" "H24" "con" "H12" "H12" "H12" "H24" "H24"
# [16] "H24"
library(limma)
# limma::avereps:这是来自 limma 包的函数,avereps 用于对重复数据进行平均值计算。
# avereps 会根据指定的 ID 进行分组,并对相同 ID 的数据取平均值
avereps_df <- t(limma::avereps(t(dat) , ID = colnames(dat)))##对相同时间序列的表达值取平均
avereps_df[1:3,1:3]
# con H12 H24
# DDX11L1 0.3882151 0.2560873 0.1818046
# WASH7P 4.0862767 2.7356243 3.1765777
# MIR6859-1 0.2544874 0.1306503 0.1723505
colnames(avereps_df)
# [1] "con" "H12" "H24"
save(avereps_df,file = 'avereps_df.Rdata')最后可以看到,得到了3组数据,分别是对照组(0h), H12(12h)和H24(24h)。
3、正式分析
load(file = 'avereps_df.Rdata')
avereps_df[1:3,1:3]
colnames(avereps_df)
## 过滤----
# 去除表达量太低或者在不同时间点间变化太小的基因等步骤
# Mfuzz聚类时要求是一个ExpressionSet类型的对象,所以需要先用表达量构建这样一个对象。
eset <- new("ExpressionSet",exprs = avereps_df)
# 根据处理NA值或者根据标准差去除样本间差异太小的基因
eset <- filter.NA(eset, thres = 0.25)
eset <- fill.NA(eset, mode = 'mean')
eset <- filter.std(eset,min.std=0)
# 0 genes excluded.
eset
# ExpressionSet (storageMode: lockedEnvironment)
# assayData: 19524 features, 3 samples
# element names: exprs
# protocolData: none
# phenoData: none
# featureData: none
# experimentData: use 'experimentData(object)'
# Annotation:
## 标准化----
# 聚类时需要用一个数值来表征不同基因间的距离,Mfuzz中采用的是欧式距离,
# 由于普通欧式距离的定义没有考虑不同维度间量纲的不同,所以需要先进行标准化
eset <- standardise(eset)
## FCM聚类参数设定----
# Mfuzz中的聚类算法需要提供两个参数,
# 第一个参数为希望最终得到的聚类的个数,这个参数由分析者直接指定
# 第二个参数称之为fuzzifier值,用小写字母m表示,可以通过函数评估一个最佳取值
c <- 9
m <- mestimate(eset) # 评估出最佳的m值
clusters <- mfuzz(eset, c = c, m = m) # 聚类
# ### Soft clustering and visualisation
# cl <- mfuzz(yeastF,c=20,m=1.25)
# acore.list <- acore(yeastF,cl=cl,min.acore=0.7)
# }
# 开发者的示例参数
## 查看结果----
# 在clusters这个对象中就保存了聚类的完整结果,对于这个对象的常见操作如下
clusters$size # 查看每个cluster中的基因个数
# [1] 1641 1476 896 3097 2451 2035 2208 3606 2114
head(clusters$cluster[clusters$cluster == 1]) # 提取某个cluster下的基因
# LINC01128 NOC2L C1orf159 C1QTNF12 NADK SKI
# 1 1 1 1 1 1
## 聚类核心
#隶属度值也可以表示向量之间的相似性。
eset
## 提取形成软聚类簇α核心的基因
clusters_genes <- acore(eset,cl,min.acore=0.5)
head(clusters_genes[[1]])
# NAME MEM.SHIP
# C1orf159 C1orf159 0.5048771
# TAS1R1 TAS1R1 0.7383337
# DDI2 DDI2 0.7399099
# CAMK2N1 CAMK2N1 0.6766280
# ECE1 ECE1 0.7718505
# HTR1D HTR1D 0.5022669
table(clusters$cluster)
# 1 2 3 4 5 6 7 8 9
# 1641 1476 896 3097 2451 2035 2208 3606 2114
unlist(lapply(clusters_genes, nrow))
# [1] 335 273 135 953 641 528 529 1416 602在实际分析数据的过程中,最关键的步骤应当是C和M值的选择。开发者也在文档中承认这两个值需要花很多功夫去确定,这也是这个R包中的缺点,但是做科研嘛~ 可不能怕麻烦的hhhh

那么简单介绍一下m和c值
具体内容一定要看原始文档,毕竟笔者也只是按照个人理解结合一些工具做的简单解释!
聚类参数的确定
FCM 参数 m 是一个关键参数,它决定了聚类分析对噪声的敏感性。当 m 接近 1 时,聚类会变得“hard”,即 FCM 算法变得与 K-means 聚类相似,此时隶属度(Membership value)值要么是 1,要么是 0,所有基因在计算聚类中心时被平等对待。 随着参数 m 的增大,低隶属度的基因对聚类中心的影响会减弱。含有大量噪声的基因表达向量通常具有较低的隶属度,因为这些基因无法很好地被单个聚类表示,而是被分配给多个聚类。 因此,选择 FCM 参数 m 可以控制噪声对聚类过程的影响。当 m 趋向于无穷大时,分区矩阵变得均匀,即所有基因几乎均等地分配到所有聚类中。通过观察不同 m 值下的聚类结果,可以深入了解数据结构。
基线聚类的构建
"hard"聚类算法(如 K-means 或 SOMs)一个主要问题是,无论数据如何,这些算法都会将对象分配到聚类中。即使数据是随机的,也会形成明显的聚类。通过"soft"聚类可以克服硬聚类中的这种缺陷(笔者认为应是减弱)。
聚类数量的确定
在选择 FCM 参数 m 之后,需要确定聚类的数量 c。FCM 算法中的聚类数 c 被逐渐增加,并对聚类结果进行了检查。观察到,随着 c 的增加,基因的隶属值在各个聚类之间更加分散,生成的聚类也变得更加相似。最终,会生成一些聚类,其中没有任何基因的隶属值超过 0.5(这个值应该是可以斟酌的),就称这些为“空聚类”,因为没有基因主要被分配到这些聚类。 空聚类的出现为设置 c 提供了依据。通过 FCM 进行的"soft"聚类反复执行,并监测非空聚类的数量, 并最终确定c值。
4.查看集群之间的耦合情况
overlap_clusters <- overlap(clusters)
pdf('mfuzz_overlap_plot.pdf',height = 4,width = 5)
p_overlaps <- overlap.plot(clusters, over = overlap_clusters, thres = 0.05)
p_overlaps
dev.off()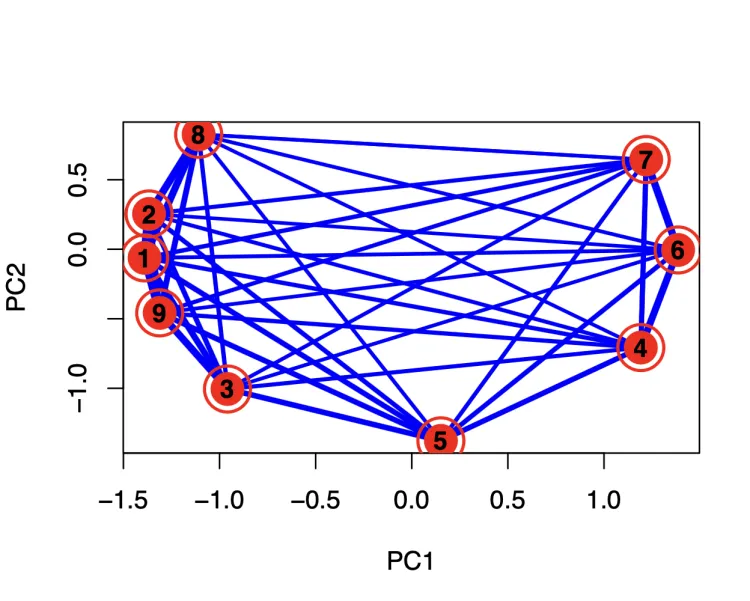
overlap(clusters)
这一步是计算聚类之间的重叠程度。overlap() 函数分析不同聚类之间的基因隶属度,以识别哪些基因在多个聚类中具有较高的隶属度。 通过计算重叠,可以评估聚类的独立性和相似性,发现哪些聚类之间存在较大的交集。这对于理解数据结构中的复杂关系非常重要。
overlap.plot
overlap.plot() 是一个可视化工具,用于展示聚类之间的重叠关系。在图中,每个节点代表一个聚类,节点之间的连线表示重叠关系,线条的粗细通常反映了重叠的程度。 这里的 thres = 0.05 是一个阈值,用于筛选显示的重叠程度。只有重叠度大于 0.05 的聚类对才会被显示,这样可以突出重要的聚类关系,避免图形过于复杂。
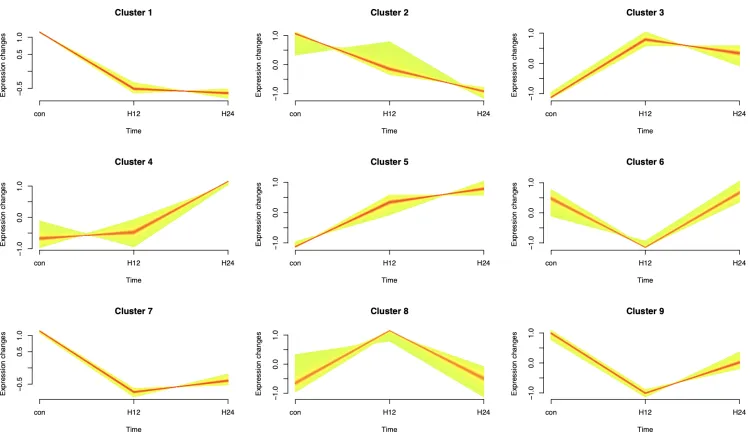
5.可视化
library(RColorBrewer)
color.2 <- colorRampPalette(rev(c("#ff0000", "Yellow", "OliveDrab1")))(1000)
pdf('mfuzz_clusters_plot.pdf',height = 7,width = 12)
mfuzz.plot(eset,clusters,mfrow=c(3,3),
new.window= FALSE,
time.labels= colnames(eset) ,
colo = color.2)
dev.off()
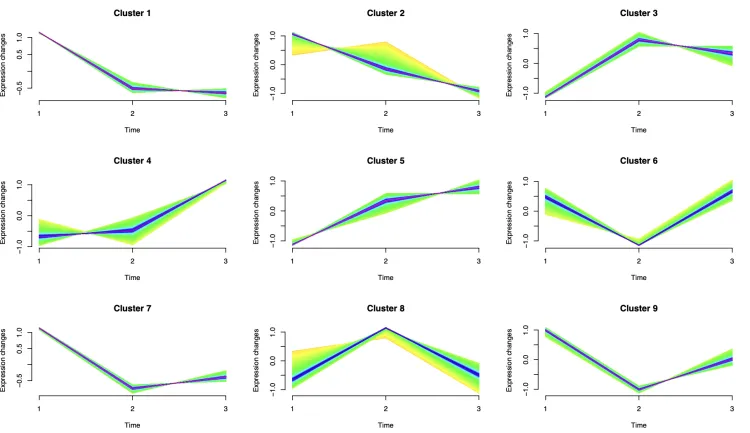
pdf('mfuzz_clusters_plot01.pdf',height = 7,width = 12)
mfuzz.plot2(eset, clusters, mfrow = c(3, 3),
centre = T,
x11 = F,
centre.lwd = 0.2)
dev.off()不同cluster的基因群随着时间变化的趋势变化~
ClusterGVis试一试
1、getClusters 选择聚类个数
用的同一批数据,直接进入分析。
library(ClusterGVis)
load(file = 'avereps_df.Rdata')
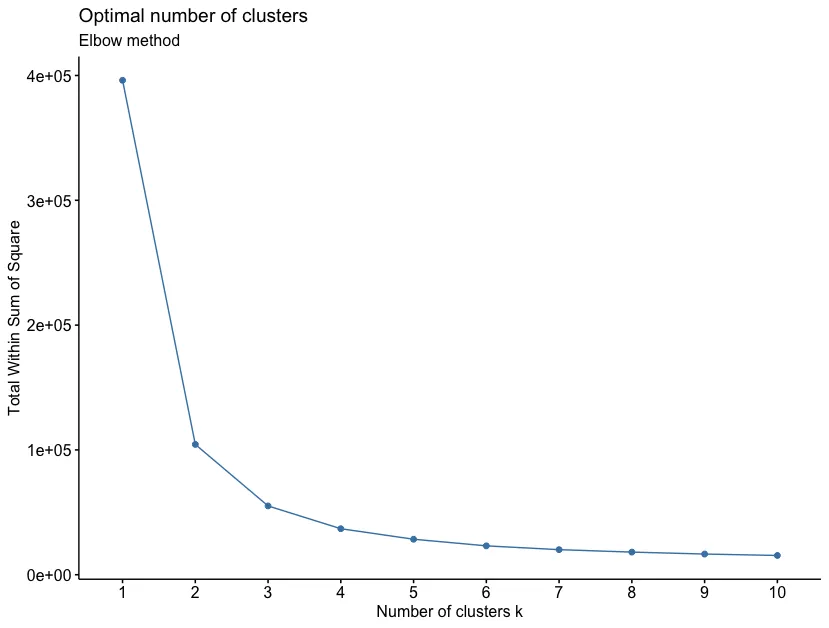
# getClusters 函数计算均方和, 用户可根据拐点确定最佳聚类个数, 首先加载测试数据:
getClusters(exp = avereps_df)
# Warning message:
# Quick-TRANSfer stage steps exceeded maximum (= 976200) 可根据拐点确定最佳聚类个数,也可以结合热图结果进行选择最佳的数量。
2、clusterData 聚类
为了跟上面数据统一,cluster设置为9
# mfuzz
cm <- clusterData(exp = exps,
cluster.method = "mfuzz",
cluster.num = 9)
# kmeans
ck <- clusterData(exp = exps,
cluster.method = "kmeans",
cluster.num = 9)3、visCluster 绘图
mfuzz/kmean结果
# plot line only
visCluster(object = cm,
plot.type = "line")
# change color
visCluster(object = cm,
plot.type = "line",
ms.col = c("green","orange","red"))
# remove meadian line
visCluster(object = cm,
plot.type = "line",
ms.col = c("green","orange","red"),
add.mline = FALSE)
# kmean结果
# plot line only with kmeans method
visCluster(object = ck,
plot.type = "line")展示一张
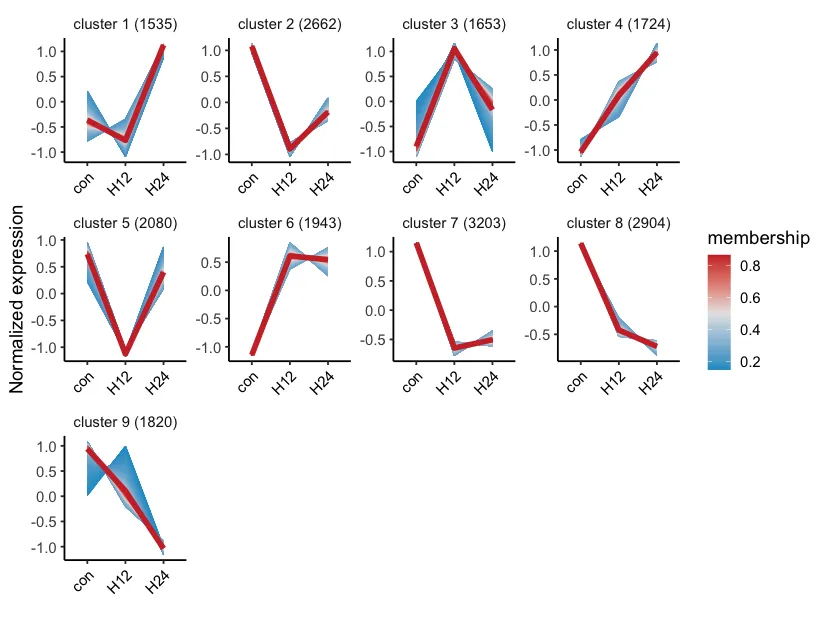
3.1 热图
# plot heatmap only
visCluster(object = ck,
plot.type = "heatmap")
# supply other aruguments passed by Heatmap function
visCluster(object = ck,
plot.type = "heatmap",
column_names_rot = 45)
# change anno bar color
visCluster(object = ck,
plot.type = "heatmap",
column_names_rot = 45,
ctAnno.col = ggsci::pal_npg()(8))
# add line annotation
pdf('testHT.pdf',height = 10,width = 6)
visCluster(object = ck,
plot.type = "both",
column_names_rot = 45)
dev.off()
# add boxplot
pdf('testbx.pdf',height = 10,width = 6)
visCluster(object = ck,
plot.type = "both",
column_names_rot = 45,
add.box = T)
dev.off()
# remove line and change box fill color
pdf('testbxcol.pdf',height = 10,width = 6)
visCluster(object = ck,
plot.type = "both",
column_names_rot = 45,
add.box = T,
add.line = F,
boxcol = ggsci::pal_npg()(8))
dev.off()
# add point
pdf('testbxcolP.pdf',height = 10,width = 6)
visCluster(object = ck,
plot.type = "both",
column_names_rot = 45,
add.box = T,
add.line = F,
boxcol = ggsci::pal_npg()(8),
add.point = T)
dev.off()
# load term info
data("termanno")
# check
head(termanno,4)
# id term
# 1 C1 developmental process
# 2 C1 anatomical structure development
# 3 C1 multicellular organism development
# 4 C2 system development
# anno with GO terms
pdf('testHTterm.pdf',height = 10,width = 10)
visCluster(object = ck,
plot.type = "both",
column_names_rot = 45,
annoTerm.data = termanno)
dev.off()
# change the line annotation side
pdf('testHTtermCmls.pdf',height = 10,width = 10)
visCluster(object = cm,
plot.type = "both",
column_names_rot = 45,
annoTerm.data = termanno,
line.side = "left")
dev.off()展示一张
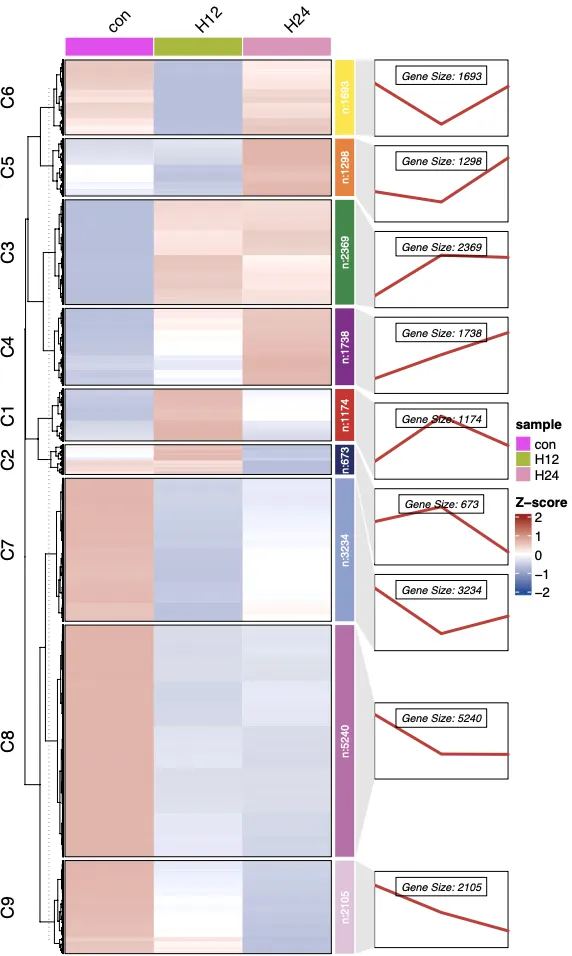
3.2还可以增加富集情况
具体不演示了
# load term info
# data("termanno")
# head(termanno,4)
# anno with GO terms
pdf('testHTterm.pdf',height = 10,width = 10)
visCluster(object = ck,
plot.type = "both",
column_names_rot = 45,
annoTerm.data = termanno)
dev.off()
# change the line annotation side
pdf('testHTtermCmls.pdf',height = 10,width = 10)
visCluster(object = cm,
plot.type = "both",
column_names_rot = 45,
annoTerm.data = termanno,
line.side = "left")
dev.off()
# remove tree
pdf('testHTtermCmlsrt.pdf',height = 10,width = 10)
visCluster(object = cm,
plot.type = "both",
column_names_rot = 45,
annoTerm.data = termanno,
line.side = "left",
show_row_dend = F)
dev.off()说实话这个R包真的是好用呀!!
参考资料:
1、Mfuzz:
Mfuzz: a software package for soft clustering of microarray data. Bioinformation. 2007 May 20;2(1):5-7. doi: 10.6026/97320630002005
https://www.rdocumentation.org/packages/Mfuzz/versions/2.32.0/topics/acore
http://mfuzz.sysbiolab.eu/
https://bioconductor.org/packages/release/bioc/manuals/Mfuzz/man/Mfuzz.pdf
2、生信技能树: https://mp.weixin.qq.com/s/EyiVXem3T_53wQMG3vRNNw https://mp.weixin.qq.com/s/kwSz5N1C0UcksFyWDXF6Ew
3、老俊俊的生信笔记: https://mp.weixin.qq.com/s/lL3v_OdOEdwrOuwchsgaFA https://mp.weixin.qq.com/s/9tv2CFI2BtYbV7j9_RYwgQ https://mp.weixin.qq.com/s/-uKyeovFaF0NFvxhxxYAwA
4、小明的数据分析笔记本 https://cloud.tencent.com/developer/article/1845577
5、ClusterGVis:
https://github.com/junjunlab/ClusterGVis
致谢:感谢曾老师以及生信技能树团队全体成员。
注:若对内容有疑惑或者有发现明确错误的朋友,请联系后台(欢迎交流)。更多内容可关注公众号:生信方舟 - END -
原创声明:本文系作者授权腾讯云开发者社区发表,未经许可,不得转载。
如有侵权,请联系 cloudcommunity@tencent.com 删除。
原创声明:本文系作者授权腾讯云开发者社区发表,未经许可,不得转载。
如有侵权,请联系 cloudcommunity@tencent.com 删除。
