课程补充----10X官方推荐的scRNA数据call SNV方法
原创课程补充----10X官方推荐的scRNA数据call SNV方法
原创
追风少年i
发布于 2024-09-18 18:17:37
发布于 2024-09-18 18:17:37
作者,Evil Genius
今天我们分享一个比较简单的,10X 官方指定的单细胞call SNV的方法。
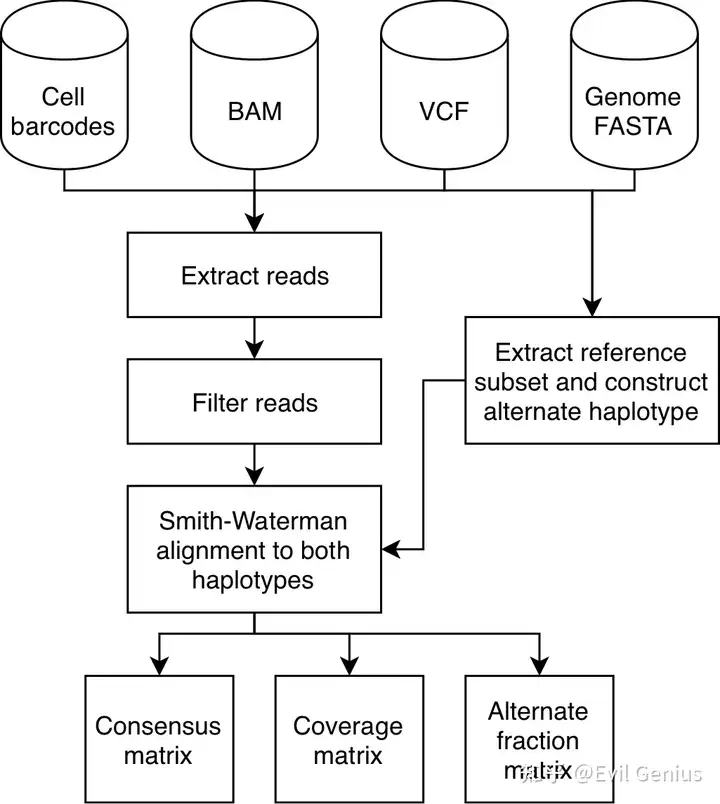
VarTrix uses Smith-Waterman alignment to evaluate reads that map to each known input variant locus and assign single cells to these variants。该过程适用于10x单个细胞基因表达数据集。
VarTrix 适用于任何格式正确的序列解析VCF。 VarTrix 适用于SNV、插入和删除。
代码示例
./vartrix -v <path_to_input_vcf> -b <path_to_cellranger_bam> -f <path_to_fasta_file> -c <path_to_cell_barcodes_file> -o <path_for_output_matrix> 其中的参数有一个比较重要,就要-v,需要WES或者WGS分析数据作为候选位点。
引用文章


记录一下,生活很好,有你更好
原创声明:本文系作者授权腾讯云开发者社区发表,未经许可,不得转载。
如有侵权,请联系 cloudcommunity@tencent.com 删除。
原创声明:本文系作者授权腾讯云开发者社区发表,未经许可,不得转载。
如有侵权,请联系 cloudcommunity@tencent.com 删除。
评论
登录后参与评论
推荐阅读
目录
