Science杂志:富集结果条形图还可以聚类吗?
Science杂志:富集结果条形图还可以聚类吗?
前面我们在学习这篇 2024 年 6 月份发表在 顶刊 science 杂志上的文献《Defining the KRAS- and ERK-dependent transcriptome in KRAS-mutant cancers》时,发现里面的图片都很美观,我们可以借来放在自己的科研文章中以提升档次。
前面给大家介绍过的:
今天再来学习一下文章中对功能富集结果的条形图进行聚类,并且还展示显著性的图。
含义:作者对一组基因 PDAC KRAS-ERK UP essential genes 进行KEGG,GOBP,GOCC 以及 REACTOME 进行 ORA 功能富集分析,根据富集的 pvalue 和 logFC 对通路进行聚类( (? 这个聚类指标很迷惑,图中横坐标展示的是log2FC,但 ORA 富集结果没有这个指标,所以这里我用的-log10(adj. p-val), 故本次绘图的通路聚类在本文中没有特殊含义,纯代码技巧)。

图注:
Fig. 4. KRAS-ERK–dependent genes are essential for cell proliferation in PDAC. (C) Overrepresentation analysis for PDAC KRAS-ERK UP essential genes using KEGG, GO, and Reactome. BP, biological process; CC, cellular component.
数据准备
1.86个 PDAC KRAS-ERK UP essential genes
PDAC KRAS-ERK UP 这个数据在文章的附件:science.adk0775_data_s4.xlsx中,关于86个 essential genes 基因的定义,我们这里就不做了,可以去看文献。我这里直接用全部的 PDAC_siKRAS_ERKi_ERKi_UP表格中的基因。
###
### Create: juan zhang
### Date: 2025-01-16
### Email: 492482942@qq.com
### Blog: http://www.bio-info-trainee.com/
### Forum: http://www.biotrainee.com/thread-1376-1-1.html
### Update Log: 2025-01-16 First version
###
rm(list=ls())
library(ggplot2)
library(clusterProfiler)
library(org.Hs.eg.db)
library(GSEABase)
library(tidyverse)
# 读取数据 一列基因集合
# PDAC KRAS-ERK UP: 表格 PDAC_siKRAS_ERKi_UP
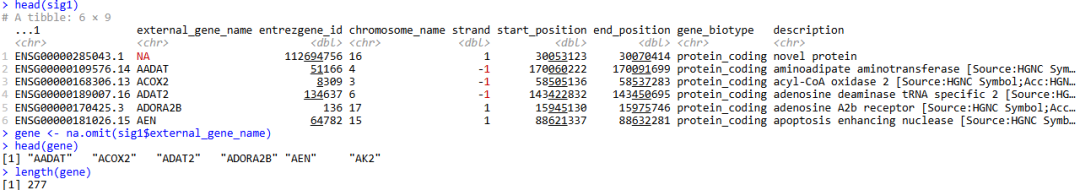
sig1 <- readxl::read_xlsx("data/science.adk0775_data_s4.xlsx", sheet = "PDAC_siKRAS_ERKi_UP")
head(sig1)
gene <- na.omit(sig1$external_gene_name)
head(gene)
length(gene)
这里共有 277个基因:
2.功能富集分析
作者用了四个基因集,KEGG,GOBP,GOCC 以及 REACTOME,我们这里都选择去 GSEA 的 MSigDB 数据库去下载 gmt 格式:https://www.gsea-msigdb.org/gsea/msigdb/human/collections.jsp
- REACTOME:https://www.gsea-msigdb.org/gsea/msigdb/download_file.jsp?filePath=/msigdb/release/2024.1.Hs/c2.cp.reactome.v2024.1.Hs.symbols.gmt
- KEGG:https://www.gsea-msigdb.org/gsea/msigdb/download_file.jsp?filePath=/msigdb/release/2024.1.Hs/c2.cp.kegg_legacy.v2024.1.Hs.symbols.gmt
- GOBP:https://www.gsea-msigdb.org/gsea/msigdb/download_file.jsp?filePath=/msigdb/release/2024.1.Hs/c5.go.bp.v2024.1.Hs.symbols.gmt
- GOCC:https://www.gsea-msigdb.org/gsea/msigdb/download_file.jsp?filePath=/msigdb/release/2024.1.Hs/c5.go.cc.v2024.1.Hs.symbols.gmt
富集:
## 读取数据库通路并ORA富集
geneset1 <- read.gmt("./MSigDB/2024.1.Hs/c2.cp.reactome.v2024.1.Hs.symbols.gmt")
geneset2 <- read.gmt("./MSigDB/2024.1.Hs/c2.cp.kegg_legacy.v2024.1.Hs.symbols.gmt")
geneset3 <- read.gmt("./MSigDB/2024.1.Hs/c5.go.bp.v2024.1.Hs.symbols.gmt")
geneset4 <- read.gmt("./MSigDB/2024.1.Hs/c5.go.cc.v2024.1.Hs.symbols.gmt")
geneset <- rbind(geneset1, geneset2, geneset3, geneset4)
head(geneset)
tail(geneset)
######################## 富集
my_path <- enricher(gene=gene, pvalueCutoff = 1, qvalueCutoff = 1, TERM2GENE=geneset)
开始绘图
这里依然是使用 ggplot2 进行绘制,ggplot2拥有强大的绘图系统。
1.绘图前数据预处理
提取数据,并增加通路的类别以及显著性符号:
# 按照fdr从小到大排序
dat <- my_path@result
dat <- dat[order(dat$p.adjust, decreasing = F), ]
# 增加一列class, 表示不同的基因集合
dat$class <- str_split(dat$ID,pattern = "_", n=2,simplify = T)[,1]
# 增加一列 -log10(p.adjust)
dat$log10PAD <- -log10(dat$p.adjust)
dat$log10PAD
# 增加一列显著性
dat$sig <- ""
dat$sig[ dat$log10PAD < 40 ] <- "****"
dat$sig[ dat$log10PAD < 30 ] <- "***"
dat$sig[ dat$log10PAD < 20 ] <- "**"
dat$sig[ dat$log10PAD < 10 ] <- "*"
dat$sig[ dat$log10PAD < 1 ] <- ""
table(dat$sig)
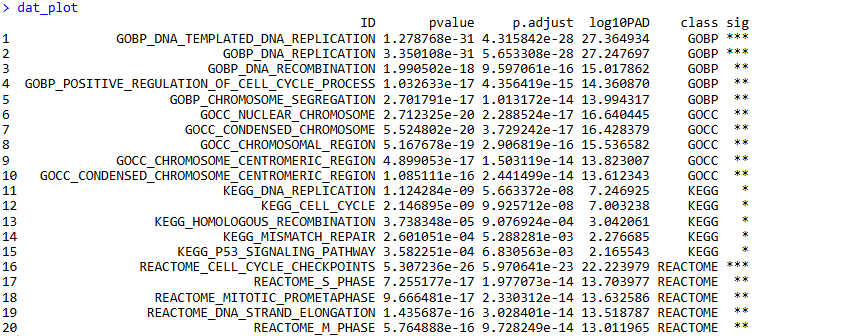
# 每个数据库按照 "p.adjust" 提取 top 5条用于绘图
dat_plot <- dat[,c("ID","pvalue","p.adjust","log10PAD","class","sig")] %>%
group_by(class) %>%
slice_head(n = 5) %>%
ungroup() %>%
as.data.frame()
dat_plot
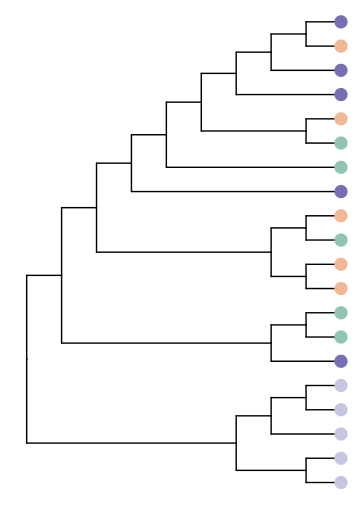
2.先绘制通路聚类树
使用 ggtree:
############################## 绘制聚类树
# 到这里的时候我感觉这个通路聚类的指标很迷惑,这里就当做单纯的绘图技巧了吧,数据没有意义
# 通路聚类至少要有两个特征
# 这里选择 pvalue, p.adjust 绘制通路聚类树
temp <- dat_plot[, c("p.adjust","log10PAD")]
rownames(temp) <- dat_plot$ID
temp
tree <- hclust(dist(temp))
tree
plot(tree, hang=-1)
# 绘图颜色
colors <- c(GOBP="#90c5b1", KEGG="#c6c6e0", REACTOME="#766fb3", GOCC="#f0b895")
colors
# 使用ggtree
p1 <- ggtree(tree, branch.length="none") %<+% dat_plot +
geom_tippoint(aes(fill=class, color=class),shape=21,size=4) +
scale_fill_manual(values = colors) +
scale_color_manual(values = colors) +
theme(legend.position = "none")
p1
树的图如下,plot(tree, hang=-1)的时候可以看到对应的通路标签:
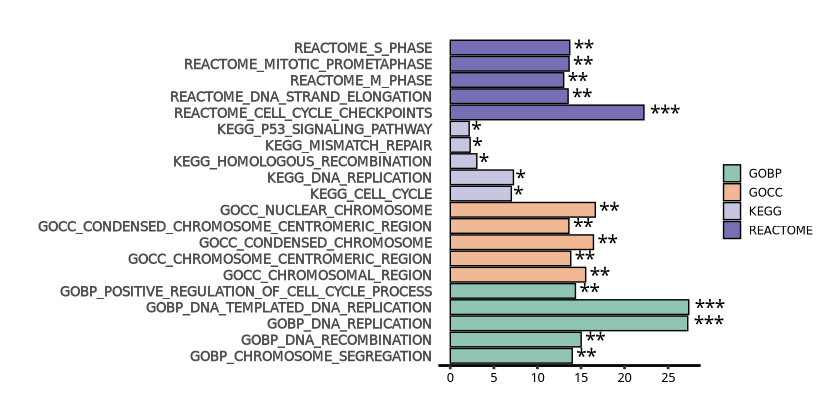
3.绘制富集条形图
ggplot 已经很熟悉了吧:
## 绘制条形图
head(dat_plot)
p2 <- ggplot(data = dat_plot, aes(x = log10PAD,y = ID,fill=class),color="black")+
geom_bar(stat="identity",position="stack",color="black") +
geom_text(aes(label = sig), hjust = -0.2, size = 5) + # 添加文本标签
labs(x="Enrichment(-log10 adj.p-val)",y=NULL) +
scale_x_continuous(expand=c(0,0)) +
coord_cartesian(clip = 'off') +
# scale_fill_brewer(palette="Paired") + # 这个色板挺好看的
scale_fill_manual(values = colors) +
scale_x_continuous(breaks = seq(0, 30, by = 5)) + # 设置x轴的刻度间隔为2
theme_classic() +
theme(axis.title.x=element_blank(),
axis.text.x=element_text(color="black",size=10),
axis.ticks.x=element_line(size = 1 ),
axis.line.x=element_line(size = 1),
# y轴设置
axis.ticks.y=element_blank(),
axis.line.y = element_blank(), # 隐藏y轴的线
axis.text.y=element_text(face = "bold",size = 10),
legend.title=element_blank(),
legend.spacing.x=unit(0.2,'cm'),
legend.key=element_blank(),
legend.key.width=unit(0.5,'cm'),
legend.key.height=unit(0.5,'cm'),
plot.margin = margin(1,0.5,0.5,1,unit="cm"))
p2
结果如下:
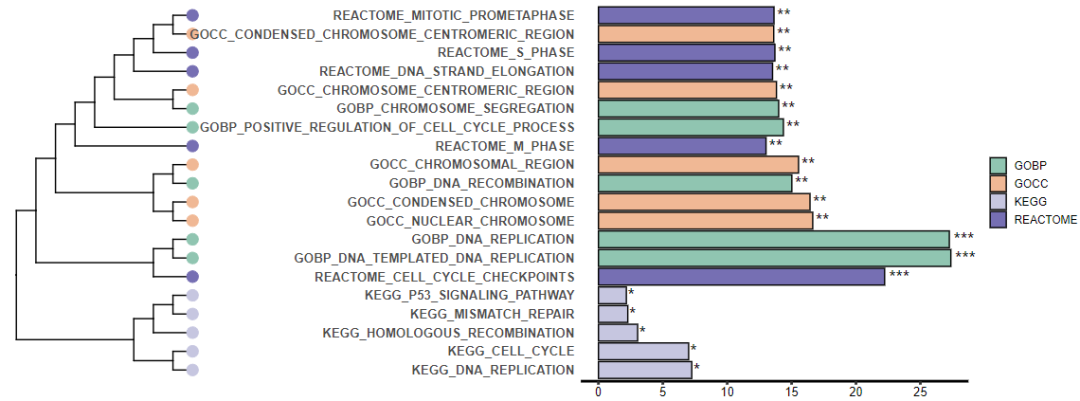
4.两个图拼在一起并保存
# 拼图
p <- p2 %>%
insert_left(p1,width=.5) %>%
as.grob() %>%
ggdraw()
p
ggsave(filename = "Fig.4C.pdf", width = 12, height = 4.5, plot = p)
结果如下:
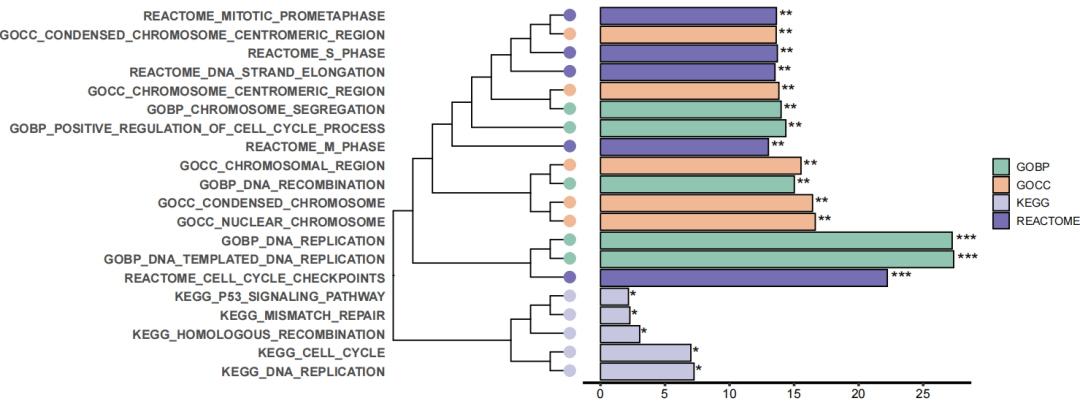
5.AI优化
用 AI 将 树与通路名字换一下位置,最终效果如下:
你学会了吗~(完)

