群体遗传三剑客第一篇:分组和不分组的PCA分析,添加解释百分比
群体遗传三剑客第一篇:分组和不分组的PCA分析,添加解释百分比
邓飞
发布于 2025-03-07 08:24:13
发布于 2025-03-07 08:24:13
大家好,我是邓飞。
之前,准备写一下群体遗传分析三剑客(搞起来!群体遗传三剑客:PCA、Admixture、进化树),第一篇来啦!
PCA是降维的一种方法,GWAS分析中经常作为协变量矫正群体分层,很多软件可以分析PCA,这里介绍一下使用plink软件和R语言,进行PCA分析,并且使用ggplot2绘制2D和3D的PCA图。
绘制后的图如下:
2-D PCA图:
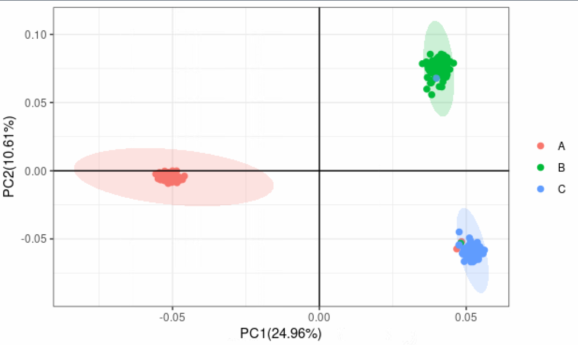
图片解释,将每个品种用不同的颜色表示,同时绘制置信区间圆圈,X坐标是PC1,解释24.9%的变异,Y坐标是PC2,解释10.61%的变异。可以看到,三个品种在PCA图里面分的比较开,C品种的有两个A和B的点,应该是异常数据。
3-D PCA图:
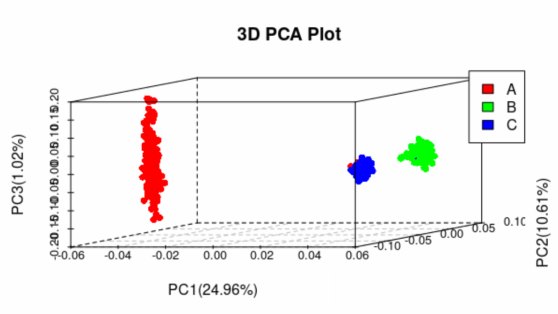
图片解释,将每个品种用不同的颜色表示,X坐标是PC1,解释24.9%的变异,Y坐标是PC2,解释10.61%的变异,Z坐标是PC3,解释1.02%的变异。可以看到,三个品种在PCA图里面分的比较开,C品种的有两个A和B的点,应该是异常数据。
基因型数据:
共有3个品种A,B,C,共有412个个体。其中:
- A品种有200个体
- B品种有100个体
- C品种有112个体
$ wc -l re2.ped
412 re2.pedSNP个数为:41013
$ wc -l re2.map
41013 re2.map计算思路:
1,对数据进行清洗,将其转化为0,1,2的形式
2,计算G矩阵
3,计算PCA的特征向量和特征值
4,根据特征值计算解释百分比
5,根据特征向量和品种标签,进行PCA的绘制
绘制代码如下:
首先,使用plink命令,将基因型数据转化为012的raw格式:
plink --file re2 --recodeA结果生成plink.raw文件。
然后使用R语言,计算PCA,并绘制PCA图。
读取数据
m012 = fread("plink.raw")
# 保留FID,IID和基因型数据
g012 = m012[,-c(3:6)]
dim(g012)
fid = g012$FID
iid = g012$IID
library(sommer)
# 整理格式,计算G矩阵
setDF(g012)
rownames(g012) = g012$IID
g012$IID = NULL
g012$FID = NULL
Gmat = A.mat(g012-1)
# 计算特征值和特征向量
re = eigen(Gmat)
# 计算解释百分比
por = re$values/sum(re$values)
# 整理格式
pca_re1 = re$vectors[,1:3]
pca_re2 = data.frame(pca_re1,Ind = iid)
pca_re2$Gen = fid
# 把PCA1,PC2,PC2的j解释百分比,命名为相应的轴
xlab = paste0("PC1(",round(por[1]*100,2),"%)")
ylab = paste0("PC2(",round(por[2]*100,2),"%)")
zlab = paste0("PC3(",round(por[3]*100,2),"%)")
# 绘制2-D PCA图
ggplot(pca_re2, aes(x=X1, y=X2,color=Gen)) + geom_point(size=2) +
# stat_ellipse(level = 0.95, size = 1) +
stat_ellipse(aes(fill=Gen),
type ="norm", geom ="polygon",alpha=0.2,color=NA)+
geom_hline(yintercept = 0) + # 添加x坐标
geom_vline(xintercept = 0) + # 添加y坐标
labs(x = xlab,y = ylab,color="")+
guides(fill=F)+
theme_bw() # 主题
# 绘制3-D PCA图
library(scatterplot3d)
pca_re2 = pca_re2 %>% mutate(colour = case_when(
Gen == "A" ~ "red",
Gen == "B" ~ "green",
Gen == "C" ~ "blue",
))
scatterplot3d(pca_re2[,1:3],color=pca_re2$colour,
pch = 16,angle=30,
box=T,type="p",
xlab = xlab, ylab = ylab, zlab = zlab,main = "3D PCA Plot",
lty.hide=2,lty.grid = 2)
legend("topright",c("A","B","C"),fill=c('red','green',"blue"))
下面是代码的解读:
这段R代码的主要目的是读取基因数据,进行主成分分析(PCA),并绘制二维和三维的PCA图来可视化数据的结构。以下是对代码各部分的详细解释:
1. 读取数据
m012 = fread("plink.raw")
fread是data.table包中的一个函数,用于快速读取文本文件。这里尝试从名为plink.raw的文件中读取数据,并将其存储在变量m012中。
2. 保留FID,IID和基因型数据
g012 = m012[,-c(3:6)]
dim(g012)
fid = g012$FID
iid = g012$IID
m012[,-c(3:6)]表示从m012数据框中移除第3列到第6列的数据,将结果存储在g012中。dim(g012)用于查看g012数据框的维度(行数和列数)。- fid = g012FID 和 iid = g012IID 分别提取 g012 数据框中的 FID(家族ID)和 IID(个体ID)列。
3. 加载 sommer 包
library(sommer)
sommer是一个用于混合模型分析的R包,这里主要使用其中的A.mat函数来计算亲缘关系矩阵。
4. 整理格式,计算G矩阵
setDF(g012)
rownames(g012) = g012$IID
g012$IID = NULL
g012$FID = NULL
Gmat = A.mat(g012-1)
setDF(g012)将g012转换为标准的数据框格式。rownames(g012) = g012$IID将g012的行名设置为IID列的值。- g012IID = NULL 和 g012FID = NULL 分别移除 g012 中的 IID 和 FID 列。
Gmat = A.mat(g012-1)使用A.mat函数计算g012数据减去1后的亲缘关系矩阵Gmat。
5. 计算特征值和特征向量
re = eigen(Gmat)
eigen函数用于计算矩阵的特征值和特征向量,结果存储在re中。
6. 计算解释百分比
por = re$values/sum(re$values)
- revalues 是特征值向量,sum(revalues) 计算所有特征值的总和,por 存储每个特征值的解释百分比。
7. 整理格式
pca_re1 = re$vectors[,1:3]
pca_re2 = data.frame(pca_re1,Ind = iid)
pca_re2$Gen = fid
pca_re1提取特征向量矩阵的前3列。pca_re2是一个新的数据框,包含前3个特征向量和个体IDiid。pca_re2$Gen = fid将家族IDfid添加到pca_re2中。
8. 命名坐标轴
xlab = paste0("PC1(",round(por[1]*100,2),"%)")
ylab = paste0("PC2(",round(por[2]*100,2),"%)")
zlab = paste0("PC3(",round(por[3]*100,2),"%)")
paste0函数用于拼接字符串,round函数用于四舍五入。这里分别计算并命名PC1、PC2和PC3的解释百分比作为坐标轴标签。
9. 绘制2-D PCA图
ggplot(pca_re2, aes(x=X1, y=X2,color=Gen)) + geom_point(size=2) +
stat_ellipse(aes(fill=Gen),
type ="norm", geom ="polygon",alpha=0.2,color=NA)+
geom_hline(yintercept = 0) +
geom_vline(xintercept = 0) +
labs(x = xlab,y = ylab,color="")+
guides(fill=F)+
theme_bw()
- 使用
ggplot2包绘制二维PCA图。 aes(x=X1, y=X2,color=Gen)指定x轴为X1,y轴为X2,颜色根据Gen列进行区分。geom_point(size=2)绘制散点图,点的大小为2。stat_ellipse绘制椭圆,用于表示每个组的分布。geom_hline和geom_vline分别添加x轴和y轴的参考线。labs用于设置坐标轴标签和颜色标签。guides(fill=F)隐藏填充图例。theme_bw()设置绘图主题为黑白主题。
10. 绘制3-D PCA图
library(scatterplot3d)
pca_re2 = pca_re2 %>% mutate(colour = case_when(
Gen == "A" ~ "red",
Gen == "B" ~ "green",
Gen == "C" ~ "blue",
))
scatterplot3d(pca_re2[,1:3],color=pca_re2$colour,
pch = 16,angle=30,
box=T,type="p",
xlab = xlab, ylab = ylab, zlab = zlab,main = "3D PCA Plot",
lty.hide=2,lty.grid = 2)
legend("topright",c("A","B","C"),fill=c('red','green',"blue"))
- 加载
scatterplot3d包用于绘制三维散点图。 - 使用
mutate和case_when函数根据Gen列的值为每个个体分配颜色。 scatterplot3d函数绘制三维散点图,指定颜色、点的形状、角度等参数。legend函数在图的右上角添加图例,显示不同Gen组对应的颜色。
本文参与 腾讯云自媒体同步曝光计划,分享自微信公众号。
原始发表:2025-03-05,如有侵权请联系 cloudcommunity@tencent.com 删除
评论
登录后参与评论
推荐阅读
目录

