资源 | 人类生物分子图谱计划(HuBMAP):3D人体参考图谱的构建与使用
资源 | 人类生物分子图谱计划(HuBMAP):3D人体参考图谱的构建与使用

Basic Information
- 英文标题:Human BioMolecular Atlas Program (HuBMAP): 3D Human Reference Atlas construction and usage
- 中文标题:人类生物分子图谱计划(HuBMAP):3D人体参考图谱的构建与使用
- 发表日期:13 March 2025
- 文章类型:Resource
- 所属期刊:Nature Methods
- 文章作者:Katy Börner | Bruce W. Herr II
- 文章链接:https://www.nature.com/articles/s41592-024-02563-5
Abstract
Para_01
- 人类生物分子图谱计划(HuBMAP)旨在构建一个健康成年人体的三维人体参考图谱(HRA)。
- 来自20多个联盟的专家合作开发了一个通用坐标框架(CCF),知识图谱和工具,用于描述人体的多尺度结构(从器官和组织到细胞、基因和生物标志物)。
- 他们使用HRA来表征随着年龄增长、疾病和其他扰动发生的变化。
- HRA版本2.0涵盖了4,499个独特的解剖结构,1,195种细胞类型和2,089个生物标志物(如基因、蛋白质和脂质),这些信息来源于33个ASCT+B表格和65个与本体论相关的3D参考对象。
- 新的实验数据可以通过(1)细胞类型注释工具(例如Azimuth)、(2)经过验证的抗体面板或(3)通过空间注册组织数据的方式映射到HRA。
- 本文描述了HRA用户故事、术语、数据格式、本体论验证、统一分析工作流程、用户界面、教学材料、应用程序编程接口、灵活的混合云基础设施以及图谱使用应用的预览。
Main
Para_01
- 人类生物分子图谱计划(HuBMAP)成立于2018年,旨在从器官和组织到细胞和典型生物标志物等各个层面构建一个全面的健康(‘非疾病’)人体参考模型。
- HuBMAP门户(https://hubmapconsortium.org)介绍了目标,并提供了实验数据和图集数据、工具以及培训材料的链接。
- 数据门户(https://portal.hubmapconsortium.org)提供实验数据集,并支持数据处理、搜索、筛选和可视化。
- 人类参考图谱门户(https://humanatlas.io)提供对图集数据、代码、程序和教学材料的开放访问。
- 人类参考图谱(HRA)包括一个通用坐标框架(CCF;参见框1),该框架有助于协调多模态数据,包括三维(3D)器官模型、组织学图像和来自单细胞分析的组学数据。
- HRA数据包括由人类专家生成的信息(例如,解剖系统;解剖结构、细胞类型、生物标志物(ASCT+B)表以及二维(2D)和三维参考对象)、映射到HRA的实验数据,以及支持不同图谱应用的增强图谱数据。
- HRA和ASCT+B表的起源和发展在之前的文献中有详细描述。
- CCF为将新的实验数据(如组织学图像、血管路径和单细胞分析)整合到不断增长的图谱中提供了定量工作流程。
- 由此产生的HRA为人体内特定三维位置上的细胞和解剖结构的共同状态提供了数据证据,这可以作为标准参考来描述生物变量(例如年龄、性别、种族和体重)和急性或慢性疾病的改变。
- 它可以通过更好地理解疾病状态下细胞类型和状态的扰动,从而通过比较患病组织与非患病组织来揭示精准医学的相关靶点,并提供与CCF匹配的参考,从而在药物开发等领域受益。
Para_02
- 当HuBMAP启动时,正在出现一些统一的概念,这些概念可以跨尺度绘制人体主要器官。
- 现有的图谱使用特定于器官的参照系(例如,大脑使用Waxholm空间,结肠使用Helmsley一维距离参照系统),但大多数这些参照系并不能映射到一个共享的人体CCF。
- 为了推进CCF的发展,在2020年3月,美国国立卫生研究院(NIH)和人类细胞图谱(HCA)联盟组织了一次与CCF分组会议联合举行的虚拟会议。
- 这导致了HRA工作组(WG)的成立。
- 在过去55个月里,工作组成员共同制定了HRA的定义和关键属性。
- 这些属性是:
- HRA定义了解剖结构和细胞类型的参考3D多尺度空间和形状,以及用于表征细胞类型的生物标志物。解剖结构、细胞类型和生物标志物经过验证后被添加到现有本体论中(例如,超解剖学本体论(Uberon)9、解剖学基础模型本体论(FMA)10,11、细胞本体论(CL)12、临时细胞本体论(PCL)13和人类基因本体论命名委员会(HGNC; https://www.genenames.org))。随着更多数据的收集,HRA将能够越来越多地展示身体形状和大小,以及细胞类型群体如何在个体间差异并随着人的一生变化。
- HRA能够通过各种机制添加新的实验数据集并将其映射到现有数据。例如,可以相对于HRA中的虚拟3D参考器官模型指定组织标本的位置;可以使用像Azimuth14这样的注释工具将单细胞基因组数据映射到HRA;并且可以使用经验证的器官映射抗体面板(OMAPs)15将单细胞分辨率空间蛋白质组学数据映射到HRA。随着未来新技术和计算方法的发展,将可能实现额外的映射和联系,如多组学数据的整合。
- HRA遵循分享科学数据的最佳实践和标准。为此,HRA需要具有权威性(应得到同行评审的学术出版物、实验数据证据或专家共识的支持);满足数字存储库的透明度、责任、用户关注、可持续性和技术(TRUST)原则16;具有代表性(覆盖所有主要人类人口统计群体并欢迎所有人贡献和使用HRA数据);开放并遵守可查找、可访问、可互操作、可重用(FAIR)原则17(任何人都可以使用HRA数据和代码,这些数据和代码以社区标准格式与链接本体论提供;作为链接开放数据(LOD)发布,连接到本体论和其他LOD;支持应用程序编程接口(API)查询和用户界面);提供详细的协议和标准操作程序(SOPs);并不断发展(例如,随着新技术、数据和方法的可用)。
Para_03
- 专家们还就图集构建和使用的SOP达成了一致,并采纳了HRA术语(见框1),这些术语来自HRA SOP术语表18。此外,HRA工作组汇集了HuBMAP整合、可视化与参与(HIVE)合作的技术负责人、HuBMAP的实验团队以及来自基因型-组织表达(GTEx)19、GUDMAP:泌尿生殖系统发育分子解剖学项目20、肾脏精准医学项目(KPMP)21,22、LungMAP23,24、大脑计划细胞普查网络(BICCN)25,26、细胞衰老网络(SenNet)27以及其他由NIH资助的联盟的专家,并得到了HCA工作的大力支持,共同开发了HRA数据、代码和门户基础设施。
- 此外,HRA工作组将HuBMAP集成、可视化与参与(HIVE)协作的技术负责人与HuBMAP的实验团队以及来自基因型-组织表达(GTEx)19、GUDMAP:泌尿生殖发育分子解剖项目20、肾脏精准医学项目(KPMP)21,22、肺图谱(LungMAP)23,24、脑细胞普查网络倡议(BICCN)25,26、细胞衰老网络(SenNet)27和其他NIH资助的联盟的专家聚集在一起,并得到HCA工作8,28的大力支持,共同开发HRA数据、代码和门户基础设施。
Para_04
- HR努力的一个重要下一步是收集用户故事,以支持图集的构建和使用,从而鼓励设计师和用户围绕三个关键问题进行对话、讨论和迭代:哪些用户涉及在特定的用户故事中?他们希望实现什么成果?他们能获得什么价值?对这些问题答案的共识有助于优先考虑用户需求,为提议的用户故事提供背景,并减少歧义。
- 同意这些问题的答案帮助确定了用户的优先需求,为提出的用户故事提供了上下文,并减少了模糊性。
Para_05
- 进行了三十多次与图集架构师(作为首席研究员或以其他方式深入参与最新一代人类图集构建的专家,包括BICCN、GTEx、GUDMAP、HCA、HuBMAP、人类肿瘤图集网络(HTAN)29、KPMP、LungMAP、(Re)building the Kidney(RBK)30和SenNet)的一对一访谈。
- 鉴于这一努力的跨学科性质,受访的图集架构师组成了一群多样化的医生、实验室和计算生物学家、工程师以及计算机和数据科学家。
- 此外,还调查了来自不同人类图集项目的六名程序员。
Para_06
- 访谈和调查结果帮助确定了建设、使用和可持续性三个方面的主要目标以及七个具体的用户故事(US 1–7)(表1)。
- 这三个目标是:
- HRA应通过将新的组织块与现有数据对齐来促进图谱构建。例如,HRA的开发者希望预测新组织块的细胞类型群体(表1中的用户场景1)并预测已知细胞类型群体的组织样本的空间起源(用户场景2)。
- HRA应包含提供洞察身体所有层面变化(例如,随着年龄增长、疾病或其他扰动)的功能。为此,研究人员和临床医生需要能够搜索和探索组织和功能组织单元(FTUs)的细胞类型和生物标志物表达值(用户场景3和用户场景4),并确定细胞之间的位置和距离(用户场景5)。
- HRA应使用鼓励协作并指导未来发展以确保长期可持续性的流程。这包括利用具有模块化、轻量级组件的架构,这些组件可以轻松共享(用户场景6),并通过HRA仪表板向研究人员、临床医生和资助者提供成功指标,以获得反馈和支持(用户场景7)。
Table 1 User stories. Feature summary, target user roles, user activities and added value for seven user stories that drive HRA development 表1 用户故事。推动HRA开发的七个用户故事的功能摘要、目标用户角色、用户活动及附加价值

Para_07
- 这三个关键目标和相关的用户故事有助于聚焦每月HRA工作组中的演讲和讨论;它们推动了HRA的发展和迭代优化。
- 每六个月,会发布一个新的HRA版本。每次发布时,现有的本体论会被扩展,并且HRA数据结构和算法会被改进,以便更好地服务于国际人类图谱社区的需求。
- 图1详细介绍了HRA第六版的主要组成部分及其相互联系。
Fig. 1: Human Reference Atlas components and linkages.

- 图片说明
◉ ASCT+B表格文件记录了器官的嵌套‘part_of’结构(例如,构成肾单位的细胞、逐步更大的解剖结构、整个器官如肾脏,肾脏是身体的一部分)。◉ 构成(位于)每个解剖结构的细胞在多层次的细胞类型分类学中组织起来,以‘细胞’为根节点,并且具有越来越专业的子节点和节点之间的‘is_a’关系。◉ 用于表征细胞类型的生物标记物可能有五种类型:基因、蛋白质、代谢物、蛋白质形式和脂质,它们被组织在一个生物标记物分类学中。◉ 灰色箭头表示交叉引用,连接其他HRA DO到ASCT+B表格。◉ HRA 3D参考对象代表1,192个3D解剖结构的形状、大小、位置和旋转,这些结构拥有516个独特的Uberon ID,涉及65个器官,并与ASCT+B表格存在交叉引用。◉ 显示的是肾脏中的‘肾乳头’和‘肾锥体’。◉ 2D参考图示记录了22个肾单位中的10个器官的3,742个2D细胞的形状、大小和空间布局,共有116种类型,并且与ASCT+B表格存在交叉引用。◉ 显示的是肾脏的肾小球。◉ 对于五个器官中的肾单位存在带交叉引用(灰色箭头)的标记训练数据,这些数据指向ASCT+B表格中的解剖结构和细胞类型。◉ 13个OMAP链接到197个AVR,并且存在与ASCT+B表格中的细胞类型和生物标记物的交叉引用。◉ 十个Azimuth参考针对健康成年器官,并且与ASCT+B表格中的细胞类型和生物标记物存在交叉引用。◉ HRApop报告从实验数据中收集的解剖结构的细胞类型群体。◉ 例如,显示女性心脏的左心房(蓝色)和室间隔(橙色),以及一个条形图,该条形图显示在这两个解剖结构中百分比最高的细胞类型(用Azimuth注释)。◉ 请注意,某些细胞类型仅出现在一个解剖结构中。◉ HRAlit数据库将HRA DO链接到现有的本体(例如,Uberon和CL)、专家ORCID、出版物证据、资金和用于HRApop计算的实验数据。
[div_box]
Results
Para_01
- HIVE 基础设施和参与组件(IEC)开发了 HuBMAP 的灵活混合云微服务架构(补充图1和方法部分),以支持通过 HuBMAP 数据门户(https://portal.hubmapconsortium.org)的数据策展、摄取、集成、访问、分析、探索和下载。
- HIVE 工具组件专注于 HuBMAP 数据门户用户界面、可视化、工作流集成和工具开发。
- HIVE 绘制组件与外部专家密切合作,开发了 Azimuth14 参考和 HRA 门户(https://humanatlas.io)。
Para_02
- HuBMAP联盟网站(补充图2)提供了对HuBMAP资源、出版物、新闻、实习项目、会员服务等的便捷访问。
- 它链接到HuBMAP数据门户和HRA门户。
- HuBMAP数据门户提供了对HuBMAP数据、APIs和用户界面的访问,并且数据和代码持续发布。
- HRA门户提供由18个项目创建的图集级数据和代码,新的HRA发布每六个月更新一次。
- 两个门户都使用知识图谱(KGs)来存储数据,HRA知识图谱定期被导入统一生物医学知识图谱(UBKG;https://ubkg.docs.xconsortia.org),以连接HuBMAP实验数据与现有本体论和HRA。
- HRA使用HuBMAP和其他实验数据计算解剖结构的细胞类型群体(见方法和补充表1中的HRA细胞类型群体(HRApop))。
- 多个HRA用户界面(见用户界面部分)部署在HuBMAP数据门户和其他门户中,以支持HRA的构建和使用。
Para_03
- Atlas构建复杂,需要社区在数据格式、API和用户界面方面达成一致。
- 预览用于在新功能集成到HuBMAP或HRA门户之前进行展示和优化。
- 主要数据存储库列在补充表2中,HRA代码存储库列在补充表3中。
Flexible hybrid cloud infrastructure for HRA and HuBMAP
灵活混合云基础设施用于HRA和HuBMAP
Para_01
- 系统集成超过50个由超过30个团队开发的开源算法并非易事。
- 为了使一个算法的输出与下一个(组)算法预期的输入兼容,需要就元数据和API调用达成一致。
- 一些对组织分割和注释至关重要的算法是由具有深厚主题专业知识的生物学家开发的,但他们对如何构建生产管道的知识有限。
- HIVE 生产开发团队与原始算法作者密切合作,将他们的算法打包,以便可以在灵活且可扩展的混合云基础设施上大规模可靠运行,以满足不断变化的需求。
Para_02
- 具体来说,由匹兹堡超级计算中心(PSC)、匹兹堡大学(Pitt)和斯坦福大学成员组成的HIVE IEC实施了一个灵活的混合云基础设施和社区参与平台,支持HuBMAP愿景在以下关键领域的实现:
- (1) 收集和输入:目前从HuBMAP数据提供商处进行半自动数据输入(https://software.docs.hubmapconsortium.org),未来将从社区合作伙伴和广大研究社区获取,以最大化效率和对构建HRA的有用性;
- (2) 集成:自动化分析和注释输入的数据,并通过UBKG将这些注释与HRA对齐;
- (3) 可发现性和可访问性:后端资源在APIs和容器、服务和文档(https://software.docs.hubmapconsortium.org)的模块化架构中的表现,减少了用户在集成搜索、查询、分析和查看HuBMAP数据以及将来在多个空间尺度和多层信息中的组织图方面的摩擦;
- (4) 互操作性:使用HuBMAP部署的UBKG及其扩展创建HuBMAP本体API(https://smart-api.info/ui/d10ff85265d8b749fbe3ad7b51d0bf0a),通过本体论在HuBMAP数据、HRA资产和社区数据之间进行翻译;HuBMAP本体API包含查询具有HuBMAP上下文内容的UBKG实例的端点(https://ubkg.docs.xconsortia.org/contexts/#hubmapsennet-context);
- (5) 分析:基础设施支持当前使用户能够通过Jupyter笔记本与HuBMAP数据进行交互分析,并在未来,针对HuBMAP和用户贡献的数据和工具进行批量工作流,包括整合和映射到HRA;
- (6) 可持续性:HuBMAP的灵活混合云基础设施(有效地利用了PSC本地资源来提供在本地比公共云成本更低的服务,如数据存储、处理、分析和下载(补充图1和方法部分))将有助于开放工具、数据和基础设施在HuBMAP项目结束后的可持续性。
Atlas construction and publication
图集构建与发布
Para_01
- HRA 数据包括由人类专家生成的数据(例如,ASCT+B 表格、OMAPs、抗体验证报告(AVRs)和二维/三维参考对象),通过注册用户界面(RUI)位置、HRA 对齐的细胞类型注释(CTann)或 OMAP/AVR 映射到 HRA 的实验数据,以及丰富的大脑图谱数据(例如,HRApop 和 HRA 文献(HRAlit));详见图 1 中关于 HRA 数字对象(DO)类型及其交叉引用的概述(有关术语和方法的详细信息,请参见方框 1)。HRA 数据、本体的使用和扩展、统一的数据处理工作流程、用户界面、文档和教学材料在这里进行了详细介绍。
- HRA 数据、本体的使用和扩展、统一的数据处理工作流程、用户界面、文档和教学材料是这里详细说明的。
Data types and status
数据类型和状态
Para_01
- HRA v.2.0(2023年12月)第六次发布包括一个解剖结构系统图,该图将主要器官分组到器官系统中(例如,消化系统和生殖系统);三个ASCT+B表格代表血液和淋巴以及周围神经系统中的分支结构;以及29个ASCT+B表格记录了其他器官的嵌套‘部分组成’结构(例如,肾脏由构成较小和随后较大FTU和器官部分的细胞组成),总计33个ASCT+B表格。
- 组成每个解剖结构的细胞在一个多层次的细胞类型分类中组织起来,其中‘细胞’位于根部,继而有更专业的子节点。细胞映射到五种生物标志物类型:基因、蛋白质、代谢物、蛋白质形式和脂质,这些生物标志物类型组织在一个生物标志物分类中。
Para_02
- 基于解剖结构的3D参考对象(图1b)包括65个器官中的1,192个三维解剖结构的形状、大小、位置和旋转,这1,192个三维解剖结构包含516个独特的本体ID。
- 一个SPARQL查询(https://apps.humanatlas.io/api/grlec/ccf.html#get-/as-3d-counts)返回所有具有Uberon ID的解剖结构(它检索了1,192个解剖结构加上65个器官,总计1,257个项目)。
- 二维参考(图1c)描述了10个器官的22个功能单元(FTU)中3,742个渲染的二维细胞的布局,这些细胞来自116种独特的细胞类型。
- 用于空间分割和机器学习模型的标记训练数据(图1d)存在于五个器官的五个功能单元中。
- 总共13个OMAP与197个AVR相关联,并与ASCT+B表格对齐。
- 细胞类型注释工具(图1f)包括Azimuth和其他针对健康成人器官的参考,并且与ASCT+B表格中的细胞类型和生物标志物有交叉引用。
Para_03
- HRA处理的一个重要部分是数据丰富化。
- 一个例子是HRApop(图1g),它涵盖了用于计算40个具有三维参考对象的解剖结构的细胞类型群体的553个组织数据集,涉及23个器官和13个独特的Uberon ID。
- 可以使用代码重现包含HRApop数据(七个数据集)的条形图31。
- HRAlit32(图1h)将HRA DOs链接到7,103,180篇出版物、583,117位作者、896,680个项目资助和1,816个实验数据集。
Data enrichment
数据增强
Para_01
- 这一HRA处理步骤确保了HRA DOs的质量高、可用且对表1中列出的用户故事和其他应用有用。
- 规范化确保原始数据结构良好,并以可通过LinkML(https://linkml.io)轻松转换为知识图谱的格式呈现。
- 在富集过程中,使用OWL推理使某些隐含关系变得明确(例如,使传递关系如子类和‘部分’变得明确);通过API从本体中添加外部元数据以增强图谱的实用性(例如,通过查询scicrunch API查找OMAP抗体信息);使用查询添加来自相关图谱的数据(例如,从流行的生物医学本体如Uberon和Cell Ontology中提取与解剖结构、细胞类型和生物标志物相关的附加元数据和层次结构);并最终将LinkML转换为知识图谱(例如,将所有内容转换并组合成Turtle格式的RDF格式图谱)。
Data publication
数据发布
Para_01
- 一个新修订和扩展版本的HRA DOs以及更新后的用户界面和API每六个月通过HRA门户(https://humanatlas.io)发布。
- 三个HRA核心本体(样本、生物结构和空间本体)7作为FAIR、版本化的LOD共享在https://lod.humanatlas.io。
- 部分数据也以关系数据库和逗号分隔值(CSV)文件的形式提供。
- RUI数据通过HuBMAP、SenNet、GUDMAP、GTEx和其他门户发布。
- 例如,HRA API通过查询HuBMAP搜索API来从HuBMAP数据生成数据集图。
- 包含所有捐赠者、组织块、组织切片、RUI数据和实验数据集信息的公共图形可以通过HRA数据集图在https://lod.humanatlas.io/ds-graph访问。
Para_02
- HRA DO处理器(https://github.com/hubmapconsortium/hra-do-processor)支持HRA数据的自动化处理,包括数据规范化、验证、图形转换、丰富化和发布。
- 最终产品是HRA知识图谱(https://github.com/hubmapconsortium/hra-kg)和一套适合托管所有数据作为LOD的平面文件。
- HRA基础设施针对部署到Amazon S3、Amazon Web Services (AWS) AppRunner和AWS CloudFront进行了优化,但也可以适应其他文件托管平台。
Para_03
- HRA 来源图谱跟踪所有 HRA DO(使用 DCAT(https://www.w3.org/TR/vocab-dcat)标准术语组织数据目录和 W3C-Prov(https://www.w3.org/TR/prov-overview)描述特定数据的来源)以及代码版本(通过 GitHub),以便可以访问 HRA 知识图谱的来源,并且每六个月可以重新计算一次 HRA 知识图谱。
Para_04
- 补充表 2 列出了 HuBMAP 数据门户(H)、HRA 数据门户(A)和演示预览(P)中使用的所有数据。
- 请注意,HRA 数据由欧洲生物信息学研究所(EBI)的本体查找服务(OLS)、斯坦福大学的 NCBO 生物门户和密歇根大学医学院的 Ontobee 镜像。
- 通过广泛使用的生物医学本体存储库发布 HRA 使其变得 FAIR;用户可以在线浏览 HRA 数据或通过 API 程序化访问它。
Usage and extension of ontologies
本体的使用和扩展
Para_01
- 数据和工作流程尽可能地与现有的本体论链接(表2)。
- HRA v.2.0的第六版使用生物结构本体论Uberon 2023-10-27(参考文献9)和FMA v.5.0.0(参考文献10,11)来表示解剖结构;
- 细胞类型使用细胞本体论(CL)v.2023-10-19(参考文献12)和PCL 2023-02-27(参考文献13)(https://www.ebi.ac.uk/ols4/ontologies/pcl);
- 生物标志物使用HGNC v.2023-09-18(参考文献33),Ensembl发布版111(参考文献34),GeneCards v.5.19:2024年1月15日(参考文献35,36)以及UniProt发布版2024_1(参考文献37)。
- Human Genome HGNC v.2023-09-18用于FTU探索者。
- 空间数据使用Dublin核心术语(DCTERMS)v.2020-01-20(https://www.dublincore.org)进行注释。
- 样本数据使用LOINC v.2022-07-11(v.2022AB)38来标准化表示性别、种族和族裔数据。
- 元本体论如DCTERMS和关系本体论39(RO)用于捕捉HRA数据内部概念之间的关系。
- 分析类型名称来自生物分析本体论(BAO)v.2023-01-31(参考文献40)和实验因素本体论(EFO)v.2023-02-15(参考文献41)。
- 鼓励使用这些本体论以保持ASCT+B表格、Azimuth和其他CTann工具以及OMAP数据之间的一致性,支持图集的构建和使用。
Table 2 Ontologies used and extended 表2 使用和扩展的本体
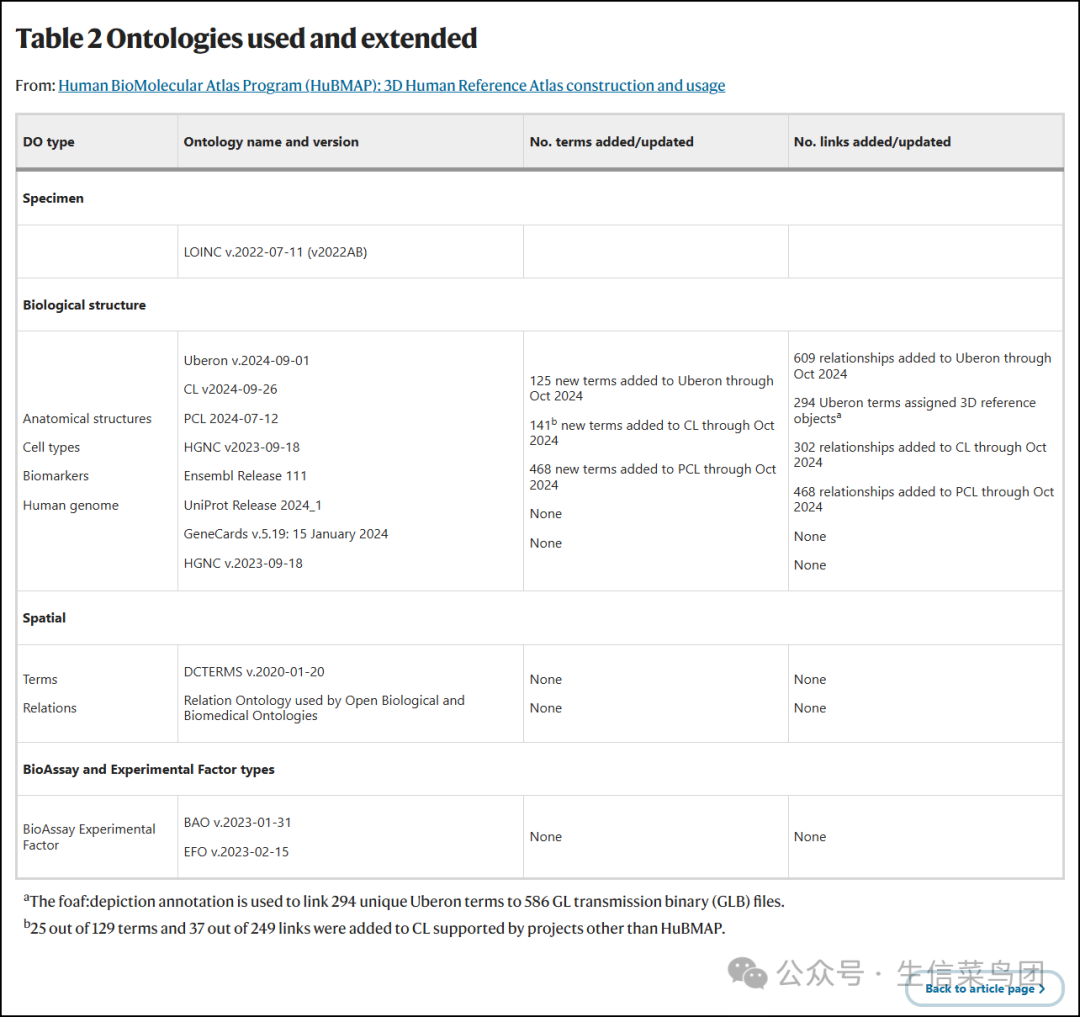
Para_02
- 跨联盟HRA工作的一个重要贡献是扩展了跨物种本体论,如Uberon和CL,以涵盖健康人类术语。
- 从2021年到2024年10月,Uberon中增加了125个解剖结构术语,Cell Ontology中增加了141个细胞类型。
- 截至2024年10月,PCL中增加了468个细胞类型,其中461个是针对人脑的(支持HRA的构建和使用)。
- PCL使用来自NS-Forest的计算得出的标记基因来定义基于sc/snRNA-seq的脑细胞类型。
- 这461个人脑细胞类型被添加到了ASCT+B表格中。
- 所有PCL细胞类型的术语都通过本体中的has_characterizing_markerset关系与生物标记基因相关联。
- 在HRA的第六次发布中,有962个解剖结构术语要么缺失于Uberon,要么尚未在ASCT+B表格中交叉映射到Uberon术语。
- 大多数缺失的术语是关于血液和淋巴血管系统、骨骼或骨骼肌系统的,并且通常比目前在Uberon中表示的更具体(例如,‘手第五指背侧分支外侧正掌数字动脉’)。
- 正在进行改进映射的工作(最近添加了约100个映射,并将在第七次HRA发布中公布)。
- 总共有119个细胞类型未映射或尚未在CL或PCL中(初步评估表明有60个),其中70%对于CL来说是全新的术语。
- 这387个生物标记物具有Ensembl ID或GeneCards ID,或者尚未映射到HGNC ID(所有这些术语都有ASCTB-TEMP ID)。
- 存在GitHub问题,旨在将新术语添加到现有本体中,以便正确表示ASCT+B表格中的数据,包括请求在Uberon中增加128个解剖结构。
- 现在有一套正式的操作程序,通过Minimal Information Reporting About a CelL (MIRACL)表单将新的细胞类型纳入CL。
- 表2的最后一列列出了添加到Uberon、CL和PCL的本体关系数量。
Unified processing workflows
统一处理工作流程
Para_01
- HRA SOPs45 详细说明了构建HRA所需的人类专家和算法步骤以及如何正确使用它。
- 在protocols.io和其他地方发布的协议用于以可重复的方式编译实验数据。
- 截至2024年1月,已有235个HuBMAP协议46(其中许多记录了生成用于HRA构建的数据所需的可重复工作流程)。
- 图1和图2概述了构建HRA和将新的实验数据映射到HRA上所需的众多步骤。
Fig. 2: Mapping experimental data to the HRA.
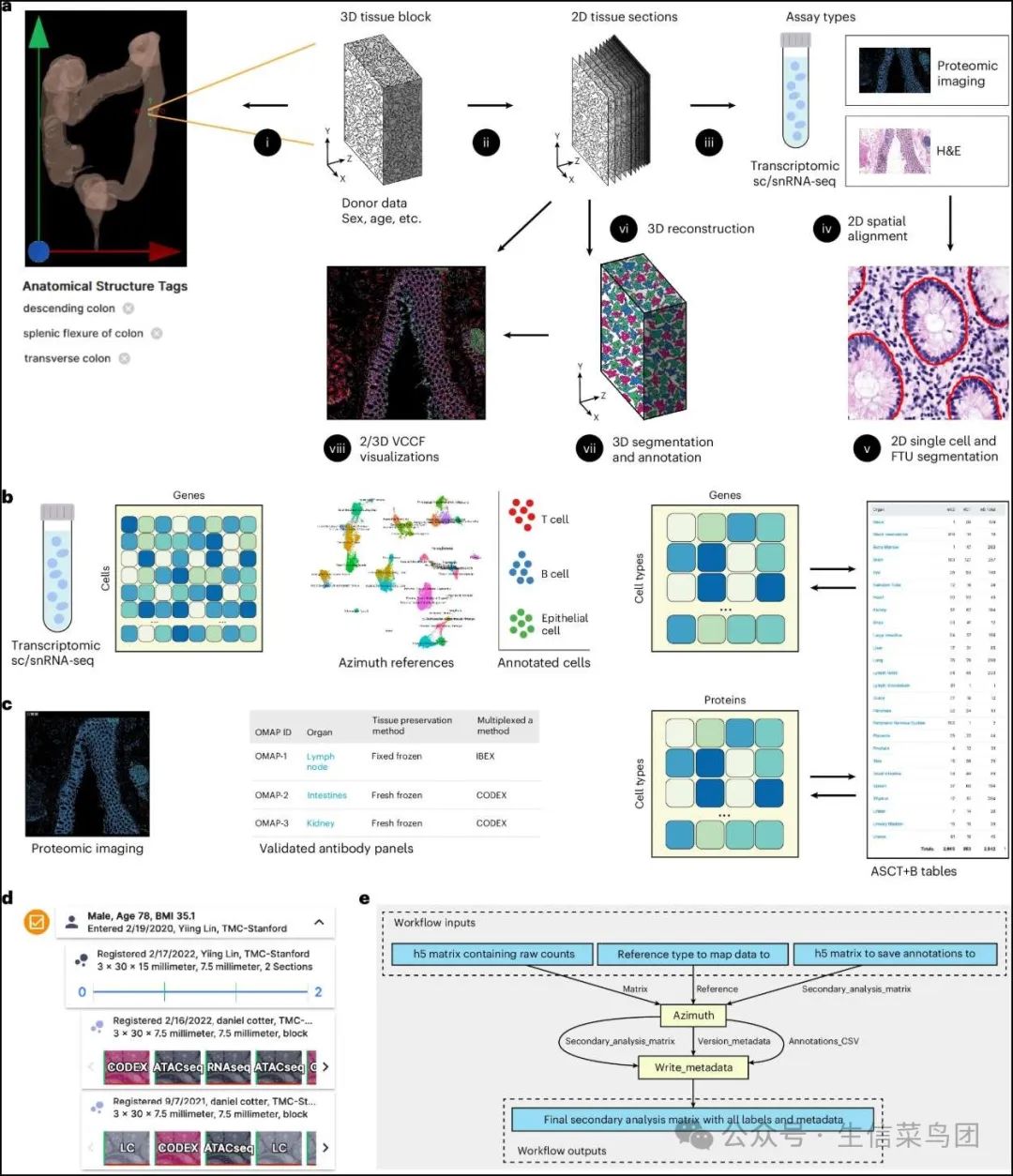
- 图片说明
◉ 一个组织块使用RUI或millitome(i)进行3D空间注册和语义注释。一个较小的部分组织块可能用于sc/snRNA-seq分析(未显示)或切成组织切片(ii)。组织切片使用一种或多种测定类型进行分析(iii)。例如,单细胞转录组学(例如,sc/snRNA-seq),OMAP对齐的空间蛋白质组学(例如,CODEX和Cell DIVE)以及高分辨率苏木精和伊红(H&E)染色的组织学图像。不同测定类型的组织切片进行空间对齐并不简单(iv)。H&E数据被用来通过训练的机器学习模型分割FTU(v)。通过计算将来自多个连续组织切片的数据进行对齐,完成组织体积的3D重建(vi),然后进行3D分割和注释(vii)。二维或三维数据被分析以识别不同类型细胞到血管的距离(VCCF可视化),作为一个多尺度CCF,在此距离内没有其他细胞非常遥远(viii)。◉ 单细胞/细胞核数据(sc/snRNA-seq)存储为细胞-基因矩阵;细胞类型使用Azimuth或其他细胞类型注释工具进行注释;结果汇总为细胞类型-基因生物标志物表达值矩阵,这些矩阵与ASCT+B表格对齐;并在各种HRA用户界面(例如,EUI和FTU探索器)中使用。◉ 使用经过验证的抗体面板链接到AVRs生成的OMAP对齐的空间数据被分析,以计算蛋白质生物标志物表达值矩阵,该矩阵通过半自动化工作流程与ASCT+B表格对齐。◉ EUI为供体(性别、年龄和身体质量指数)、数据提供者(上传日期、联系人姓名和隶属关系)、组织块和切片(尺寸、数量、日期和RUI注册的联系信息)以及数据集(测定类型)提供完整的来源,并链接到HuBMAP数据门户、其他数据门户或出版物中的原始数据。◉ CWL工作流详细说明了哪些工具(黄色)运行在哪些输入/输出数据(蓝色)上。显示的是Azimuth细胞类型注释工作流。
Para_02
- HuBMAP 联盟为多种数据类型开发了统一的计算处理管道:单细胞(sc)/单核(sn)RNA测序、sc/snATAC测序、基于多重抗体的空间蛋白质组学(CODEX(最近更名为PhenoCycler)和Cell DIVE)、多重离子束成像(MIBI)、Slide-seq和Visium测序空间转录组学以及荧光原位杂交空间转录组学等。
- HuBMAP 计算管道全部是开源的,并在GitHub上作为CWL工作流发布,包装了Docker镜像中的工具(也可以通过Singularity执行),补充数据(基因组索引/注释和深度学习模型)被构建到发布的Docker镜像中,以实现便携性和可重复性。
Para_03
- HuBMAP单细胞/单核RNA测序流程(https://github.com/hubmapconsortium/salmon-rnaseq,也用于空间转录组学测序如Slide-seq和Visium)基于Salmon准映射方法,并对内含子和外显子序列进行基因表达定量,后续分析使用Scanpy进行,并通过scVelo计算RNA速度。
- sc/snRNA-seq流程的输出使用Azimuth细胞类型注释工具的自动化版本进行注释;这些目前包括心脏、肺和肾脏,随着新的Azimuth参考集成到HuBMAP处理基础设施中,还将计算附加注释。
Para_04
- HuBMAP 成像管道(方法)是端到端的分析方法,接受原始图像,必要时执行照明校正、背景减除和拼图缝合,然后进行细胞和细胞核分割,并将表达和分割掩膜图像写入多通道 OME-TIFF 文件。
- 通过空间过程和关系建模(SPRM)进一步处理表达和掩膜图像,该过程使用 CellSegmentationEvaluator 工具计算图像和分割质量指标,创建细胞相邻性映射,为每个细胞和细胞核计算特征。
- 对细胞、细胞核和图像像素进行无监督聚类,计算每种聚类类型区分一个簇与其他簇的生物标志物,并将结果写入 CSV 和 HDF5 格式供终端用户和 HuBMAP 数据门户使用。
Para_05
- 为了构建HRApop(图1g),我们整合了来自HuBMAP的445个公共数据集,SenNet的两个数据集,CZ CELLxGENE的两个集合中的91个健康数据集(包括‘成人心脏细胞’和‘LungMAP——来自广泛年龄健康捐赠者的数据’)以及GTEx的15个单细胞数据集(方法部分)。结果,基于单细胞转录组学(例如sc/snRNA-seq)和OMAP对齐的空间蛋白质组学(例如CODEX和Cell DIVE),我们获得了23个器官中40个解剖结构的细胞类型群体数据,涉及13个独特的Uberon ID。
- 三个器官(大肠、小肠和皮肤)的数据是通过转录组学和蛋白质组学数据计算得到的细胞类型群体。
Para_06
- 对于 HRAlit32(图1h),映射到了HRA中的DOs的专家有583,117人,出版物有7,103,180篇,资助项目有896,680个,实验数据集有1,816个(方法)。
User interfaces
用户界面
Para_01
- HuBMAP门户(https://hubmapconsortium.org;补充图2)介绍了HuBMAP的目标,并链接到实验和图谱数据、工具和培训材料。
- HuBMAP数据门户(https://portal.hubmapconsortium.org)支持实验数据的摄入、搜索、探索和下载。
- HRA门户(https://humanatlas.io;补充图3)支持HRA数据的构建、访问、探索、使用和下载。
Para_02
- ASCT+B 报告器 3(https://humanatlas.io/asctb-reporter;补充图 4)支持人体器官专家编写和审查 ASCT+B 表格和 OMAP。
- 存在详细的操作流程(SOPs)45 和视频教程59,60,超过 170 位独特的专家通过使用此工具作为作者和/或审稿人,根据第六版 HRA 相关 DO 中列出的独特 ORCID ID 数量,为 HRA 做出了贡献。
Para_03
- Azimuth14 (https://azimuth.hubmapconsortium.org; 补充图5) 是由 HuBMAP 开发的,用于自动化处理、分析和解释 sc/snRNA-seq 和 ATAC-seq 数据。
- 它的基于参考的映射管道读取细胞-基因矩阵,并执行标准化、可视化、细胞注释和差异表达(生物标志物发现)分析(图1f和图2b)。
- 结果可以在应用程序内探索或下载以进行额外分析。
- 在 HuBMAP 中,Azimuth 以生产模式使用,以自动注释 sc/snRNA-seq 数据集。
- 存在跨表来关联 Azimuth 细胞类型与 ASCT+B 表术语和本体 ID。
Para_04
- RUI60(https://apps.humanatlas.io/rui;补充图6和SOP61)支持将人体组织块注册到3D CCF,并自动分配基于表面网格级别碰撞事件与Uberon和FMA本体论相关的解剖结构注释。
- 解剖结构注释与ASCT+B表和实验数据相结合,使得预测通常存在于解剖结构和碰撞组织块中的细胞类型成为可能。
- RUI以JSON格式输出记录注册数据(例如,组织块通用唯一标识符(UUID)和三维大小、位置和旋转以及基于边界框的解剖结构注释)以及来源数据(例如,操作员姓名和日期)。
- RUI作为一个独立工具可供任何人使用,以贡献与HRA对齐的空间数据。
- 它已完全集成在HuBMAP、SenNet和GUDMAP数据摄取门户中,但需要身份验证。
Para_05
- 探索用户界面(EUI)(https://apps.humanatlas.io/eui;补充图7)支持全身器官、组织和细胞水平上的组织样本和元数据的可视化浏览(表1,美国3号)。
- 2024年1月,来自351名捐赠者和19个财团/研究的901个人体组织切片和4,221个数据集被RUI注册到HRA 3D CCF。
- 用户可以根据捐赠者人口统计信息(例如性别和年龄)或数据来源(例如财团/研究和技术)进行筛选。
- 他们可以搜索特定的解剖结构、细胞类型或生物标志物来探索与解剖结构相碰撞的组织切片数量,以及位于这些解剖结构中的细胞类型或其特征性生物标志物(根据ASCT+B表格)。
- 用户还可以使用可调节的探测球运行三维空间搜索,在右侧按需探索详细信息,并链接到Vitessce62,63可视化工具在HuBMAP数据门户中,以及链接到其他数据门户中的数据和工具。
- 包含所有HRA数据的EUI作为一个独立工具可用,支持探索已映射到HRA的所有实验数据。
- EUI针对HuBMAP、SenNet和GTEx数据门户进行了定制、品牌化和完全集成,以支持财团特定数据的探索(补充图8)。
Para_06
- Vitessce62,63(http://vitessce.io)是一个用于可视化探索实验数据的工具,Azimuth 参考(补充图 5),HRA 分割和注释或细胞间距离分布可视化(补充图 9),参见大图使用部分的预览。
- see previews in the Atlas usage section.
Para_07
- 交互式FTU探索者64(https://apps.humanatlas.io/ftu-explorer;补充图10)支持在它们的二维空间环境中探索细胞类型以及平均生物标志物表达矩阵(表1,US 4)。例如,组织数据(包括细胞类型群体和基因或蛋白质表达水平的数据)可以与健康的HRA参考数据进行比较,以确定细胞数量、细胞类型或平均生物标志物表达值的差异,从而为临床决策提供信息。
- 这有助于确定细胞数量、细胞类型或平均生物标志物表达值的差异,从而为临床决策提供信息。
Para_08
- HRA Organ Gallery65,66(https://github.com/cns-iu/hra-organ-gallery-in-vr; 补充图11)支持对HRA 2.0的65个三维参考对象中的1,192个解剖结构进行多尺度探索。
- 使用Meta Quest VR设备,用户可以选择男性或女性参考身体;然后他们可以选取特定器官并用双手进行探索。
- 为了实现每秒60帧的视图更新率,使用了从原始HRA 3D参考对象派生的较低细节层次的模型。
Para_09
- HRA API(https://humanatlas.io/api/;补充图12-14)支持对所有HRA DOs以及实验性HRApop数据进行编程访问。
- 用户首先选择一个API服务器和路由,输入查询参数,然后查看查询响应,请参阅方法部分以获取详细信息。
- HRA 仪表板(https://apps.humanatlas.io/dashboard)将 HRA、出版物和实验数据与世界人口数据进行比较。
- 补充图 15a 显示了 HRA 调查受访者和组织数据捐赠者按年龄组划分的人口金字塔,以及与世界人口的比较,还包括 HRA 专家和出版物作者的职业年龄段人口金字塔。
- 补充图 15b 展示了调查受访者、HRA 组织捐赠者、HRA 专家、论文作者和世界人口的种族构成,以百分比表示。
- 补充图 15c 中的颜色地图显示了覆盖在世界地图上的论文作者数量。
- CCF-HRA 数据仪表板有助于了解 HuBMAP 数据中有多少已被 RUI 注册(https://hubmapconsortium.github.io/hra-data-dashboard)。
Documentation and instructional material
文档和指导材料
Para_01
- 2024年1月,HuBMAP数据门户提供了8篇出版物及其相关数据集的访问权限,50多份技术文档(https://software.docs.hubmapconsortium.org/technical)以及指向protocols.io上的235个实验协议的链接;HRA门户链接到20个标准操作程序(https://zenodo.org/communities/hra)和可见人体大规模开放在线课程(VHMOOC;https://expand.iu.edu/browse/sice/cns/courses/hubmap-visible-human-mooc),该课程包含39个视频、4个自测题和3个测验、2个实践教程,以及入门和结业调查(补充图16)。
Previews of Atlas usage
Atlas使用预览
Para_01
- 两个示例预览展示了在HuBMAP开发的地图数据和代码如何用于深入了解病理学,并参见推动HRA构建和使用的用户故事(表1)。
- 所有数据和代码在GitHub上公开获取67,68,并在Dryad上公开获取69。
- 细胞距离分布代码可通过HRA门户70获得,以支持表1和用户故事5。
- 通过Dryad发布了关于人类肠道的CODEX多路复用成像数据集的细胞类型注释。
- 计划在未来版本中将全部数据和代码集成到HuBMAP数据门户的工作流程中。
Perivascular immune cells in lung
肺部血管周围免疫细胞
Para_01
- 正常的肺功能依赖于气流与血流的精确匹配以实现正常的气体交换。
- 免疫细胞的异常存在和活动导致血管膜渗漏和水肿,这会使气体交换膜增厚,并且气道内黏液和细胞碎片的积聚会导致气流与血流之间的不匹配。
- 持续的炎症最终会导致纤维化。
- 先前使用单细胞RNA测序数据和CellTypist通用参考数据集的工作发现了以前未被充分认识的器官特异性特征以及T细胞和B细胞的聚集。
- 最近在粘膜免疫领域的出版物展示了人类肺组织中免疫细胞在聚集中的分离及其在异常调节血管功能中的作用。
- 包括纤维化和富含免疫细胞区域在内的分子和细胞变化最近在患有支气管肺发育不良(BPD)的儿童肺部被成像出来,这是一种发生在早产后的慢性肺病。
- Vitessce工具在图3a(左)中用于可视化组织数据。
- 细胞距离分析和可视化用于比较地可视化和量化健康成人和BPD肺部特定区域的细胞密度,以展示相对于最近的血管内皮细胞核,多种细胞类型相对接近程度的评估,使用单细胞空间蛋白质生物标志物。
Fig. 3: Human Reference Atlas usage.
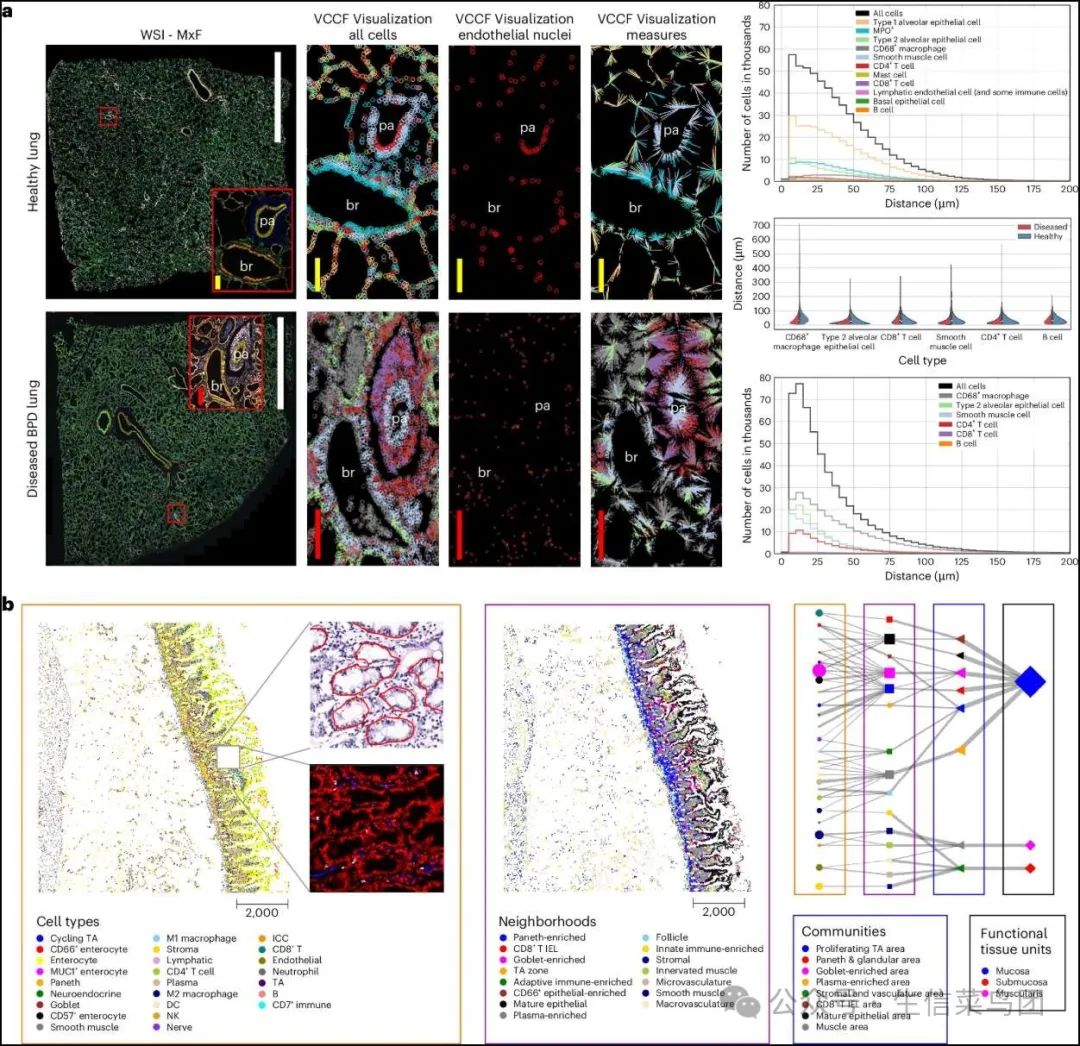
- 图片说明
◉ HRA可以用于比较包括内皮细胞、上皮细胞和肌肉在内的组成血管、气道和进行气体交换的功能性肺结构的实质细胞以及包括巨噬细胞在内的驻留免疫细胞与局部血管(VCCF可视化)在健康(顶部)和患病(底部)肺中的分布情况,使用多重免疫荧光显微镜图像,其中包含细支气管(br)和伴随的小肺动脉(pa)。标尺:白色5毫米;红色200微米;黄色100微米。右侧的图表显示了健康肺(顶部)和患病BPD肺(底部)中存在细胞类型的距离分布;中间的小提琴图显示了在两个数据集中常见的细胞类型的距离分布对比。数据集可在GitHub81上获取。◉ 多级细胞邻域可以计算出来,用于分析和传达FTU的结构和功能;左侧展示了带有细胞类型注释的组织图像以及H&E染色放大图像和FTU分割(红线框出)的放大图像,中间展示了邻域,右侧展示了FTU、邻域、社区和细胞类型的层次结构。数据集可在GitHub81上获取。◉ ICC,卡哈尔间质细胞;TA,过渡增殖细胞;NK,自然杀伤细胞;DC,树突状细胞;IEL,上皮内淋巴细胞。
Para_02
- 展示了应用于健康肺组织(上排,28种抗体面板)和BPD组织(下排,25种抗体面板)的PhenoCyclerR多重免疫荧光分析法(WSI-MxF)的全片显微镜图像。
- 使用数字缩放突出显示了关注的相似区域(红框,MxF-ROI),聚焦于细支气管(br)和伴随的小肺动脉(pa)。
- 在BPD肺部,注意到两种结构周围有免疫细胞聚集,主要是CD3+淋巴细胞。
- 为了评估血管CCF(VCCF)以定位免疫和其他肺细胞,将细胞类型用特定颜色掩码(见图例),并测量到最近内皮细胞核的距离(红圈)。
- 细胞到最近内皮细胞的测量结果显示为由细胞类型着色的辐条。
- 简而言之,图形表示定量地表明细胞密度增加,主要为CD4+,但不是CD8+淋巴细胞,以及髓系免疫细胞,这些细胞位于病变肺部血管附近的密切位置。
- VCCF可视化显示,在病变肺部与健康肺部相比,内皮细胞嵌入围绕肺动脉(pa)聚集的淋巴细胞群中。
- 对细胞群体(每种细胞类型的细胞数量及其平均生物标志物表达值)以及细胞间和细胞到FTU的空间分布模式的分析有助于理解组织和细胞紊乱,这些紊乱导致肺病器官衰竭。
- 在本例中,病变组织的气体交换膜因血管外免疫细胞聚集而增厚,使得细胞距离到最近内皮细胞的分布压缩且夸大(参见图3a右侧的图表)。
- 如所示,在二维图像上的测量提供了新的见解;然而,考虑到复杂的气道和血管分支系统以及肺泡非常薄、高度多细胞的气体交换膜,细胞分割和确定肺部相对位置特别具有挑战性。
- 预计类似分割、细胞间和细胞到FTU测量应用于三维肺组织体积将识别目前未被充分认识的健康和疾病中的关系(表1,US 5)。
- HuBMAP代码可用于人类肺组织,了解特定免疫细胞浸润的空间组织如何与疾病病理生理学相关,揭示针对人类疾病的靶向治疗潜力。
- 代码和数据可在参考文献68中获得。
Hierarchical cell type populations within FTUs
FTU内的层级细胞类型群体
Para_01
- FTU分割算法用于组织学数据和配对空间数据的分层细胞群分析可以结合起来,以分析和传达FTU在各个尺度上的结构和功能(详见方法部分)。
- FTU分割算法作为标准HuBMAP工作流程的一部分运行(目前仅限于肾脏中的肾小球,很快将扩展到大肠中的隐窝和脾脏中的白髓)。
Para_02
- 我们举例说明了一种以前为分析跨尺度的细胞类型邻域而开发的分层细胞邻域分析方法,并将其应用于健康的人类肠道(参见Github上的Jupyter Notebook)。
- 我们将这些尺度命名为:细胞‘邻域’、‘社区’和‘功能组织单元’。
- 在不同尺度上计算相似的细胞邻域、社区和组织单元类似于我们认为人们形成邻里、城市和州的方式。
Para_03
- 目前,HuBMAP数据门户支持基于抗体的多重成像数据的细胞分割,但缺乏对这类数据集进行细胞类型注释的能力。这一功能正在积极开发中(方法)。因此,为了展示这个用户故事,使用了单独处理过的肠道数据79(包含细胞类型注释)。使用Van Valen实验室当前开发版本的细胞类型模型对同一数据集的细胞类型预测也可以通过https://cns-iu.github.io/hra-construction-usage-supporting-information获得。该模型版本是在多个跨越组织类型和多重成像模式的数据集上训练的。与预测数据一起还包括使用Mesmer生成的细胞分割掩膜。
Para_04
- GitHub67上的Jupyter Notebook演示了如何读取之前发布的关于人类肠道的CODEX多重成像数据集69,识别细胞类型如何对应更大的多细胞结构,并支持探索这些高级别细胞邻里关系之间的联系。
- 通过在组织坐标中可视化数据,我们可以观察到潜在的分层或一致的FTU结构,例如小肠近端空肠中的肠隐窝的重复结构(见图3b,左)。此外,我们可以在不同级别的细胞邻里关系上量化这些关系,并将它们表示为网络图(图3b,右),其中线条的粗细表示下一级别中的细胞百分比。
- 请注意,组织样本和图表使用完全相同的节点颜色编码和命名。
Usage statistics
使用统计
Para_01
- 2023年7月至2024年10月期间,超过33,500名独立用户访问了HuBMAP联盟网站(https://hubmapconsortium.org)。
- 这些用户访问了480个不同的页面;访问次数最多的前六个来源分别是Google、pathwaystoscience.org、nature.com、Bing、X/Twitter和psc.edu。
- 2021年1月至2023年12月期间,87,310名独立用户访问了HuBMAP数据门户(https://portal.hubmapconsortium.org)中的382,384页;访问次数最多的前五个来源分别是nature.com、hubmapconsortium.org、humancellatlas.org、azimuth.hubmapconsortium.org和humanatlas.io。
- Azimuth支持上传了27,000个数据集,并对超过366,000,000个细胞进行了类型注释。
- 2023年6月至2024年10月期间,HRA门户请求完成了1,194,130次,HRA API请求完成了524,358次;访问次数最多的前五个来源分别是HuBMAP实体API、GTEx门户(https://gtexportal.org)、HuBMAP数据门户、SenNet数据门户(https://data.sennetconsortium.org/search)和EMBL-EBI(https://www.ebi.ac.uk)。
- 通过NIH3D网站,3D参考对象被访问了3,065次。
- HRA OWL文件通过NCBI生物门户本体浏览器(https://bioportal.bioontology.org/ontologies/CCF)被访问了1,325次,通过EBI OLS本体浏览器(https://www.ebi.ac.uk/ols/ontologies/ccf)被访问了11,531次。
- 共有310名学生注册了VHMOOC,并花费了5,652小时浏览材料、进行自我测试以及参与实践社区。
Discussion
Para_01
- 这篇资源论文描述了对多尺度人类表型组架构(HRA)的构建和使用具有广泛实用价值、兴趣和重要意义的数据、代码和工具。
- HRA 努力和不断发展的数据和代码基础设施在多个方面是新颖且独特的:
- (1)HRA 集成了从整个人体到单细胞水平的多种测定类型。
- (2)它提供了标准操作程序(SOPs)和工具,将来自65个器官的人体组织空间上和语义上地注册到一个共同的脑图谱(CCF)中。
- (3)它将解剖结构、细胞类型和生物标志物链接到本体论,并在需要时扩展现有的本体论。
- (4)HRA 提供了多样化的接口,允许用户探索和检查各种HRA DOs(三维参考对象、ASCT+B表格、OMAP等)、实验数据以及参与联盟的文档,特别是HuBMAP数据(HRA门户、ASCT+B报告器、RUI、EUI、细胞间距离分布可视化、交互式FTU浏览器和HRA器官画廊在虚拟现实中的应用)。对于每个用户界面,我们提供了补充图3、4、6、7和9-11),包含高分辨率截图和详细注释。
- (5)HRA 开发是社区驱动和协作的;每月工作组会议为战略决策提供信息;由30多个团队开发的50多个开源算法被系统性地整合进一个灵活且适应性强的系统架构中,为许多图集项目增添了价值;新的HRA数据和代码每六个月通过HRA门户和本体服务向公众发布。
- 由此产生的HRA是一种多尺度、多模态、三维数字化产品,统一了跨器官、解剖结构尺度、人口统计学标记、测定类型的生物医学知识,并将其与本体论链接起来,使人类参考数据可计算。
Para_02
- 第六次发布的HRA存在几个已知的局限性,这些问题将在未来的版本中解决。从第八次发布(于2024年12月中旬发布)开始,所有HRA DO及其完整的来源都被包含在HRA知识图谱中。
- 目前,CL中尚未捕捉到细胞状态,也未涵盖由单细胞技术产生的特定细胞类型;然而,HRA已经开始使用具有‘CL本体术语:细胞状态或特异性术语’格式的细胞类型注释(例如,‘静息的胰腺星状细胞’映射到‘胰腺星状细胞 CL:0002410’,该术语在CL中或‘MUC1阳性的肠上皮细胞’映射到‘肠上皮细胞 CL:0000584’),以及一个与CL匹配的置信度术语(例如,skos:narrowMatch(用于细胞状态或新的细胞类型)或skos:exactMatch(用于与CL中的细胞类型完全匹配的情况)),以便它们能够在HRA知识图谱和UBKG中表示,并在计算社区确定如何本体地表示此类细胞的方法后进行更新。
- 当没有完全匹配时,HRA知识图谱中的术语将被赋予ASCTB-TEMP ID,而通过细胞类型注释模型注释的单个细胞将被赋予细胞类型ID,以便在未来可用时促进后续更新。
Para_03
- 此外,现有的将新实验数据映射到HRA的工作将在三个方面得到扩展:(1) HuBMAP计划添加多个新的Azimuth参考(例如,用于大肠和小肠),并更新现有的参考(例如,用于肾脏和肺部),以便通过交叉引用捕获具有额外/修订细胞类型注释和CL术语及CL ID的新数据;(2) 在第七个HRA发布版本中发布了八个新的OMAP,并且第八个HRA发布版本中还有更多的OMAP正在进行中,这大大增加了可以映射到HRA的空间数据集数量;(3) 从第八个HRA发布版本开始,新的三维器官将被添加到RUI:股四头肌和小腿三头肌骨骼肌、食道和淋巴管。
- ,
Para_04
- 目前,HRA知识图谱和API驱动了HuBMAP、SenNet、GUDMAP、GTEx数据门户以及CZ CellGuide中的不同2D和3D用户界面。
- 根据美国6(见表1),我们开始开发额外的轻量级网络组件,以便于访问HRA数据,并在其他网站上展示HRA功能(https://apps.humanatlas.io/us6)。
- 此外,我们正在实现各种HRA仪表板(美国7;https://apps.humanatlas.io/dashboard),以传达HRA执行的操作;用于HRA构建的实验数据(完整的来源);现有本体如何扩展以捕捉健康人类术语和关联;谁在使用HRA数据、工具和API;以及该地图集的代表性如何。
Para_05
- 最后但同样重要的是,我们将扩展HuBMAP数据门户和HRA门户之间的互连。
- 具体来说,我们将把新的HRA发布物纳入HuBMAP UBKG,以便在HuBMAP数据门户中支持的解剖结构、细胞类型和生物标志物与现有的本体论和三维空间参考框架保持一致。
- 随着HuBMAP团队开始编译三维数据集,需要比较现有的算法来支持多个后续组织切片的空间对齐,这类似于二维细胞分割方法的做法51,52。
- 预计三维数据将显著提高HRA的质量和预测能力。
Para_06
- 社区对HRA用户故事、数据、代码、用户界面和培训材料的输入是受欢迎的,鼓励有兴趣了解更多或为HRA工作做出贡献的专家注册参加每月在线工作组活动。
-
Methods
Para_01
- 人类专家生成的数据和实验组织数据被用来构建HRA(图1)。
- 新的实验数据通过(1)三维空间注册;(2)使用悬浮基(例如,sc/snRNA-seq);或(3)空间(例如,CODEX83、Cell DIVE84、IBEX85、86、成像质谱流式技术87和其他多路复用、基于抗体的蛋白质成像平台)与HRA对齐的检测类型映射到HRA(图2)。
Expert-generated data
专家生成的数据
ASCT+B tables
ASCT+B表格
Para_01
- ASCT+B表格(https://humanatlas.io/asctb-tables;图1a)是由专家使用ASCT+B报告员(补充图4)根据标准操作程序汇编的。
- 请注意,脑部ASCT+B表格的独特之处在于它是通过共同细胞类型命名方法计算得出的,该方法将关键的细胞类型特征(例如,脑区和皮层层)、广泛的细胞类型类别和基因生物标志物信息串联到注释中。
- 这些标准操作程序编号为88。
- 这种方法是基于共同细胞类型命名方法89进行的。
Para_02
- 从第六版HRA开始,新的和修订的表格列出了CL中存在的细胞类型父母,这些细胞类型大约有600种,目前拥有ASCTB-TEMP ID(临时本体术语和ID),因为它们尚不存在但通过HRA努力系统地添加到CL中。
- 这使得在CellGuide(https://cellxgene.cziscience.com/cellguide)和其他工具中展示完整的细胞分类成为可能。
- 例如,补充图17展示了CZ CellGuide对神经元(CL:0000540)的可视化,显示了CL本体分类,其中'神经元'细胞类型用绿色突出显示,连同它的父类('神经细胞',这是'体细胞',而体细胞是'动物细胞')以及子节点(例如,'GABA能神经元'和'谷氨酸能神经元')。
- 交互式可视化位于https://cellxgene.cziscience.com/cellguide/CL_0000540。
Para_03
- HuBMAP特别关注的是开发详细的血液血管ASCT+B表格,以支持VCCF(https://humanatlas.io/vccf)。
- VCCF中包含的相关数据包括血管及其分支关系,以及相关的细胞类型和生物标志物、血管类型、吻合口、门脉系统、微血管、FTU、与三维参考对象的链接、血管几何形状(长度和直径)以及与血管供应或引流的解剖结构的映射。
2D and 3D reference objects
2D和3D参考对象
错误!!! cannot unpack non-iterable NoneType object
Para_02
- ASCT+B 表格在第六版 HRA 发布中包含了 4,499 个独特的解剖结构和 1,195 个细胞类型的与本体对齐的术语。
- 对于其中一些解剖结构和细胞类型,存在解剖学对齐、空间明确的参考对象。
- 具体而言,有 10 个器官中的 22 个 FTU 的 2D 图解,包含 116 种细胞类型的 3,742 个细胞。
- 此外,还有 65 个 3D 参考对象(包括男性和女性、左右器官)中的 1,192 个解剖结构,具有 516 个独特的 Uberon ID,以及 37 个独特的 Uberon ID。
- 交叉表将每个 2D/3D 解剖结构和细胞类型与其在 ASCT+B 表格中的相应术语关联起来(参见 SOP 部分)96。
Segmentation masks
分割掩码
Para_01
- 不同的工具被用于支持人工专家对图像的手动分割(将图像中的每个像素分配给一个对象,如单个细胞、FTU或解剖结构)。
- 在HRA努力中,器官专家使用QuPath工具生成FTU和血管的二维分割掩膜(见SOP)。
- DeepCell Label(https://label.deepcell.org)被用于获取单细胞的二维分割掩膜。
- 由此产生的‘金标准’分割和注释数据(图1d)被用来训练机器学习算法,以便实验数据集可以自动分割(图2a(v))。
OMAPs and AVRs
OMAPs和AVRs
Para_01
- OMAPs(https://humanatlas.io/omap)是为特定的样本保存方法和多路复用成像技术设计的抗体集合,旨在允许对它们被验证的组织中存在的解剖结构和细胞类型进行空间映射(图1e)。
- OMAPs是湿实验室验证的抗体,专家最初通过使用文献、可用的抗体搜索引擎以及可能还有ASCT+B报告器(补充图4和SOP101)来识别它们作为多路复用基于抗体的成像实验的候选物。
- OMAP中的抗体链接到专家生成的HuBMAP AVRs(https://avr.hubmapconsortium.org和SOP102),这些AVRs提供了关于个体抗体用于多路复用基于抗体的成像测定的表征细节。
- 抗体验证既昂贵又耗时,因此这些资源旨在帮助其他研究人员取得成功,并减少多路复用基于抗体的成像研究所需的时间和资金。
Cell annotation references
细胞注释参考
Para_01
- 大部分单细胞数据是单细胞或单核RNA测序数据。
- 细胞类型注释工具(图1f)如Azimuth14、CellTypist71,103和流行的Vote(popV)104常用于根据基因表达谱对细胞进行聚类,然后根据已发表的基因表达谱将这些Uniform Manifold Approximation和Projection105聚类分配到细胞类型。
- 补充表1显示了这三种工具每种器官可以分配的细胞类型的数量(最右列)——与ASCT+B表格和3D参考对象库中的细胞类型数量相比(中间列);第二列显示了通过HuBMAP、SenNet、GTEx和CZ CELLxGENE数据门户可以获得的数据集数量。
- 请注意,某些器官的数据集(例如,尿膀胱)不存在。
Para_02
- 人类专业知识是编制跨表所需,这些跨表将由这三种工具分配给细胞的标签与CL中的术语关联起来。
- 将细胞标签映射到CL可以部分自动化;然而,如果研究人员提供的标签被写出来而不是作为缩写列出,则这种方法更为有效,因为不同的研究小组不使用标准化的细胞类型缩写。
- 当细胞类型尚未存在于CL中时,自动映射到CL进一步受阻,在这种情况下,通常会使用一个父细胞类型作为占位符,直到可以将确切的细胞类型添加到本体论中。
- 因此,构建使用实验数据支持的最具体细胞类型的跨表是可取的。
- 根据可用于添加单细胞RNA测序发现的新细胞类型的活跃编辑者/馆藏者的数量,新术语的优先级排序和收集支持文献需要时间。
- 由此产生的跨表是器官特异性的,并且它们作为特定于细胞类型的注释跨表发布,将三种工具分配给的任何细胞类型与ASCT+B表格中的相应术语关联起来,请参见示例106。
Experimental data
实验数据
Para_01
- HuBMAP 数据门户(https://portal.hubmapconsortium.org)使用微服务架构(补充图1)通过混合本地和云的方法提供数据和代码,采用联邦身份管理、UUID 和完整的来源信息进行数据管理和数据安全。
- 该架构支持多样化的统一分析管道和交互式探索工具的工作流和容器。
- 这种架构使得可以大规模地摄取数据,根据需要调整元数据格式,增加新的算法和工作流,并确保所有服务在生产阶段的速度和可扩展性。
- 截至2024年1月20日,HuBMAP 数据门户向公众开放了来自213名捐赠者的2332个数据集。
- 其中,360个数据集是sc/snRNA-seq,79个数据集是空间OMAP对齐的数据集。
Tissue collection and RUI registration
组织收集和RUI注册
Para_01
- RUI60(https://apps.humanatlas.io/rui)被实施以支持组织块在HRA CCF中的空间注册;补充图6。
- 它在过程中收集样本ID、捐赠者元数据以及来源信息(谁注册了数据以及何时注册)。
- 具有对组织样本的空间和捐赠者数据了解的专业人士使用RUI来注册他们的组织样本——如有必要,在SOP61的支持下由指定的HRA注册协调员协助。
- 或者,有一种更协作的工作流程,在这种工作流程中,注册协调员在主题专家的指导下更积极地进行注册。
- 这些工作流程在两个专门的SOP中进行了详细说明,解释了如何使用RUI61以及注册协调员的责任107。
- 接下来,注册协调员使用位置处理器工具108将组织样本元数据与匿名捐赠者元数据(性别、年龄、体质指数、种族等)和出版物元数据(DOI、作者、出版年份等)结合起来。
- 一旦样本被注册并且元数据得到丰富,注册协调员将联系主题专家检查准确性和完整性。
- 然后,注册协调员发布经过验证的注册集,使其通过EUI(https://apps.humanatlas.io/eui;补充图7)可访问。
Para_02
- 组织块注册可以通过使用‘millitome’(https://humanatlas.io/millitome)来简化和实现更高的可重复性,这是一种辅助湿实验室科学家以可重复的方式从单一器官切割并注册多个组织块的装置。
- 这种3D打印设备设计用于固定一个新获取的器官,并配备有切割槽,可以引导碳钢刀片进行均匀切割,详见HRA的millitome目录(https://hubmapconsortium.github.io/hra-millitome)以根据供体性别、器官侧别、器官大小和切割间隔访问和定制器官millitome。
- 每个millitome套装包含一个用于3D打印millitome可重现表面几何形状的STL文件,以及一张查找表,将millitome位置与研究团队分配的组织样本ID相关联。
- 使用millitome切割器官后,科学家们会在查找表上记录样本,并提交这些数据供HRA millitome协调员审核。
- 一旦包裹完成,数据将被添加到EUI中,供科学家们审查,以验证组织大小、位置和方向的注册准确性。
- SOPs详细说明了millitome的构建和使用方法。
sc/snRNA-seq transcriptomic data annotation
sc/snRNA-seq转录组数据注释
Para_01
- sc/snRNA-seq转录组学数据集是从四个数据门户使用hra-workflows-runner下载的(https://github.com/hubmapconsortium/hra-workflows-runner)。
- 对于来自HuBMAP和SenNet的数据(每个数据集仅来自一个供体),使用搜索API(HuBMAP,https://search.api.hubmapconsortium.org/v3;SenNet,https://search.api.sennetconsortium.org)来获取所有现有细胞-基因矩阵的H5AD格式文件列表,并下载这些文件以及供体元数据。
- 对于GTEx,从https://gtexportal.org/home/singleCellOverviewPage下载单个H5AD文件。
- 对于CZ CELLxGENE,数据集存储在集合中,一个集合可以包含多个数据集和供体;工作流运行器读取使用CZI Science CELLxGENE Python API(https://chanzuckerberg.github.io/cellxgene-census/python-api.html)编译的所有健康成人数据集索引;它将集合拆分为唯一的供体-数据集对;并使用三种细胞类型注释工具:Azimuth14、CellTypist71和popV104,111(补充表1)处理所有H5AD文件。
- Azimuth(https://azimuth.hubmapconsortium.org)为十个独特器官(肺和扁桃体有一个修订版v.2,在这里使用)提供特定于器官的人类成人参考;对于Azimuth,有针对七个器官(三维空间参考器官不适用于血液、脂肪组织和骨髓)的226种独特细胞类型的HRA交叉映射106。
- 对于CellTypist(https://www.celltypist.org),有针对13个器官和总共214种独特细胞类型的交叉映射。
- 对于popV(https://github.com/YosefLab/PopV),我们提供了针对22个器官和134种独特细胞类型的交叉映射。
- 所有三种工具中共有574种独特的细胞类型。
- 工作流运行器输出四个文件:(1)按细胞类型注释工具划分的所有sc转录组学数据集的细胞摘要;(2)与供体和出版物信息对应的元数据文件;(3)所有sc蛋白质组学数据集的细胞摘要;以及(4)与供体和出版物信息对应的元数据文件112。
- 这四个文件在富集阶段用于构建atlas级别的HRApop数据。
Cell and FTU segmentation for spatial data
空间数据的细胞和FTU分割
Para_01
- 抗体基础的多重成像数据集一旦通过Ingest门户上传到HuBMAP数据门户,就会使用统一的CWL工作流程进行细胞和细胞核分割。
- CODEX数据集(https://github.com/hubmapconsortium/codex-pipeline)的全细胞分割使用Cytokit113和Cell DIVE(https://github.com/hubmapconsortium/celldive-pipeline)进行。
- MIBI数据集(https://github.com/hubmapconsortium/mibi-pipeline)使用Deepcell的Mesmer模型进行处理。
- 使用CellSegmentationEvaluator51对产生的细胞分割结果分配分割质量评分。
- 即将到来的三维空间蛋白质组学数据集的细胞分割由3DCellComposer115与训练过的二维分割器结合提供。
Para_02
- FTU分割在Periodic Acid-Schiff/苏木精和伊红染色的组织学数据集中是通过两个Kaggle竞赛开发的代码进行的。
- 目前的生产流程包括支持肾脏中的FTU,以及当组织学数据集可用时将运行的大肠和脾脏。
Cell type annotation for spatial proteomic data
空间蛋白质组学数据的细胞类型注释
Para_01
- 细胞分割后,使用抗体元数据对CODEX数据集中的标记通道进行空间细胞类型注释,很快将扩展到MIBI和Cell DIVE。
- OMAPs将数据集中的标记面板与ASCT+B表中的细胞类型联系起来。
- SPRM包(https://github.com/hubmapconsortium/sprm)计算各种统计分析,包括所有细胞的平均标记表达。
- Van Valen实验室开发了一种语言信息视觉模型DeepCellTypes116,用于跨组织类型和成像技术分类细胞类型。
- 该模型涵盖了30多种细胞类型,并将在有新的多路复用成像数据可用时进行更新。
- DeepCellTypes可以在https://github.com/vanvalenlab/deepcell-types获取。
- 除了这个模型外,还有不同的团队使用不同的方法注释细胞类型,例如手动标记结合聚类或基于图的网络如STELLAR117。
- Hickey等人69,117,118的肠道数据集使用了手动和STELLAR方法的组合进行注释。
Spatial alignment for 2D multi-omics data
二维多组学数据的空间对齐
Para_01
- 不同分割掩膜的空间结构对齐,参见支持信息中的图2a(iv),用于多组学检测数据分析和/或将空间转录组学数据与苏木精和伊红成像数据对齐可以使用STalign等工具进行。
- 在STalign中,分割后的细胞空间位置被栅格化成图像表示,以便与结构匹配的苏木精和伊红图像对齐。
- 由于组织在数据收集过程中可能会旋转、拉伸或以其他方式变形,因此执行了仿射和微分同胚对齐。
- 这种对齐是通过优化目标函数来实现的,该函数旨在最小化目标图像(栅格化的细胞位置)和源图像(苏木精和伊红图像)之间的像素强度差异,并受到正则化惩罚的约束。
- 所学到的变换被应用于原始分割后的细胞空间位置,将点移动到对齐的坐标空间。
- 这种二维空间对齐有助于下游分子和细胞类型组成比较以及跨技术的整合。
Spatial data 3D reconstruction
空间数据三维重建
Para_01
- 空间对齐多个连续组织切片以支持3D组织块重建(图2a(vi))已使用MATRICS-A120进行,适用于皮肤数据。
- 开发了用于3D组织块重建的附加工具,包括SectionAligner、3DCellComposer和CellSegmentationEvaluator。
- SectionAligner将一系列2D组织切片图像作为输入,分割每个切片中的每一块组织,并将各个部分的切片对齐到一个3D图像中。
- 3DCellComposer使用各种经过训练的2D细胞分割器(如Mesmer)之一,通过CellSegmentationEvaluator自动优化参数设置来将每个3D图像分割成单个细胞。
Atlas-enriched data
富集数据
Mesh-level collision detection
网格级碰撞检测
Para_01
- 提取部位通过专门为高效的空间注册使用网格表面而开发的代码进行后处理。
- 为了提高组织注册过程中的性能,RUI 使用包围盒碰撞检测来确定(近似但快速)的实时交集。
- 为了优化准确性,在富集阶段使用表面网格碰撞检测来确定给定RUI位置与任何解剖结构之间的精确交集体积,这些解剖结构是基于网格级别的碰撞器相交的。
- ‘基于3D几何的组织块注释:组织块与解剖结构之间的碰撞检测’的代码可在GitHub上获取。
- 该API已部署到AWS。
HRAlit
HRAlit
Para_01
- 来自第六次发布的HRA DOs(涉及4,499个解剖结构,1,295种细胞类型和2,098个生物标志物)与7,103,180篇论文相关联,这些论文关联到583,117位作者、896,680个资助项目和1,816个实验数据集。
- 由此产生的HRAlit数据库作为网络包含21,704,001条记录,8,694,233个节点和14,096,735个链接。
- 该数据库已被挖掘以识别主要专家、重要论文、资金趋势或与现有本体的一致性,从而支持系统的HRA构建和使用。
- 所有数据和代码位于https://github.com/cns-iu/hra-literature。
HRApop
HRApop
Para_01
- HRApop提供了特定细胞类型存在的实验数据证据以及3D参考模型存在的数据集和解剖结构的平均生物标志物表达值。
- 在HRA的第六次发布中,有1,192个解剖结构属于516种类型(独特的Uberon ID)来自65个器官(包括男性/女性和左侧/右侧)。
Para_02
- 实验数据集在用于HRApop构建时必须满足三个标准:它们必须(1)使用RUI进行空间注册;(2)具有细胞类型群体数据(例如,可以通过CTann工具(补充表1)注释的H5AD文件或通过蛋白质组学工作流程注释);(3)来自具有质量保证/质量控制的数据门户,或者已经在同行评审的论文中发表。
- 没有额外的修饰语句。
Para_03
- 为了构建HRApop v.0.10.2,我们从四个数据门户下载了9,613个H5AD单细胞转录组学数据集:HuBMAP、SenNet、CZ CELLxGENE和GTEx。
- 恰好有5,118个H5AD数据集是健康的,并且可以使用Azimuth、CellTypist或popV进行注释(补充表1)。
- 此外,我们从HuBMAP下载了两篇论文中发布的74个单细胞蛋白质组学数据集(论文编号69、120)。
- 总的来说,553个数据集(其中479个sc/snRNA-seq转录组学和74个空间蛋白质组学数据集)满足了三个标准,并被用于HRApop的构建。
Para_04
- HRApop v.0.10.2版本通过为具有RUI注册提取站点和细胞类型群体的数据集进行预测验证和优化。
- 随后,它被用来预测2,004个HuBMAP、166个SenNet和4,789个CZ CELLxGENE数据集中缺失的细胞类型注释或空间起源信息。
VCCF distances and Vitessce visualizations in 2D
VCCF距离和Vitessce二维可视化
Para_01
- 为了构建一个VCCF90,91,92,已经开发了一段代码来测量和绘制不同细胞类型到血管细胞类型的距离(见SOP124)。
- 距离图可以在组织切片上叠加使用Vitessce62,63用于二维数据,并通过自定义代码120,125在二维和三维中进行探索;图2a(viii)和3a提供了示例。
- 此外,还提供了一个新工具Cell Distance Explorer,用于可视化二维和三维数据集中的细胞间图和距离分布(https://apps.humanatlas.io/cde)。
Hierarchical community analysis of cell types
细胞类型的层级社区分析
Para_01
- 分层社区分析使细胞类型自动检测多级功能性转换单位成为可能。
- 该方法使用来自空间数据集的单细胞标签和x、y坐标。
- 对于本文展示的预览示例(图3b),数据集是健康人肠道的CODEX多重成像数据集。
- 原始的多重成像数据经过分割、归一化,并使用抗体标记物的z标准化以及Leiden无监督聚类进行聚类。
- 通过深度学习算法(STELLAR)将细胞类型传播到附加样本,用于空间单细胞数据的细胞类型标签传递。
Para_02
- 一旦分配了细胞类型标签,就会通过聚类每个细胞周围的最近邻(n = 10)向量来计算细胞邻域。
- 使用类似的策略,通过将邻域作为标签,并采用更大的窗口大小进行最近邻搜索(n = 100),可以识别较大的结构(称为社区69)。
- 同样地,为了识别主要组织单元,使用社区标签,并在聚类向量之前采用更大的最近邻窗口(n = 300)。
- 一旦识别出所有组织结构,可以通过网络图连接和可视化来自不同层次组织结构的主要成分之间的连接。
- 目前,每个节点按级别组织,并连接到下一个空间层(例如,从细胞类型到邻域,再到社区)。
- 该代码已存放在GitHub67上。
Atlas validation
Atlas验证
Para_01
- 每个HRA中的DO都通过人工专家审查或算法手段进行验证。
- HRA DO数据格式根据类型不同而不同:ASCT+B是以CSV格式存储的,3D参考器官以GLB格式存储,2D FTU以可缩放矢量图形存储等。
- 当这些数据被规范化为LinkML格式时,源数据会被处理,并且原始数据中的结构错误会被识别。
- 一旦数据被规范化,LinkML将用于验证转换后数据的结构,包括确保数据类型和URL符合可接受的标准。
- 这一步可以捕获基本错误,包括格式不正确的URL、缺失的数据以及可能导致下游问题的错误数据类型。
- 除此之外,某些特定类型的DO还会经过更高级的语义检查,以确保所使用的本体术语确实存在,并且DO中的断言也出现在可信的本体中,如Uberon和Cell Ontology。
- ASCT+B表的验证最为严格,涉及EBI团队的详细审查和报告。
- 在编写这些表格时,新/更新的术语和关系会在Ubergraph的最新本体版本中发布(例如第六次HRA中的Uberon 2024-01-18和CL 2024-01-05),并且每周都会在https://hubmapconsortium.github.io/ccf-validation-tools/生成报告,以帮助表格作者获得最高质量的数据供HRA使用。
Flexible hybrid cloud microservices architecture
灵活的混合云微服务架构
Hybrid cloud
混合云
Para_01
- IEC 开发了一种混合云基础设施,利用本地和公共云资源的独特优势——每一种都在强大的可扩展存储旁边配备了强大的可扩展计算——提供了灵活地主动适应不断发展的技术的能力,并响应 HuBMAP 联盟和更广泛的测绘社区的需求。
- 作为这一战略的关键部分,HIVE IEC 在 PSC 存储、处理和归档了 HuBMAP 数据。
- 这种方法提供了灵活的访问方式,因为 HuBMAP 数据的主要副本可以以低成本存储在本地,但随后可以在任何公共或本地资源上可用,而不必承担重大的行业标准数据出口费用。
- 此外,它还提供了免费且低摩擦的访问,因为研究人员无需创建公共云账户即可运行基本分析,或者通过访问与 PSC 国家超级计算基础设施共址的完整 HuBMAP 数据库来运行更大的分析,该数据库免费提供给研究界使用。
Microservices architecture
微服务架构
Para_01
- HuBMAP微服务架构(补充图1)是通过基于用户中心设计的敏捷开发实践构建的,微服务之间通过REST API128使用Docker编排在AWS和本地资源上进行通信。
- 每个微服务专注于提供特定的功能。
- 服务被打包到单独的Docker容器中。
- 这种Docker容器的编排在开发、测试和生产实例中定期构建和重建,这允许独立操作和监控。
- 这种微服务架构支持实验数据摄取、注释、分割、搜索、过滤和可视化所需的一系列不断发展的算法的插拔式功能,以及图谱构建和使用。
- 补充图1展示了资源、API和应用程序层,包含示例模块(补充信息网站显示了一个交互版本,用户可以点击任何模块来访问详细信息)。
- 依赖于其他服务的核心服务是实体API,它由Neo4j图形数据库支持,提供所有与HuBMAP数据相关的来源和元数据信息的存储(创建、检索、更新和删除)。
- 搜索API允许通过AWS托管的OpenSearch搜索引擎搜索所有来源和元数据,该引擎保存了实体API维护的所有信息的副本。
- HuBMAP的身份验证和授权模型利用Globus Auth服务(https://globus.org),登录服务符合OAuth2标准(https://oauth.net/2),提供可以在服务之间传递的用户令牌。
- 这些令牌可以在中央进行验证,并通过Globus组服务与定义的组链接,提供用户授权。
- 其余的服务提供了支持数据摄取和管理(摄入API)以及唯一实体跟踪和ID生成(UUID API)的应用程序特定功能。
HRA cloud infrastructure
HRA云基础设施
Para_01
- HRA应用程序,包括HRA门户、HRA知识图谱、EUI和RUI都部署在网页上并通过AWS或GitHub页面托管。
- 对于需要服务器端逻辑的应用程序,Docker容器会被创建、测试并通过GitHub Actions自动构建,通过持续集成/持续部署发布到Amazon Elastic Container Registry,然后通过AWS AppRunner或Amazon Elastic Container Service进行部署。
- 对于主要作为静态文件提供的应用程序,它们会通过GitHub Actions自动进行测试和构建,并被复制到Amazon S3用于服务提供,或者推送到分支以供GitHub页面部署。
- 除了GitHub页面外,静态和服务器驱动的应用程序都有Amazon CloudFront作为前端,提供一个支持服务网络的服务网格,该网格支持处理网页请求、跟踪使用情况、将请求代理到在AWS AppRunner或Elastic Container Service中运行的服务以及缓存频繁使用的文件和响应。
- 虽然AWS在HRA云基础设施中被广泛使用,但这项技术非常适合适应其他平台。
Reporting summary
报告摘要
Para_01
- 关于研究设计的更多信息,请参阅本文链接的Nature Portfolio报告摘要。
- ,
Data availability
Para_01
- 所有 HuBMAP 数据均可通过 HuBMAP 数据门户在 https://portal.hubmapconsortium.org 获取。
- Azimuth 引用可以在 https://azimuth.hubmapconsortium.org 获取。
- HRA 数据和代码可在 HRA 门户 (https://humanatlas.io) 获取。
- HuBMAP 和 HRA 的主要和次要数据存储库列在补充表 2 中,HRA 的代码存储库在补充表 3 中。
Code availability
Para_01
- 代码可在三个不同的 GitHub 组织中获取:(1)https://github.com/hubmapconsortium 是 HuBMAP 组织,HRA 由此开始;(2)https://github.com/cns-iu 是印第安纳大学网络科学中心拥有的组织,初始实验性 HRA 代码从这里开始;(3)https://github.com/x-atlas-consortia 最近创建用于托管跨联盟代码,包括 hra-kg、hra-pop、hra-apps 和 hra-api。支持信息位于 https://cns-iu.github.io/hra-construction-usage-supporting-information。
- Supporting information is at https://cns-iu.github.io/hra-construction-usage-supporting-information。
- Basic Information
- Abstract
- Main
- Results
- Flexible hybrid cloud infrastructure for HRA and HuBMAP
- Atlas construction and publication
- Data types and status
- Data enrichment
- Data publication
- Usage and extension of ontologies
- Unified processing workflows
- User interfaces
- Documentation and instructional material
- Previews of Atlas usage
- Perivascular immune cells in lung
- Hierarchical cell type populations within FTUs
- Usage statistics
- Discussion
- Methods
- Expert-generated data
- ASCT+B tables
- 2D and 3D reference objects
- Segmentation masks
- OMAPs and AVRs
- Cell annotation references
- Experimental data
- Tissue collection and RUI registration
- sc/snRNA-seq transcriptomic data annotation
- Cell and FTU segmentation for spatial data
- Cell type annotation for spatial proteomic data
- Spatial alignment for 2D multi-omics data
- Spatial data 3D reconstruction
- Atlas-enriched data
- Mesh-level collision detection
- HRAlit
- HRApop
- VCCF distances and Vitessce visualizations in 2D
- Hierarchical community analysis of cell types
- Atlas validation
- Flexible hybrid cloud microservices architecture
- Hybrid cloud
- Microservices architecture
- HRA cloud infrastructure
- Reporting summary
- Expert-generated data
- Data availability
- Code availability

腾讯云开发者

扫码关注腾讯云开发者
领取腾讯云代金券
Copyright © 2013 - 2025 Tencent Cloud. All Rights Reserved. 腾讯云 版权所有
深圳市腾讯计算机系统有限公司 ICP备案/许可证号:粤B2-20090059 深公网安备号 44030502008569
腾讯云计算(北京)有限责任公司 京ICP证150476号 | 京ICP备11018762号 | 京公网安备号11010802020287
Copyright © 2013 - 2025 Tencent Cloud.
All Rights Reserved. 腾讯云 版权所有