跟着NC杂志文献学习处理scATAC-seq和bulk ATAC-seq数据
跟着NC杂志文献学习处理scATAC-seq和bulk ATAC-seq数据
生信技能树
发布于 2025-11-20 11:34:22
发布于 2025-11-20 11:34:22
前面我们把 ATAC-seq 和 scATAC-seq 数据分析的标准流程都学完了。现在来看一下实际的数据处理,今天的文献来自2021年4月15号发表在 Nature Communications 杂志上,标题为《Single cell regulatory landscape of the mouse kidney highlights cellular differentiation programs and disease targets》。
这个文献同时测了bulkATAC-seq 和单细胞ATAC-seq,可以同时学习这两个内容以及进行技术之间的比较。 我们技能树最新一期生信入门就在11月3号开课,还没上车的可以看一看瞄一瞄,详细介绍页面可以点击:生信入门&数据挖掘线上直播课11月班,完全适合0基础小白。
文献主要内容
本研究通过单细胞分辨率描绘了发育期和成年小鼠肾脏的开放染色质与基因表达图谱。我们发现基因表达高度依赖于远端调控元件(增强子),揭示了肾脏细胞中关键细胞类型特异性转录因子及主要基因调控网络。
- 「Foxl1」:足细胞定向分化发生较早且与持续的Foxl1表达相关;
- 「Hnf4a」:肾小管细胞则遵循更复杂的分化路径——Hnf4a与近端小管命运相关,
- 「Tfap2b」:Tfap2b与远端小管命运相关。
文章技术路线如下:
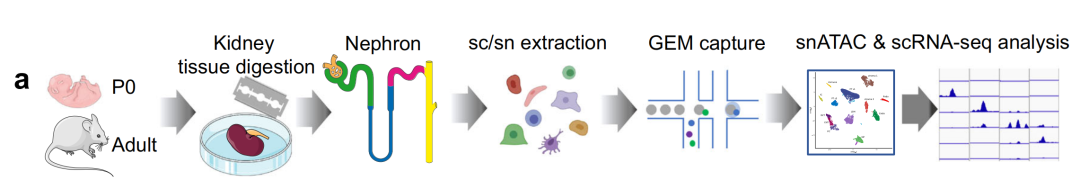
小鼠肾脏的单细胞可及性染色质图谱
样本情况」:对出生第0天(P0)、3周龄(P21)和8周龄(P56)的小鼠肾脏进行了单核ATAC测序(图1a)。小鼠出生时仍存在肾单位祖细胞,且肾单位形成过程持续至约第21天=。同时,在匹配的发育阶段进行了bulk(全肾脏)ATAC-seq分析。
scATAC-seq聚类结果」:基于开放染色质信息进行了细胞类型注释,共28,316个细胞进行分析(图1b)。采用 SnapATAC 方法对细胞进行聚类分析,获得13个细胞簇。
注释」:根据标记基因的可及性特征,成功鉴定了代表以下细胞类型的聚类(图1d):
- 肾单位祖细胞 nephron progenitors:Uncx和Cited1、
- 内皮细胞 endothelial cells:Egfl7
- 足细胞 podocytes:Nphs1和Nphs2
- 近端小管第一段和第三段细胞 proximal tubule segment 1 and segment 3 cells:Akr1c21、Slc34a1和Slc5a2、Kap
- 髓袢 loop of Henle:Slc12a1和Umod
- 远曲小管 distal convoluted tubule:Slc12a3和Pvalb
- 连接小管 connecting tubule:Trpv5
- 集合管主细胞 collecting duct principal cells:Aqp2和Fxyd4
- 集合管闰细胞 collecting duct intercalated cells:Atp6v1g3和Atp6v0d2
- 基质细胞 stromal:Col3a121
- 免疫细胞 immune cells:C1qb
识别细胞类型特异性的开放染色质区域
采用 MACS2 对每种细胞类型分别进行peaks 识别,通过合并这些峰区域构建了完整的开放染色质图谱。
下面其他图:
图e」:snATAC-seq数据与 bulk ATAC-seq数据峰值检测结果对比。双数据集共同识别的峰值显示为蓝色,数据集特异性峰值显示为灰色。
图f」 :展示scRNA-seq数据中细胞类型特异性基因表达的小提琴图。
图g」:显示P0数据中snATAC-seq基因活性评分与基因表达值皮尔逊相关系数的热图。每行代表scRNA-seq数据中的细胞类型,每列代表snATAC-seq数据中的细胞类型。

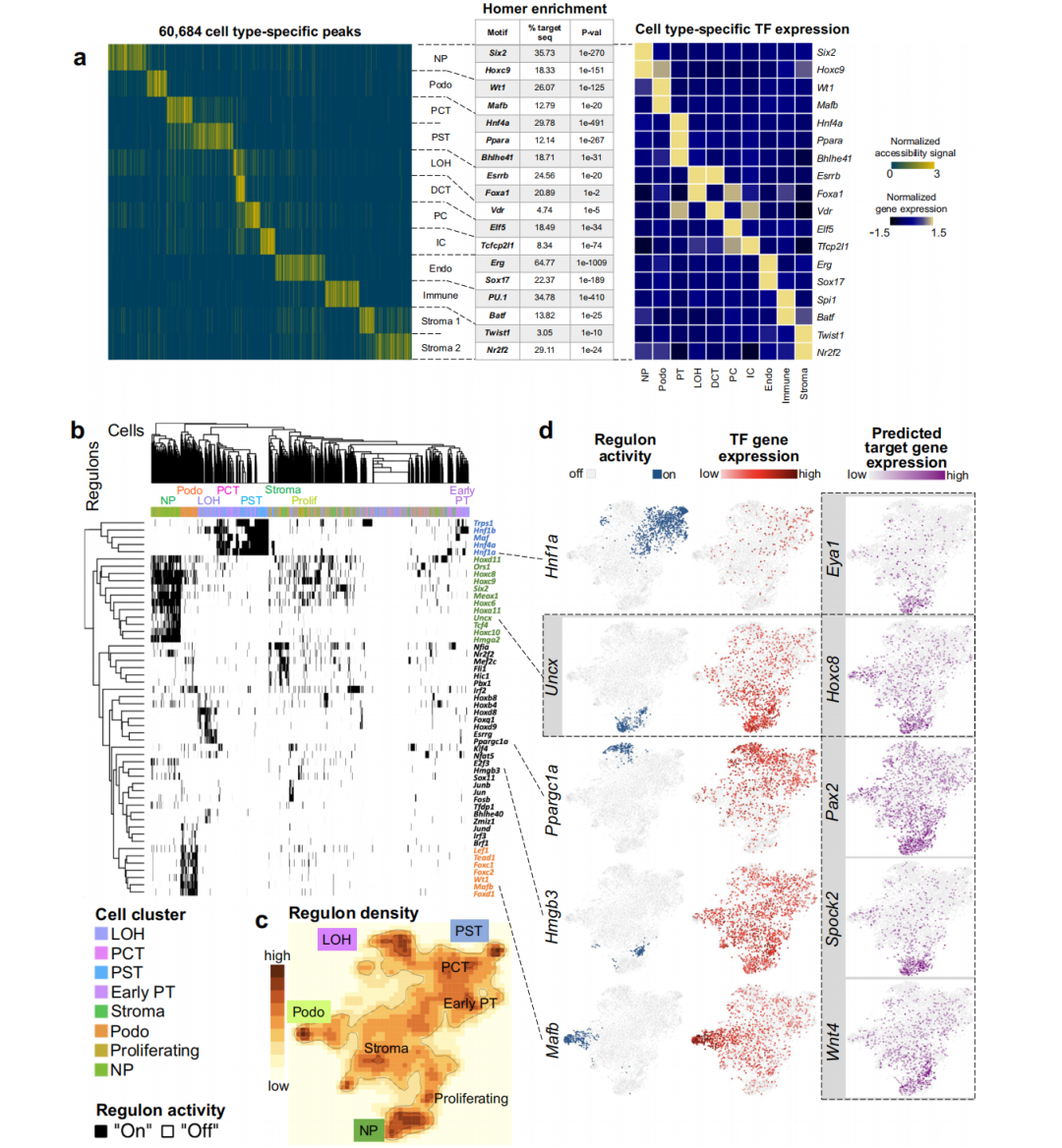
细胞类型特异性调控景观
在13种细胞类型中鉴定出60,684个差异可及性开放染色质峰(DAPs)(图2a),这些峰大部分呈现单一细胞类型高特异性:
图a」 左图:展示所有细胞类型特异性差异可及性峰(DAPs)的热图(黄色代表开放染色质,蓝色代表关闭染色质)(峰值位点信息见补充数据5)。中图:基于Homer软件进行的细胞类型特异性 motif 富集分析示例(完整结果见补充数据6)。右图:与各细胞类型 motif 富集结果对应的转录因子表达z值热图。
图b:」通过SCENIC算法推断的细胞类型特异性调控子热图。调控子活性被二值化为"开启"(黑色)或"关闭"(白色)。
图c」:通过SCENIC算法推断的调控子密度tSNE分布图,用以表征调控子状态的稳定性。
图d」:代表性调控子的tSNE可视化:近端小管(Hnf1a)、肾单位祖细胞(Uncx)、髓袢(Ppargc1a)、增殖细胞(Hmgb3)和足细胞(Mafb)的调控子活性(蓝色开启,灰色关闭)及转录因子基因表达(红色渐变)。Uncx调控子的靶基因表达示例如下(Eye1、Hoxc8、Pax2、Spock2和Wnt4,紫色渐变)。
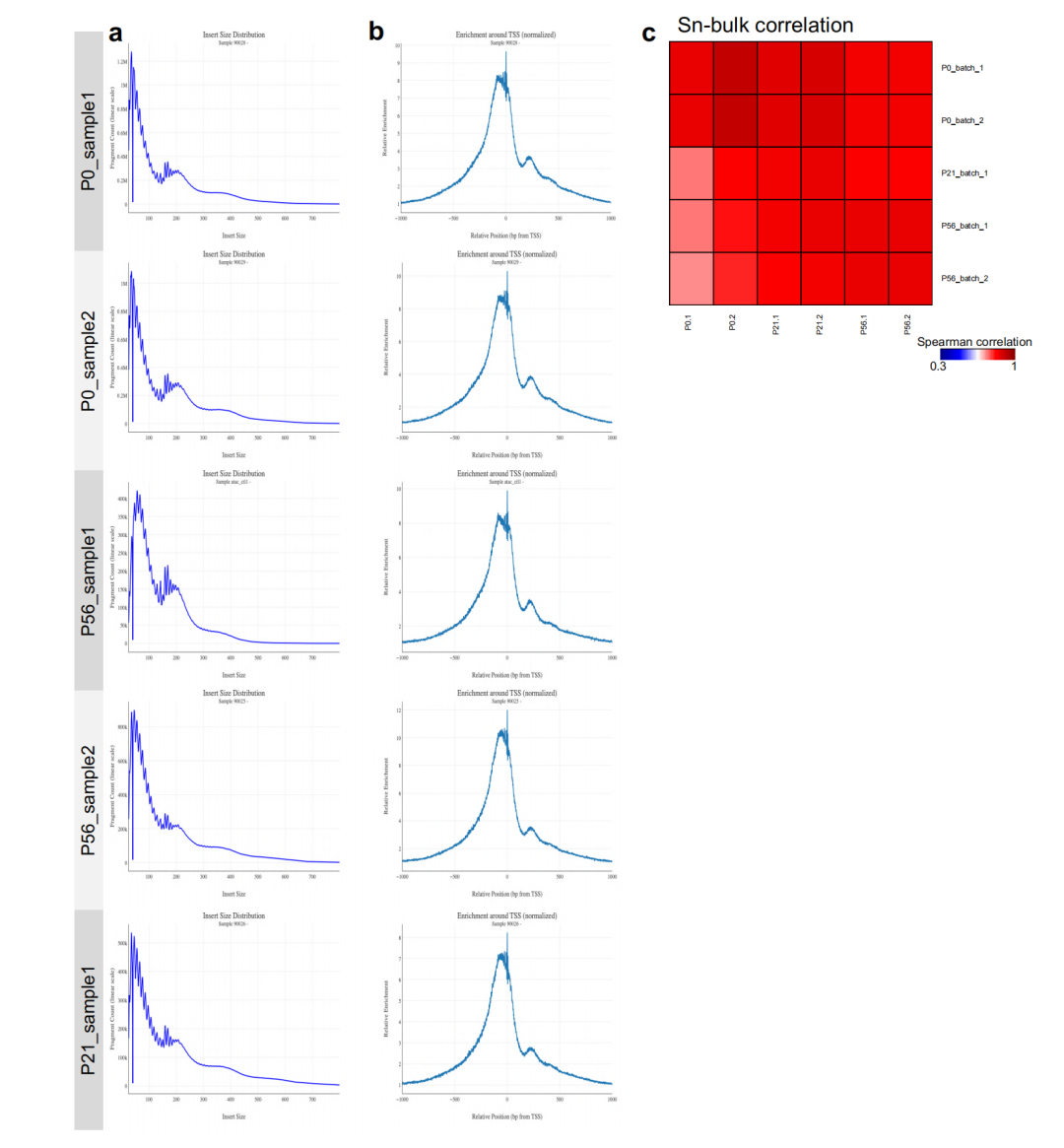
文献中ATAC-seq的质控图
所有样本合并后的snATAC-seq数据集显示出预期的插入片段周期性(「附图1a」),且在转录起始位点(TSS)呈现显著信号富集(「附图1b」),表明数据质量优良。snATAC-seq数据与批量ATAC数据高度一致(相同时期样本间的斯皮尔曼相关系数>0.84,「附图1c」)
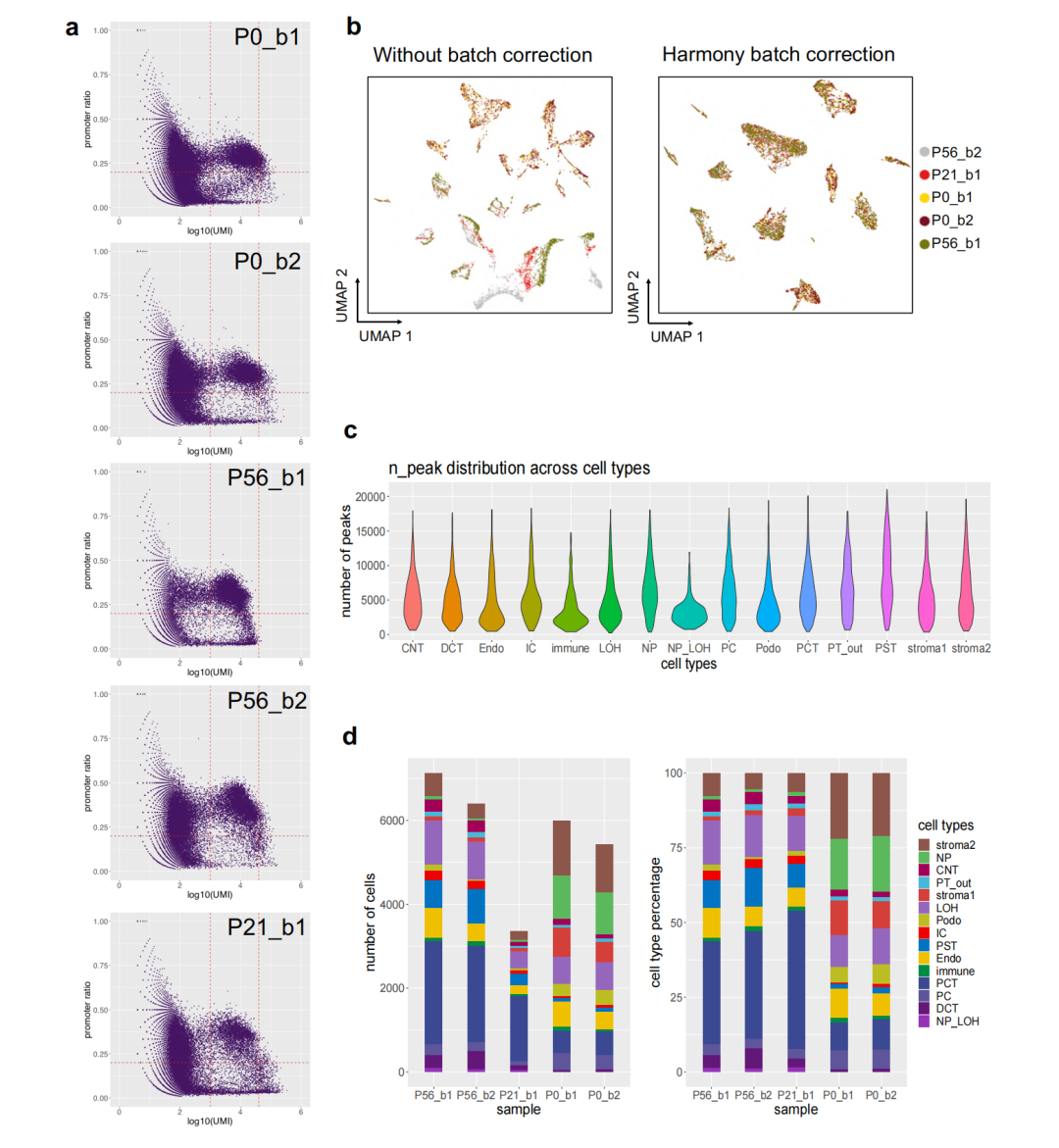
snATAC-seq数据的质量控制与数据处理:
(a) 点图展示5个样本中 UMI(x轴)与启动子比例(y轴)的分布情况
(b) 批次校正前后 snATAC-seq 数据集的 UMAP 可视化
(c) 小提琴图显示 snATAC-seq 数据集中各聚类簇的可及峰数量分布,表明其具有相似分布特征
(d) 堆叠条形图展示不同 snATAC-seq 批次中已识别细胞类型的绝对数量与百分比分布
数据情况
作者不仅公开了原始数据,还通过可检索网站提供了经处理的数据集:
snATAC-seq数据见:https://susztaklab.com/developing_adult_kidney/snATAC/
scRNA-seq数据见:https://susztaklab.com/developing_adult_kidney/scRNA/
峰值轨迹IGV视图见:https://susztaklab.com/developing_adult_kidney/igv/
原始数据和bigwig文件在GEO中:https://www.ncbi.nlm.nih.gov/geo/query/acc.cgi?acc=GSE157079
# snATAC-seq
GSM4752160 P0 mouse kidney snATAC-seq sample 1
GSM4752161 P0 mouse kidney snATAC-seq sample 2
GSM4752162 3-week mouse kidney snATAC-seq sample 1
GSM4752163 8-week mouse kidney snATAC-seq sample 1
GSM4752164 8-week mouse kidney snATAC-seq sample 2
# scRNA-seq
GSM4752165 P0 mouse kidney scRNA-seq sample 1
GSM4752166 adult mouse kidney scRNA-seq sample 1
# bulk ATAC-seq
GSM4752167 P0 mouse kidney bulk ATAC-seq sample 1
GSM4752168 P0 mouse kidney bulk ATAC-seq sample 2
GSM4752169 3-week mouse kidney bulk ATAC-seq sample 1
GSM4752170 3-week mouse kidney bulk ATAC-seq sample 2
GSM4752171 8-week mouse kidney bulk ATAC-seq sample 1
GSM4752172 8-week mouse kidney bulk ATAC-seq sample 2
其实作者还放了代码:https://github.com/Zhen-Miao/dev-kidney-snATAC
是不是很棒,下载下来做做看~
如果上面的内容对你有用,欢迎一键三连!
本文参与 腾讯云自媒体同步曝光计划,分享自微信公众号。
原始发表:2025-10-29,如有侵权请联系 cloudcommunity@tencent.com 删除
评论
登录后参与评论
推荐阅读
目录

