综述《适应性免疫受体库分析》
综述《适应性免疫受体库分析》
三兔测序学社
发布于 2025-11-20 11:37:25
发布于 2025-11-20 11:37:25
摘要:
B细胞和T细胞受体库组成了个体的适应性免疫受体库(AIRR)。AIRR是抗原特异性受体的集合,驱动适应性免疫反应,而适应性免疫反应也刻印在个体的免疫组库中。AIRR可以反映在如自身免疫、感染性疾病和癌症等疾病的发生发展。因此,AIRR分析可以帮助人类疾病的诊断、预后和治疗,实现个性化治疗。目前,高通量测序、高维统计分析、计算结构生物学和机器学习被用于研究AIRR随时间变化以及抗原刺激而发生的重塑与动态变化。本综述概述了AIRR实验和计算分析背后的概念和最先进的方法,并介绍了相关应用的多样性,同时还讨论了AIRR分析中一些突出的问题,如采样、测序深度、实验变异和计算偏差,同时展望了未来 AIRR 分析应用以了解和预测适应性免疫反应的前景。
1.前言
适应性免疫应答是由一系列适应性免疫受体(AIRs)识别抗原后引发的,这些受体包括B细胞受体(BCRs)和T细胞受体(TCRs),分别由B淋巴细胞和T淋巴细胞表面表达。BCRs和TCRs都是由免疫球蛋白超家族(IgSF)的两条链组成的。所有的AIR链都有两个不同的区域:在蛋白的5'端含有抗原结合基团的可变(V)区,以及在蛋白质的3'端的恒定(C)区。每个BCR由两个相同的免疫球蛋白重链(IgHs)组成,它们通过恒定IgH区(IgHC)锚定在细胞表面,同时还有两个相同的免疫球蛋白轻链(IgLs),它们通过二硫键与重链结合(图1)。TCR由两条不同的链组成,它们都通过各自的C区锚定在细胞表面(图1)。有两种不同的TCR异二聚体:大多数T细胞表面表达α/β链TCR(TCRαβ),而γδT细胞则表达γ/δ链TCR(TCRγδ)。每个BCR和TCR的抗原结合位点都位于由其链对的V区形成的三维结构中。BCR可以识别蛋白质、核酸和脂质等天然抗原(图1),而TCRαβ只能与由抗原提呈细胞表面表达的主要组织相容性(MHC)分子提呈的抗原肽结合(图1)。TCRγδ可以识别由各种非经典MHC分子提呈的肽或脂质。个体能够产生多样的B细胞受体(BCR)和T细胞受体(TCR),归功于仅存在于有颌脊椎动物体细胞中复杂重组机制所产生的。适应性免疫受体库多样性使免疫系统具有辨识大量抗原的能力,识别病原性和共生微生物、宿主来源分子以及过敏原等抗原物质。
1.1AIRR的产生
自从发现B细胞和T细胞的起源以来,AIRR已经被广泛研究。在哺乳动物中,BCR和TCR组库主要在初级淋巴器官(PLOs)中产生,分别是产生B细胞的骨髓与产生T细胞胸腺。体细胞重组机制从更大的基因片段库中组装出一组功能性免疫球蛋白(IG)或TCR(TR)基因——可变(V)、多样性(D)和连接(J)片段——所有这些片段组合在一起形成BCR或TCR的V区。IGL、TRA或TRG基因的V和J基因片段以及IGH、TRB和TRD基因的V、D和J基因片段都编码在不同的基因座和染色体上。通过体细胞重组产生的BCR和TCR的V区,可以进一步分为四个高度保守的框架区(FRs)和三个互补决定区(CDRs),这些CDR区域展现出更多的序列多样性。CDR1和CDR2是由V基因片段种系编码的,而CDR3是由V、(D)和J基因之间核苷酸的随机插入和删除产生的。在重组过程中,CDR3在长度和序列上是高度可变的,这极大地促进了AIRR的高度多样性以及对抗原的识别。在这个VDJ重排过程结束时,每个B淋巴细胞和T淋巴细胞在其表面分别表达多个拷贝的独特BCR或TCR,其特征是一组特定的V、D和J等位基因组合以及独特的CDR3。这些淋巴细胞在遇到次级淋巴器官中的特异性抗原后会迅速进行克隆扩增。
在B细胞分化晚期,体细胞突变可能发生在BCR重排后V区任何位置,即体细胞超突变(somatic hypermutation,SHM)。SHM导致克隆谱系的形成(不依赖于VDJ重排,而是相同VDJ组合下序列突变所产生谱系),该谱系表达经过DNA序列突变,与亲本BCR序列有所差别的BCR,通常对特定抗原的亲和力有所增强,因此在氨基酸水平上也发生了变化(图1)。此外,B细胞在遇到抗原刺激时,可以通过在重链恒定区(CH)内发生染色体内的缺失重组进行免疫球蛋白类别转换。这意味着来自同一亲本克隆的后代细胞可以产生不同类型的抗体,即具有不同C区和不同效应功能的抗体,而不改变其抗原特异性。
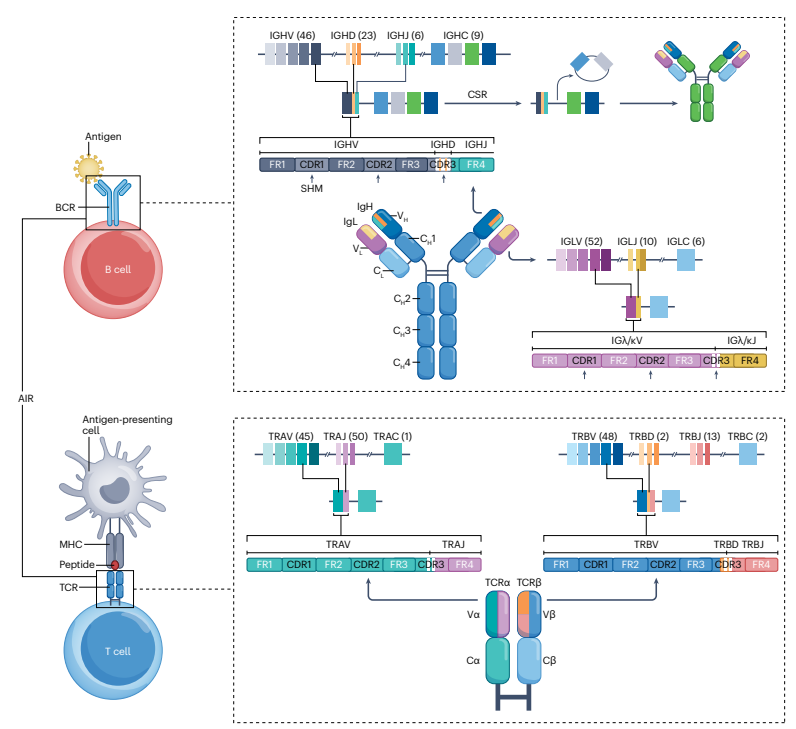
图1|适应性免疫受体生成和结构。
1.2 适应性免疫受体库的多样性与特异性
在初级淋巴器官中生成的潜在独特AIR的数量估计约为10^19个TCRs和10^13个BCRs,在人类中,这种多样性可以通过体细胞高频突变(SHM)进一步增加BCRs的多样性。最近,通过统计建模预测的AIRR多样性为10^61。然而,由于宿主所能容纳的淋巴细胞数量有限(人类约为8×10^11个,小鼠约为10^8–10^9个),在个体中仅存在潜在的BCR和TCR库的一小部分。此外,在初级淋巴器官和次级淋巴器官中,AIRR都受到各种选择事件的影响,这些事件发生在TCR和BCR与自身抗原或非自身抗原相互作用过程中(图2)。在初级淋巴器官中,这样的选择机制是通过筛选能够识别非自身抗原的T/B细胞,清除具有高亲和力自身反应性T/B细胞,构建了既具有免疫多样性又具有免疫平衡的适应性免疫库。
AIRR的高度多样性对于形成对抗病原体的免疫力以及维持宿主稳态至关重要。个体的AIRR揭示了正在进行的免疫反应的信息,同时也反映了先前的抗原接触,因为这些信息在适应性免疫记忆细胞TCR或BCR库中有所体现。此外,对免疫细胞受体特异性的研究导致开发了具有价值的实验研究工具(例如基于抗体的检测方法),诊断工具(例如基于血清的诊断方法),疾病预防(疫苗设计)和治疗(例如基于TCR、嵌合抗原受体T细胞(CAR-T细胞)和抗体的疗法。除了人类和小鼠外,AIR基因位点与重组机制也在有袋动物、鸟类和鲨鱼等其他脊椎动物中进行了研究,这些研究揭示了物种间的差异,促进了多个物种的AIR基因的标准描述和注释成为可能。此外,AIRR分析已被应用于探索病毒感染或在进行初次免疫加强接种后B细胞和T细胞免疫反应和记忆形成的动力学研究。因此,研究不同物种的AIRR可能会开辟如如进化和比较免疫学等新的研究途径。
然而,自克隆选择理论提出以来,关于AIRR的多样性、特异性和功能的这些基本问题在半个多世纪里一直没有得到完全解答。个体在特定时间点内AIRR多样性及其随时间的波动以及在特定AIRR内不同淋巴细胞克隆的数量,每个克隆的克隆大小,对特定抗原特异性的克隆的数量以及交叉反应的程度都是急需解决的问题。此外,在淋巴细胞个体发育、选择事件、外部和内部扰动中,它们的适应性免疫组库是如何形成的仍不清楚。最后,关于个体如何对特殊病原抗原建立有效的免疫反应(没有特定的TCR,BCR被激活,没有特定克隆被激活),同时避免过度的组织损伤。另外AIRR在多大程度上推动了病理性自身免疫的发展。回答这些问题需要对AIRR进行深入的定量研究才能知晓。因此,由于技术的进步,现在可以通过bulk和单细胞测序(AIRR-seq)以及空间组织水平来研究AIRR,结合机器学习方法对AIRR-seq数据进行分析,构建数据驱动的预测模型。基于对个体内部和跨个体AIRR多样性以及AIRR克隆结构的了解,这些进展有望帮助回答上述大部分问题。AIRR-seq领域在过去十年中取得了突破进展。在本文我们重点介绍AIRR研究的实验设计和计算分析的关键概念,包括机器学习。针对每个被讨论的概念,我们都提供了应用示例。此外,我们强调可重复性以及实验和计算优化的需求,并强调该领域当前遇到的难题。
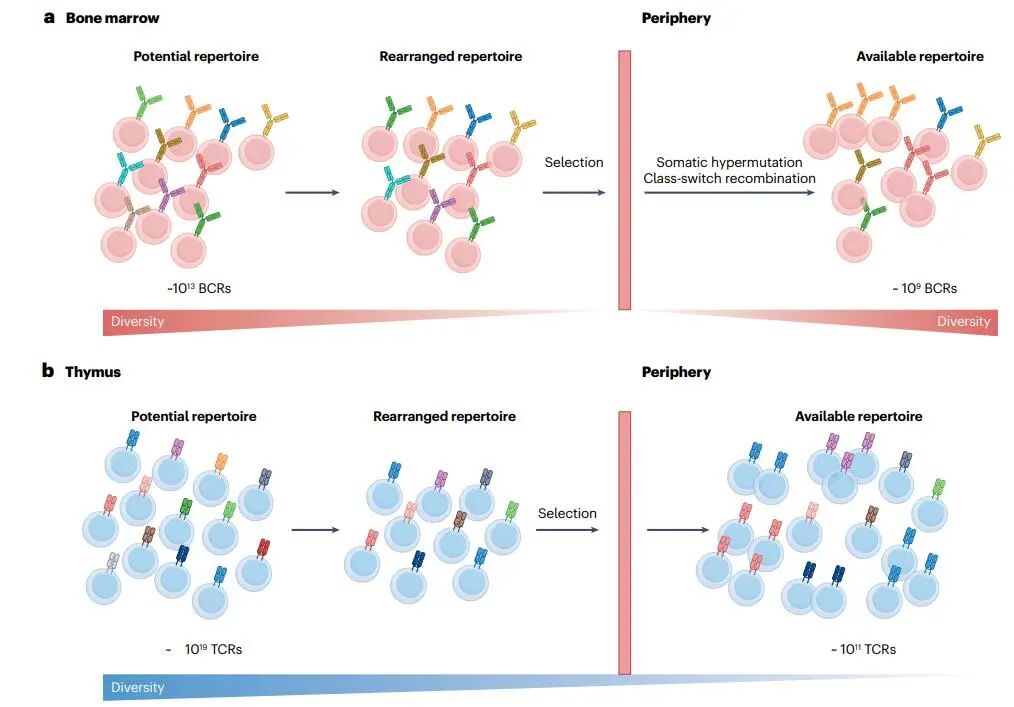
图2:免疫组库多样性的改变
2.实验
最初,基于细胞生物学的方法,尤其是流式细胞术,用来检测那些表达特定V基因片段的T,B细胞的相对丰度。由于VDJ重组机制的复杂性推动了分子生物学方法的发展,如CDR3谱分型或免疫分析,这使得描述性、定性的AIRR分析成为可能。随着高通量测序(HTS)方法的出现,定量AIRR分析在2009年才建立,该方法可同时对多达数百万个DNA和RNA分子进行测序。随后研究者不断努力来改进了实验方法,减少技术偏差并确保可靠的重复性,以及采用新技术如单细胞TCR和BCR测序和空间转录组学。为了确保AIRR实验评估的可靠性与准确性,必须考虑多种因素,包括生物样品的类型、核酸起始模板的选择、文库制备方法和高通量测序标准方案等实验细节。
2.1 生物样本的类型
实验中使用的样本类型既取决于样本的可获得性,也取决于要解决的生物问题。B细胞和T细胞可以从生物体液或新鲜和保存的组织器官中收集。冷冻和福尔马林固定石蜡包埋(FFPE)样品则需要进行下游处理(图3a)。虽然在实验动物模型中,器官和组织可以很容易地收集用于AIRR分析,但在人体中,只能从器官捐赠者获得整个组织或器官,或者是只有在常规临床护理下的活检样本。这使得在自身免疫、炎症或感染性疾病以及液体或实体癌症的背景下研究组织驻留的适应性免疫受体成为可能,在实体肿瘤中可以捕获到驻留有适应性免疫细胞的肿瘤微环境。
血液由于其可及性、非侵入性采集和相对丰度方面具有优势,在大多数转化和临床研究中仍然是首选的样本类型。如果可能的话,强烈建议从样本的采集可以从外周血单个核细胞(PBMC)开始,而不是从全血开始,特别是当使用RNA作为文库制备的起始材料时,因为全血含有约80%的β-珠蛋白RNA,这可能会降低对白细胞转录物的检测。此外,为了保持全血中RNA的质量,冷冻与稳定性是必不可少的,这与细胞分选不兼容。考虑到血浆中游离细胞DNA的浓度增加,它也可以用于癌症研究。或者,淋巴细胞可从其他来源收集,如滑膜和脑脊液或支气管肺泡灌洗液。强烈建议从新鲜样品开始实验以保存原始AIRR成分。只要组织中的细胞保持完整,FFPE或冷冻组织也是可以进行AIRR分析。当组织在石蜡中放置很长一段时间,AIRR质量就会下降,并且冷冻组织在冷冻与解冻循环中容易丢失脆弱的细胞。在处理这些样品时,必须检查核酸的质量和数量以及淋巴细胞浸润情况以确保有可以分析所需的材料。
最后,要考虑研究对象是整体的淋巴细胞还是分选细胞亚群(图3b)。大多数AIRR数据是通过bulk测序技术从数百万个B细胞或T细胞中收集的。虽然研究总细胞可以减少操作时间和实验成本,但功能相关的AIRR修饰和对细胞亚群多样性的见解可能被忽视。例如,浆细胞中的mRNA水平比初始B细胞高10至100倍,因此这可能会对所获得结果的生物学解释产生偏差。所以,在进行细胞分选之前,需要根据其表型和功能特征。在进行细胞分选时,如果需要研究稀有细胞亚群对特定病理的贡献,建立一个高效的分选策略至关重要,该策略应结合速度、效率和细胞纯度。最后,虽然Bulk测序没法研究 AIR 链配对,但单细胞技术可以在不超过 10^4个细胞的样本实现AIR链配对研究,例如小细胞亚群、肿瘤活检细胞或具有特定抗原特异性的分选细胞。
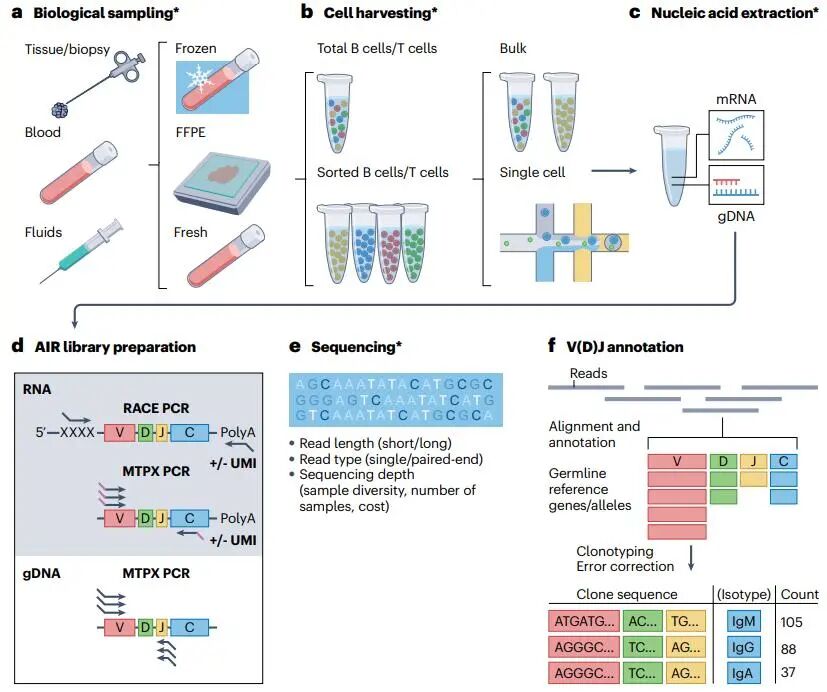
图3 | AIRR样本采集、制备和测序
2.2核酸起始模板
文库制备最合适的起始材料取决于可用于分析的淋巴细胞数量和样本来源。这些材料可以是混合/分选的细胞样本中的基因组DNA(gDNA)或mRNA。一方面,gDNA的数量与细胞数量成正比,因为每个重排在一个细胞中只有一个拷贝,这提供了gDNA分子与细胞计数之间的完美线性关系。尽管gDNA文库能够量化BCR或TCR克隆型,但它们需要更高的起始浓度,当研究的样本尺寸有限时,这将是一个障碍。另一方面,使用mRNA作为起始材料不能量化克隆型的绝对丰度,但与gDNA相比,它提供了10-100倍的敏感性,因为每个转录本在给定细胞中以多个拷贝存在。因此,mRNA提供了更大的可及性,特别是对于核酸数量有限的小样本。当然前提是样本能够被很好的处理,因为RNA对容易降解。此外,起始模板使用更有可能被有效翻译并产生功能性BCR或TCR的mRNA转录本,可以减少AIR-seq数据的背景噪声。而使用gDNA则不确定是否产生功能性VDJ重排,因为也会检测到父母等位基因相反的非功能性的重排。最后,mRNA可以鉴定免疫球蛋白同种型,而gDNA可变区和恒定区被内含子隔开。目前,单细胞AIRR-seq完全是基于RNA的,因此细胞活力对于确保每个单细胞的RNA正确封装至关重要。重要的是,必须检查样本遗传物质的浓度和质量,因为这两个因素可以决定所选择的模板是否可以在后续步骤中使用。
2.3扩增和文库制备
对基于 DNA 和基于 RNA 的扩增方法的基础研究已经评估了它们在bulk测序中的具体优势和劣势,并强调在研究免疫组库时的实验设计选择适当方法的重要性(图 3d)。适合于 gDNA 和 mRNA 模板的多重 (MTPX) PCR, 是使用针对 J 基因或 C 基因的引物以及针对V 基因的多种引物的混合物。该方法的一个缺点是在反应中使用大量引物可能会出现导致竞争性扩增某些基因片段,另外一些基因片段则少有扩展。为解决这一问题,可以使用快速扩增 5' 互补 DNA 末端 (5' RACE)。5' RACE 仅适用于 mRNA 模板。该方法依赖于逆转录酶活性,在 cDNA 的 5' 端加入GGGG接头序列,然后使用该接头作为后续套嵌 PCR 扩增的靶区,另外一端的引物则靶向 C 区,这种方法是不需要引物靶向转录本 V 基因进行特异性扩增的方案。但是,根据使用接头连接或接头掺入的实验方案中,也可能会出现接头掺入效率低的问题。此外,它更容易出错,因为逆转录酶的出错率比用于 gDNA 基因组 MTPX 方法的 DNA 聚合酶的出错率高得多。尽管如此,5' RACE 策略正变得越来越受欢迎,因为它不需要基因特异性引物,因此可以捕获新的等位基因变体。
现在已经有多种商业化与实验室方法用于 AIRR-seq。基于 MTPX 的试剂盒正变得越来越受到商业供应商的欢迎,其中 iRepertoire和 Adaptive Biotechnologies 率先开拓了这一领域,而 Illumina、Archer、Cellecta 和其他公司也正在这一领域崭露头角。基于 5' RACE 的方案也由若干家公司提供,尽管没有 MTPX 那样多。目前可以从 Takara Bio、NEB 和 BGI 获得 5' RACE 试剂盒。Milaboratories 现在提供针对人类和小鼠样本的 不同的 MTPX 和 5' RACE 产品。目前也有许多基于 RACE或 MTPX的实验方案来用于免疫组库研究。
对单细胞免疫组库测序的方案与针对多细胞测序的方案使用相同的文库制备技术,但其在细胞分离策略上存在差异,这决定了每次运行中的细胞数量。例如,基于平板的方法可将单个细胞分离到 96 孔或 384 孔板中,这可实现对单个细胞进行可靠的定性分析,尽管每次运行的细胞数量有限且成本较高。相比之下,10x Genomics Chromium 和 Illumina 的 液滴技术广受青睐的原因在于它可将 10,000-100,000 个单个细胞分别包裹在单独的液滴中,每个液滴中都含有细胞裂解、逆转录和分子标签所需的试剂。尽管与其它方法相比,基于液滴的单细胞技术成本相对较低,但其灵敏度较低且更有可能产生双细胞液滴。现在,通过新推出的 TCR 组库测序方案,可实现更深层次的通量,该方案可对单次实验中的百万个细胞进行分析。这些技术分别由 Parse Biosciences 和 Omniscope 公司提供,它们通过在每个转录本上添加条形码,从而摆脱了微流体方法的束缚。
尽管近年来PCR试剂和方案有了飞快的改进,允许整体无偏见的扩增,在文库制备过程中仍然会发生错误,包括对特定序列组成有偏向性的扩增;DNA分子的随机扩增;以及产生错误序列的技术伪影,也称为 jackpot 突变。 jackpot 突变可能在 PCR 的早期循环中发生,导致错误序列的指数级扩增。伪影还可能来自 PCR 模板在相似序列之间的切换,产生杂交序列。可以采取实验和计算错误校正策略,单独或联合使用帮助检测和消除这些失真。例如,使用引物对照,即具有已知特性的生物或合成 AIR 序列,如克隆序列和频率,可以帮助检测酶促效率和扩增偏倚性。其他策略包括在基于 RNA 的扩增方法中引入独特的分子标识符 (UMIs)。UMIs 是短序列条形码,在 cDNA 合成过程中可以附着到每个基因模板上,这样每个分子都会附带一个独特的条形码。这使得能够识别最初来自同一模板分子的重复序列,从而可以准确地量化每个克隆型的丰度,并更正扩增错误。
在测序之前,需要对获得的文库进行表征(俗称库检),以确保目的产物成功扩增、纯化和片段大小合适。检查包括文库是否在适当片段长度处有一个清晰的单一峰,而这可以根据扩增方法和所分析的AIR链类型来判断,并使用凝胶系统和专门测序检测设备如例如安捷伦的Bioanalyzer或TapeStation进行检测与分析。
2.4 高通量测序—文库构建与测序
选择合适的高通量测序(HTS)平台,要考虑读取长度(短读与长读)、读取类型(单端读取用于部分扩增子覆盖,或双端读取用于完整扩增子覆盖)以及测序深度(即每个样本应检测到的转录本数量),测序深度也要考虑细胞群体的多样性(见图3e)。最合适的测序平台应确保准确且充分地覆盖所研究的细胞群体,同时有效控制错误率和实验成本。
为了可靠地进行可变区和C区的单倍型分析,现有专用的长读(>1 kb)测序平台。短读测序定义为生成150-300 bp的读取长度,可以覆盖完整的CDR3区域和可变的V(D)J区域。然而,短读平台通常在序列的两端具有较差的碱基调用质量,这可能导致测序错误。通过执行双端测序,可以减少测序错误,因为它可以对重叠区域进行比对,尽管这样可能会导致当其中一个读取质量较差时,克隆型深度下降。当使用2×300 bp的双端短读测序,可以可靠地分配V基因和V等位基因,因为它覆盖了完整的重排区域,通过检测完整的CDR1以及发生SHM的CDR2序列(见图1)。测序引物的位置在很大程度上决定了V基因和C区的覆盖比例(C区的覆盖对于免疫球蛋白亚型的确定是至关重要的)。
UMI(唯一分子标识符)可用于纠正测序错误,这在研究免疫球蛋白的细胞内克隆多样性和抗体形成时尤为重要。然而,每个UMI选择合适读取数阈值是一个关键步骤,因为过于严格的阈值可能导致克隆库覆盖范围急剧减少,并过滤掉潜在的有信息价值的低频读取片段。将UMI与深度测序相结合可以减少这种损失。然而,这种策略在研究数量稀少的细胞群体时依然具有挑战性。
随着文库的数量和浓度增加时测序深度会下降,因为每次测序运行的读取容量有限。此外,过度测序已被证明会改变小样本的克隆分布,并产生噪声。因此,测序深度应根据样本的大小和多样性以及每次测序运行中的样本数进行调整。相比之下,当分析大样本时,增加测序深度更为适宜,而测序重复则有助于更好地覆盖真实的克隆多样性和探索克隆重叠。
MTPX测序平台能够在一次运行中高效地对大量样本进行深度测序,这样可以降低成本,但也增加了跨样本污染的风险。然而,可以通过使用独特的双索引,在文库适配器中引入独特的双索引来解决这个问题。这些索引确保了通过过滤掉因索引跳跃(不同文库间的双索引交换)导致的读取,确保了准确的去重。
在规划AIRR-seq实验时,生物样本类型、起始模板材料、文库制备方法和测序平台的选择都是重要的考虑因素。尽管目前尚未为这些步骤建立金标准,但建议尽可能在同一项目中对样本进行统一处理,以减少实验偏差。首先,建议在同一实验中使用相同类型的样本,因为样本类型会影响收集到的细胞和核酸材料的质量和数量,可能导致处理方式不同的相同样本之间出现多样性差异。其次,遵循单一的测序方案有助于减少由于测序错误和测序深度引起的变异,从而确保AIRR-seq数据的准确比较和解读,因为这些因素通常依赖于平台和技术。因此,实施如混合细胞群体作为并行生物学对照的策略被提议用于检测和纠正AIRR-seq实验中的批次效应。例如,通过使用具有预定义V(D)J重排的淋巴细胞(B细胞或T细胞)混合物,可以在不同的样本批次和测序运行中,将其相对丰度与预定义的丰度进行比较。
3.测序数据分析
AIRR 数据预处理和分析从测序完毕后下机释放的原始测序数据开始,到分析结果进行生物学解释为止,本章节从整体到局部细节开始进行描述免疫组库分析过程,表 1 和 2 总结了可用于 AIRR-seq 数据分析的大量工具。
3.1数据预处理
主要测序平台生成的未处理的AIRR-seq数据通常是FASTA或FASTQ输出文件。AIRR 数据预处理的主要目标是控制测序数据质量、校正 PCR 和测序错误、注释等位基因、根据预定义的序列特征(例如特定的 V(D)J 序列)组装克隆型,并以人类可读的表格格式输出,其中列是序列特征(V、D、J基因类型、FR区和CDR序列),行通常是唯一的克隆型(图 3f)。我们还提供了补充表 1,列出了每一步可用的工具以及这些工具是否支持群体和单细胞AIRR数据。
首先,纠错步骤包括根据 Phred 分数(衡量碱基质量的一种指标)过滤低质量的读取片段、根据测序相似性对Reads进行聚类或根据 UMI 对读取进行分组。基于 UMI 的分组可以由多种处理软件实现(补充表 1),并且可以在BCR 数据中用于将SHM产生的真实突变与 PCR 和测序错误区分开来。Gupta 等人提供了使用 pRESTO 对 BCR 测序数据进行详细 UMI 基础校正的工作流程。第二,在纠错过程中或过后,进行 germline 基因或 germline 等位基因注释。在这里,经过校正的Reads被比对到到一个物种特异性 germline 数据库,以确定每个Reads的 germline 基因信息、框架外区域 (FR) 和可变区 (CDR) 以及 BCR 数据中的体细胞突变 (SHM)(SHM 计数和 SHM 类型)。虽然通常使用单个 germline 基因参考数据库对所有供体的 AIRR 数据进行注释,但现在越来越多的做法是为每个供体构建参考数据库,以更准确地展示 BCR 和 TCR germline 等位基因。这种 germline 等位基因特异性注释对于下游比较以及对 BCR 数据中 SHM 的准确展示将非常重要,因为个体特异性多态性容易被错误地识别为 是SHM。尽管对 BCR 基因进行了大量研究以确定 germline 多态性,但对 TCR 的等位基因分析才刚刚开始。第三,具有预定义组装特征的测序reads被聚集到一个克隆型中,并提取其丰度。第四,经过校正和标准化的数据输出供下游分析。标准输出格式是 AIRR 社区 (AIRR-C) 成员开发的 MiAIRR 格式。值得注意的是,AIRR 数据也可以从bulk和scRNA-seq数据中重建,不过有效信息较少,此类工作流程不在本教程的讨论范围内。
3.2 AIRR数据探索与分析
AIRR数据的分析可以分为不同层次的分析,从描述性分析到预测建模再到推断AIR的特异性。AIRR数据分析的第一步是计算AIRR总结性统计信息,这些信息主要描述种系和克隆计数信息。随后,进行更详细的分析关注AIRR多样性、AIRR组成相似性、克隆结构和机器学习辅助的AIRR推断或预测。图4a提供了AIRR数据一般分析思路,补充表2中所有工具(除单细胞分析类别外)均可应用于bulk免疫组库测序序列数据。一些工具可用于单细胞,尽管尚不清楚如何处理配对链数据多样性、系统发生学、聚类和机器学习方法分析。
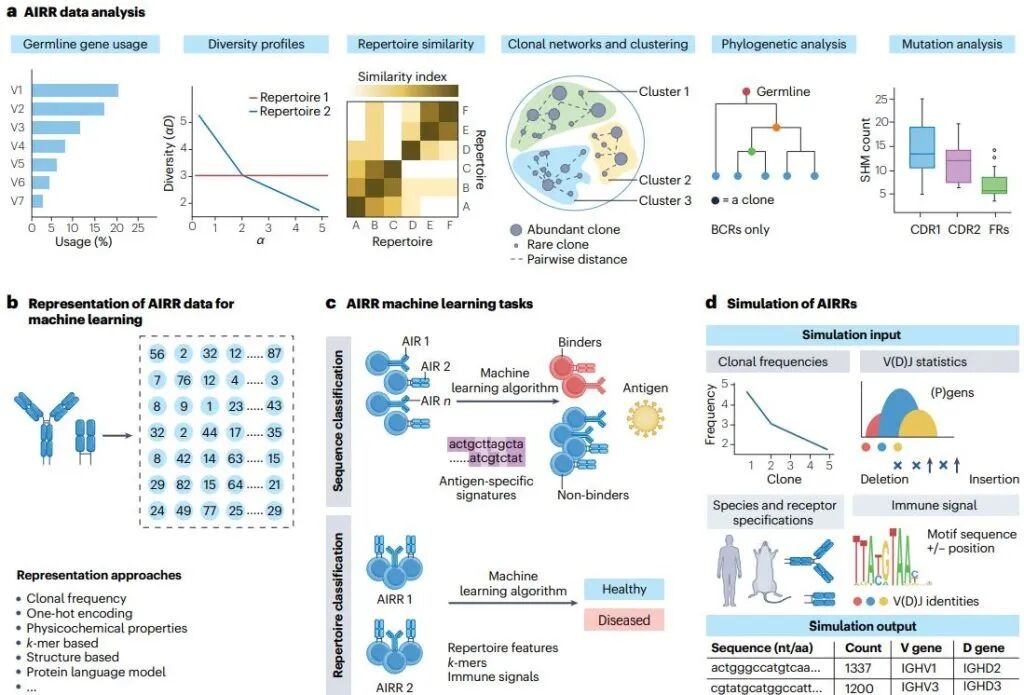
图4 | AIRR-seq数据的生物信息学下游分析
AIRR概要统计。每个AIRR内的恒定区V、D和J基因使用频率(某个恒定区基因在某个AIRR内被使用的频率)以及CDR3计数信息是AIRR的基本描述特征。除了微小的变异外, 胚系基因在个体间的使用通常是稳定的。在不同免疫状态下,外周血单核细胞水平的基因库使用也相似。但是,在一些 B 细胞和 T 细胞亚群以及细胞发育阶段中观察到了免疫组库的差异。在个人之间基因库使用有重大差异通常意味着文库制备过程中存在技术问题。尽管在可比状态下个人之间基因库使用通常相似,但 比较不同大小的细胞群体(如初免细胞与抗原接触过的细胞)中,CDR3 数量在样本之间可能由于技术偏差性会产生很大差异,。在预期相似测序量的样本之间,如果观察到 CDR3数量差异性大,那应该在进行下游数据分析之前进行研究并进行均一化校正,避免队后续研究结果造成影响。
AIRR多样性。AIRR多样性通常使用首先在生态学中开发的多样性测量方法(用于动植物种群丰度计数和比较)来计算。这些多样性测量方法同时考虑物种(例如克隆型)丰富度(不同物种的唯一数量)和物种丰度分布。简而言之,一个有n个克隆型的AIRR的多样性使用Hill多样性公式(Rényi熵的指数)计算,该公式包括了常用多样性测量方法中的许多特殊情况。Hill函数的一些特殊情况与AIRR领域中的多样性指数有关:物种丰富度指数、指数型香农-威纳指数、辛普森指数的倒数、基尼指数、Pielou指数和伯格-帕克指数。两个AIRR可能因为使用的多样性指数不同而产生截然不同的α多样性值,多个多样性指数比单个多样性指数更准确。给定实验样本估计AIRR的总多样性仍然是一个待解决的挑战,目前还没有提出令人满意的解决方案。此外使用多样性指数来衡量AIRR克隆扩增的状态。为此,将Hill多样性值除以样本的物种丰富度,得到一个称为“均匀性”的度量。均匀性在接近零和一之间取值,衡量克隆型频率分布(一个AIRR的克隆型频率向量)与均匀分布的偏离程度。可以通过使用基于熵的多样性指数来估计跨样本多样性。原始细胞群体具有很高的均匀性,而经历过抗原的AIRRs则具有相对较低的均匀性。
AIRR克隆进化树分析:AIRR网络结构结构定义了AIRR内所有AIR(抗原受体)之间的多对多序列相似性景观。鉴于AIRR序列的高度多样性,AIRR网络结构结构分析能够识别AIRR中高/低序列相似性区域,并将其与抗原结合等生物学功能关联。该网络结构结构的数学基础是所有克隆型之间的全对全距离矩阵,该矩阵可转化为网络图(节点代表克隆型,边代表预设的序列相似性阈值)。相似性阈值通常采用全局序列比对方法计算,包括Levenshtein距离和Hamming距离,默认阈值为1个核苷酸或氨基酸差异,但更大差异范围也有研究。当克隆型数量超过10^5时(现代AIRR实验数据集的典型规模),距离矩阵的计算将面临显著挑战,为此开发了专门的大规模克隆网络计算流程imNet。对于包含数千节点的网络,可使用igraph、NetworkX、Geph或Cytoscape等软件进行可视化。通过图属性和网络分析可量化AIRR网络结构结构特征,这些特征既可在AIRR整体水平(每个网络一个系数)分析,也可在抗原受体(克隆型)层面(每个克隆型一个系数)描述。AIRR整体水平分析系数包括度分布、聚类系数、网络直径和同配性—节点的度表示其连接边数(即相似克隆型数量),而度分布则统计整个库中不同连接频率的克隆型占比。例如,幂律分布网络呈现少数高度连接克隆型和多数低连接克隆型,这种网络结构结构可能与抗原驱动的克隆扩增相关;而指数分布网络则反映初始库中克隆型连接度的均匀分布。在克隆型层面,PageRank等参数可量化两个CDR3克隆型在网络中的相似性重要性。值得注意的是,克隆型层面的相似性分析可扩展至短链氨基酸基序(k-mers)的识别,因为短肽段(如k-mers)能影响表位结合亲和力。通过共享k-mers对相似AIR进行聚类已成为预测抗原/表位特异性受体的常用,近期研究还整合了转录组信息以增强预测能力。
B细胞谱系发育学。当接触抗原时,B细胞会通过BCR(B细胞受体)可变区的扩增和突变产生分化,形成从初始未突变B细胞到记忆B细胞和经历体细胞高频突变(SHM)的浆细胞的发育谱系。研究抗体库的进化过程,能揭示疫苗和病原体如何影响机体的体液免疫反应。要推断单个B细胞间的祖先进化关系,通常通过克隆谱系(clonal lineage)的序列构建谱系树。克隆谱系指源自相同重组事件的受体序列集合,具有共同祖先。构建谱系树时,常规预处理步骤是按V/J基因和CDR3长度对序列分组,但具体流程可能因谱系定义而异。谱系树也可通过数据驱动方法识别。B细胞谱系分析常用最大简约法和最大似然法等标准算法构建系统发生树,但难以确保所得生物树完全准确。为适应B细胞特性,开发了IgPhyML等上下文感知算法(如优先热点突变,抑制冷点突变)。此外,BCR库常含数百个独立克隆,传统模型需单独处理各克隆谱系,影响效率。采用库级模型(共享部分参数)可提升精度。近期开发的Dowser R包[223]实现了从轻重链BCR序列推断B细胞谱系,并支持迁移、分化和同型转换等过程的统计建模。第三,B细胞群体数据中克隆丰度差异显著,纳入丰度信息可提高树推断准确性。最后,免疫球蛋白树可视化可通过多种工具实现。
AIRR组成的相似性:对AIRR组成的比较——无论是胚系基因层面还是CDR3序列层面——都是鉴定共享克隆型的关键,这些克隆型可在同一受试者的不同细胞群体或组织中出现,也可在不同个体间出现。这类克隆型通常称为公共克隆型;在初始库中,它们的存在可部分归因于V(D)J重组统计学或趋同重组。例如,较短的CDR3具有更高的产生概率,因此更可能被生成并观察到。公共克隆型也可能反映中枢选择的偏好,或抗原驱动的选择,因此在相同免疫遭遇或疾病背景下,可在不同个体间观察到共享克隆型。用于库比较的方法包括:仅用存在/缺失计算克隆重叠的指标(如Jaccard指数);同时考虑频率信息的指标(如Morisita–Horn指数或Jensen–Shannon散度指数)。借助CompAIRR工具,可快速鉴定公共克隆型,尤其适用于跨大规模数据集;该工具还能快速发现相似序列,即样本间仅差几个氨基酸的克隆型。值得注意的结构分析表明,AIRR间的结构相似性可能高于序列相似性。
近期,基础库统计与多样性度量已整合基于序列的相似信息,以在计算库间与库内相似性时纳入高度相似的序列。更通用的ImmuneREF工具通过整合多项AIRR及序列与频率特征(包括基因使用、克隆扩增与克隆重叠),量化库间相似性,使研究者能利用计算与实验免疫学可解释的金标准解读库间差异。然而,由于序列相似性的微小差异可能导致抗原结合能力不同,若从抗原结合视角出发,这些度量可能无法准确代表AIRR多样性。
随着深度学习方法的兴起,仅基于序列的抗体结构预测已愈发常见。现已出现专门针对抗体(较少针对TCR)的结构预测工具,可大规模预测数十万乃至数百万抗体结构,实现库级别的结构比较。结构比较备受关注,因为AIR的三维结构决定其与抗原的相互作用及结合特性。序列相似的AIR可能采取不同构象,反之亦然。需注意,尽管AIR结构预测性能持续提升,但随CDR3长度增加,预测准确度会下降,或出现顺式酰胺键错误、立体化学错误、原子冲突等结构不精确问题。
3.3 基于 AIRR 数据的预测分析
AIRR 既是健康与疾病的决定因素,也是其传感器;然而,其复杂的结构使得与抗原结合或由此产生的免疫反应相关的特征难以被直接提取,而这些特征恰恰决定了免疫相关结局。本文将这些 AIRR 特征统称为“AIRR 基序”。这些免疫信号通常位于 CDR3 区域。机器学习工具利用模式识别与函数逼近技术,从(大量)数据中识别组内模式,早在十余年前就被提出用于 AIRR 预测性分析。机器学习可以发现统计关联,例如 AIRR 数据与免疫状态或表位结合之间的关系,理想情况下,这些关联能够实现可泛化的预测,不仅追求高预测性能,也期望获得对 AIR 生物学的生物学洞见。因此,人们希望机器学习模型具备可解释性。目前,可用于研究免疫信号如何在 AIRR 中编码的机器学习与深度学习方法激增,图 4b–d 展示了这些方法。
基于序列与基于组库的机器学习应用
基于 AIRR 的机器学习大致可分为基于组库和基于序列的任务,部分工具列于补充表 2(非穷尽)。基于序列的机器学习聚焦于利用序列级标签(如抗原(表位)特异性或人群水平共享出现)对 AIR 序列进行分类。其预测可用于药物发现、抗原治疗药物、抗体治疗药物与 TCR 治疗药物的计算机设计,或潜在地用于组库范围的抗原特异性序列注释。基于组库的机器学习则强调利用 AIRR 进行分类与供体免疫状态预测,包括识别疾病存在、近期疫苗接种或特定病原体暴露史等因素,在免疫诊断领域具有重要应用价值。此外,基于组库的机器学习也可用于推断与疾病状态相关的 AIR 或 AIR 序列基序。
AIRR 数据编码与嵌入用于 AIRR 机器学习分析 AIR 序列是长度不一的氨基酸链。数据编码即为每个氨基酸分配数值,使序列可被机器学习算法使用。编码方式多样:one-hot、k-mer、氨基酸尺度或整序列编码。近来,神经网络生成“嵌入”——在高维向量空间表示序列,相似序列邻近,不相似远离(相似性可非序列编辑距离,而是如结合相似性)。嵌入源于自然语言处理,表单词语义。训练神经网络预测目标变量可获嵌入。近期,基于数百万蛋白或 AIR 序列的蛋白语言模型,可捕获长程依赖,用于嵌入、聚类、预测及生成功能。联合编码序列与结构可提升抗体及 TCR 的互补位-表位相互作用预测;联合嵌入 AIR 序列与转录组谱可揭示 TCR 序列与转录组互依,识别此前未发现的疾病特异性 T 细胞簇。
AIRR 机器学习基本流程 AIRR机器学习基本流程:在选定“基于组库”或“基于序列”的机器学习策略后,AIRR机器学习(AIRR-ML)的基本流程始终包含以下环节:数据准备——收集并清洗数据,将其划分为训练集、验证集和测试集;特征工程与特征选择——通过特征工程创建能提升模型效果的数据表示,包括筛选重要变量(特征选择)、数据缩放、归一化、编码等步骤;模型训练——在训练数据上拟合模型,最小化预测输出与真实输出之间的差异;模型评估与调优——在验证数据上计算准确率、精确率、召回率、F1值等指标,根据结果调整超参数、学习率或更换算法以提升性能;最终测试——使用测试集评估模型的泛化能力,确保无过拟合,并可采用交叉验证进一步确认;迭代优化——整个流程是迭代的,可根据评估结果返回前面的任意步骤进行调整(如嵌套交叉验证);为简化上述流程,开发者推出了开源软件生态immuneML,提供命令行与直观的Galaxy Web界面,包含完整、可复现、可共享的工作流文档,支持大规模AIRR-ML方法基准测试,帮助发现当前研究空白,指引未来方向。
4. 应用
AIRR 分析目前被用于解决多种基础与生物医学问题。本节按“结果”部分详述的分析方法类型,对应展示其应用;图 5 给出合成示意图。
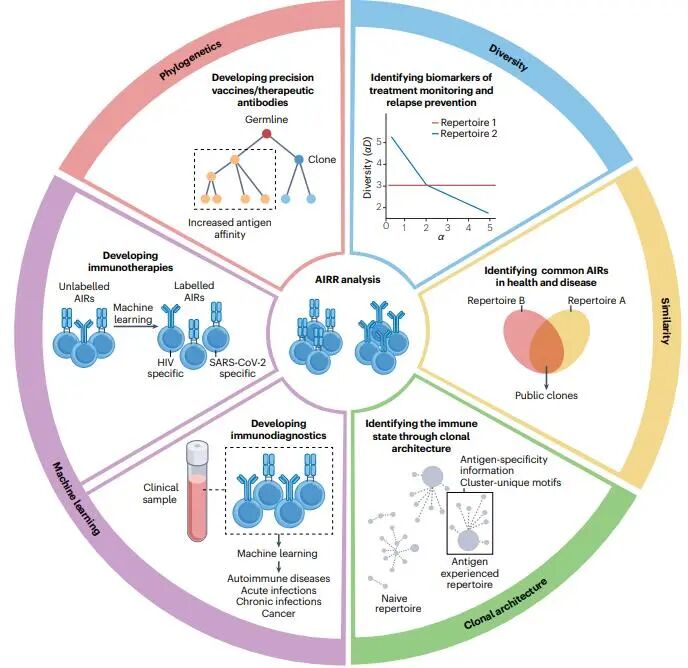
图5 | AIRR分析在疾病诊断和免疫治疗发展中的应用。
4.1 AIRR 多样性
产生巨大 AIRR 多样性的机制仍未完全阐明。然而,一条给定 AIR 序列通过 V(D)J 重组被生成的概率(即生成概率,Pgen)可以量化。已开发出一种基于非生产性重排学习的概率模型,用于估算每次重排事件的生成概率,涵盖片段选择、基因修剪、核苷酸插入及链配对。该模型实现在 OLGA(免疫球蛋白氨基酸序列优化似然估计)工具中,可为任意 TCR 或 BCR CDR3 序列赋予 Pgen 值。OLGA 的应用显示,并非所有重排都以相同概率产生;某些高概率重排特异性针对病毒表位。一致地,研究发现胸腺优先生成能与多种无关人类病毒互作的 TCR。这些观察表明,AIRR 并非随机多样,而是偏向高度保护且平衡的状态。更近的研究显示,单卵双胞胎或近交小鼠的免疫球蛋白 V(D)J 重组规则及序列生成概率存在差异,提示表观遗传等非遗传因素会影响重组过程,进一步彰显 AIRR 的复杂性。值得注意的是,当前的库生成模型尚未考虑新近发现的胚系 AIR 基因多态性对 V(D)J 基因使用及功能性 B 细胞与 T 细胞库的影响。
AIRR 的多样性首先受到胸腺(针对 T 细胞)和骨髓(针对 B 细胞)内选择事件的塑造,随后又因外周抗原暴露而进一步加剧。在小鼠中,浆细胞(已遭遇抗原)与前 B 细胞及脾脏初始 B 细胞(未遭遇抗原)之间的组库多样性差异显著,突显了抗原 encounter 对克隆分布的影响。BCR 组库的差异既可源于 B 细胞克隆扩增,也可来自类别转换重组或体细胞高频突变(SHM)。事实上,虽然多数高度扩增的克隆型被证明具有抗原特异性,但在组库的其余部分,抗原结合与非结合克隆型大致各占一半;并且在多克隆或单克隆谱系水平上,抗体-抗原结合亲和力与克隆扩增程度或 SHM 数量并无相关性。由于 TCR 组库不发生 SHM,研究者通过比较小鼠生理状态下初始与活化的调节性 T 细胞组库发现,活化后克隆多样性降低,主要因克隆扩增增多所致。
在病理背景下,探究 AIRR 多样性可揭示不同适应性免疫细胞亚群的动态变化及其在特定疾病中的作用,并有助于监测患者对治疗的反应。例如,有研究报道多发性硬化症患者脑脊液和外周血 CD8⁺ TRB 重排组库中克隆扩增增加;在 1 型糖尿病中也观察到类似现象,患者胰岛和淋巴结内 TCRβ 链组库多样性下降。这些结果共同支持“该病发病依赖于 T 细胞、并由潜在组织特异性抗原驱动”的观点。克隆扩增增加亦见于克罗恩病和系统性红斑狼疮患者的 BCR 组库,而在 ANCA 相关血管炎或 IgA 血管炎患者中则未观察到此种变化。
4.2 克隆架构
通过研究AIR序列相似性,可揭示健康状态下组库的克隆架构及其在病理条件下的动态变化。AIR可依据序列相似度、共享氨基酸基序和/或理化性质进行聚类。研究发现,初始B细胞与T细胞组库围绕保守的公共序列形成高度连接的网络,这与组库生成和选择的偏倚有关;相反,抗原经验细胞的组库在个体间序列相似度较低,反映个人抗原刺激史及正在进行的免疫应答。抗原挑战、HIV或SARS-CoV-2感染、治疗性抗体干预以及肿瘤发展过程中,均观察到克隆架构的改变。此外,将序列连接度与V(D)J重组统计的基线预期比较,可识别接受癌症免疫治疗患者及SARS-CoV-2感染者外周血中扩增或收缩的克隆型。研究还表明,相似序列极可能识别同一肽-MHC配体,从而形成特异性群组;将这些序列与带抗原注释的公共数据集匹配,可推断群组的抗原特异性。该策略已用于识别在病毒感染、自身免疫病和癌症患者中富集、而健康供体中缺乏的序列群组。总之,在病理背景下研究AIRR序列架构,单独或结合公共数据库中功能验证的抗原特异性序列,有助于识别与特定疾病相关的免疫应答基序、AIR特异性及亲和力。
4.3 B细胞系统发育
构建系统发育谱系树有助于解答B细胞克隆扩增、体细胞高频突变(SHM)及抗原驱动选择等问题。例如,B细胞系统发育已用于分析HIV感染期间B细胞亚群的分化,或食物过敏情境下连续的同种型转换。此外,一项针对可测量免疫球蛋白序列进化的系统发育检验发现,流感疫苗接种后仅在生发中心B细胞中检测到可测量的进化,从而否定了“季节性流感疫苗效果差是因为无法诱导流感特异性B细胞进化”的假设。
还有研究利用系统发育方法识别共享共同未突变祖先的序列,以预测抗体亲和力及关键的高亲和力突变。将量化树差异的系统发育方法(Unifrac)应用于年轻与老年个体在基线及流感疫苗接种期间的组库,发现免疫衰老——即B细胞组库的老化——与初始库收缩及谱系内多样性减少相关。
另一例中,多发性硬化症患者中枢神经系统内驻留B细胞的亲本克隆被证实起源于外周,这提出外周激活的淋巴细胞可能是该病主要驱动因素的问题。理解这一过程有助于阐明针对外周B细胞的治疗如何影响浸润患病组织的B细胞。
4.4 4 AIRR组成相似性
如前所述,公共克隆型常在个体内部及个体间的组库中出现,其存在可归因于偏好性重组和/或中枢选择,以及感染后或慢性疾病背景下的外周抗原特异性选择。因此,鉴定并表征公共克隆型可揭示健康与疾病状态下的共同受体选择模式。例如,胸腺选择后,胸腺细胞最丰富TCR序列内的CDR3β重叠增加,凸显胸腺选择对特定序列的偏好作用,无论这些序列位于何种细胞亚群。
无关个体间也存在公共克隆型,尽管双胞胎共享比例更高。重要的是,公共克隆型是疫苗接种、感染、自身免疫病及恶性肿瘤免疫应答的核心组成部分。例如,暴露于同一抗原的人类出现趋同BCR进化,产生携带保护性抗体的公共克隆型,这些抗体可能特异性针对挑战抗原,未来可用于治疗性抗体开发。
尽管已观察到BCR与TCR多样性随年龄增长而下降,但短期内或长期内组库动态变化的研究仍较少。研究显示,个体记忆B细胞亚群在数月时间跨度内具有高度克隆持续性。此外,在感染前时间点于记忆区室中检出部分SARS-CoV-2反应性T细胞克隆,表明预先存在的交叉反应记忆T细胞参与了针对SARS-CoV-2的免疫应答。更广泛而言,对健康个体进行为期1个月的BCR组库分析揭示了个体内部及个体间的显著变异。
4.5 AIRR-ML 应用
基于高水平的抗体和T细胞受体多样性以及个体之间不可忽视的AIR序列相似性,使免疫学家建议,趋同的AIRR特征可能有助于整体健康状态的维持,而这些特征的最终富集或丧失可能会导致稳态的丧失。机器学习方法可以用来识别这些特征。在免疫库层面,AIRR可用于疾病分类的首个有力证明是通过从大约600名CMV+和CMV–个体的外周血中识别出公共TCRβ链特征。独立队列中的记忆CD4+ T细胞也发现了类似的模式,提供了公共TCR与病原体特异性T细胞反应密切相关的证据。有趣的是,CMV特异性TCRβ链特征仅由164个序列组成,当仅使用原始数据的三分之一时,分类准确率从>90%降至近乎随机频率(≈50%),这表明,大规模数据集对于检测与免疫状态相关的免疫信号是必要的。
确实,针对~1,000个合成AIRR数据集的机器学习分析,这些数据集包含约250,000个AIRR,涉及信号出现频率和免疫库大小等不同参数,显示出相对简单的机器学习算法,如L1惩罚逻辑回归,即使在公共克隆型仅出现在50,000个AIR序列中的1次时,也能够达到较高的预测准确性。目前,仅存在少量大规模的AIRR数据集。从877名系统性红斑狼疮患者和206名类风湿性关节炎患者的数据集中,可以基于TCRβ链免疫库区分这些自身免疫疾病,而来自1815名COVID-19患者和3500名健康个体的AIRR数据则揭示了COVID-19特有的模式,这些模式在诊断后的早期和康复后均可见。虽然这些方法仅依赖于通过共享公共克隆型检测免疫状态(检测疾病),但一种较新的方法利用了三种不同的机器学习表示,包括总体AIRR组成、通过编辑距离进行的抗原特异性序列的趋同聚类以及从BCR和TCR序列中提取语言模型特征,以对SARS-CoV-2(n = 63)、HIV(n = 95)和系统性红斑狼疮(n = 86)以及健康对照组(n = 217)进行分类。
AIR-抗原结合的序列预测可以在序列层面或结构层面(或混合方法)进行。大多数基于序列的方法已被应用于预测AIR-抗原结合的问题(例如抗体-抗原预测或TCR-pMHC预测)。关于抗体-抗原结合和TCR-pMHC预测已经发布了大量的综述。由于T细胞表位主要是线性的,因此TCR-pMHC结合预测方法主要涉及基于序列的预测,只有一些较新的聚类和机器学习方法也探索了结构数据的整合。除了基于序列的AIR-抗原结合预测外,基于AIRR序列的模拟工具,如IGoR、OLGA和immuneSIM,使得以适度的计算资源生成大量AIRR序列成为可能。这些工具的优势在于生成的序列数据几乎与实验数据相同。特别是,ImmuneSIM、simAIRR和LIgO允许将序列基序纳入生成的序列,从而使与抗原结合相关的基序建模成为可能。因此,这些模拟数据可用于预测AIR特异性的任务,无论是以二元还是多类别的方式进行。这些预测涉及根据其抗原结合行为对序列进行分类,或者用于AIRR基础的机器学习,应用于免疫诊断。
然而,尽管基于序列的数据集相对比结构数据集更容易生成,但基于序列的机器学习往往缺乏对结合位点和构象表位结合的细致描述,这使得解析构象抗体-抗原结合变得困难。结构信息要么隐式地用于通过促进表位识别来构建特征(例如使用带缺口的k-mer编码),要么显式地直接整合到机器学习任务中。通过深度突变扫描,可以获得关于残基对结合影响的更细致信息,这揭示了一个复杂的AIR-抗原结合景观。AIR-抗原结合预测的一个关键未来应用是在计算机上对AIRR数据集进行抗原结合信息的注释。这使得能够进行抗原或表位特异性的定量诊断分析,并进行个体、抗原和免疫状态之间的比较。除了AIR-抗原结合预测,基于序列的AIRR-ML方法还可以应用于设计新的AIR序列。这可能用于模拟、改进免疫治疗剂的设计或抗体药物开发。
5. 可重复性与数据存储
AIRR-seq数据的快速积累和巨大潜力促使科学家与产业界人士共同制定了AIRR研究的实验与计算分析标准及对照体系,并推动数据存储与共享的规范化。AIRR-C(AIRR社群)于2015年成立,是一个以研究型组织,负责协调高通量测序(HTS)技术的使用,以统一AIRR-seq研究设计。其核心使命是为AIRR-seq数据的生成、注释和存储制定指南与标准,便于更广泛的研究群体使用。
5.1 实验可重复性
如同任何新兴研究领域,AIRR研究所涉及的实验流程在初期发展迅速却缺乏标准化,这阻碍了跨研究、跨数据集的比较分析。此外,实验本身高度复杂——包括生物样本保存、流式细胞术、(靶向)核酸提取、引物设计与浓度、PCR反应、测序技术等诸多环节——凸显了AIRR-seq文库制备和数据分析过程中可能出现的多种偏倚与误差。因此,亟需建立AIRR-seq数据生成的标准与对照,以提供关键的可重复性保障并最大限度减少实验误差。 这一迫切需求促成了AIRR-C内部“生物资源工作组”的成立,其目标是制定对照策略,推动AIRR-seq研究的标准化。这些策略虽并非适用于所有实验平台或场景,但包括:使用样本特异性条形码以检测样本交叉污染;在可获得的情况下,采用标准化样本制备试剂盒(无论是商品化还是定制),因其可提供标准化的分析材料和优化的实验流程;在AIRR-seq文库中掺入“加标”对照序列。 其中,加标对照序列策略颇具前景,但仅当这些对照序列能模拟AIRRs的天然多样性与复杂性,同时又能与样本中的AIRRs明确区分时,才能发挥最大效用。此外,设置并行生物对照(例如可更好捕捉AIRs多样性的人类淋巴样细胞混合物)可在AIRR-seq文库构建与测序的每一步进行质量监控。然而,该对照中的基因重排序列并非预先定义,在出现PCR和/或样本污染时可能带来问题。
5.2 数据共享与计算可重复性
随着 AIRR-seq 数据量激增,向社区提供原始数据集及其配套元数据,可促进这些数据在二次分析中的使用,或将多组同类数据整合以获得更大统计效能,从而推动计算策略(尤其是机器学习方法)的进步,并催生新的科学发现。依据 FAIR 原则(可发现、可获取、可互操作、可重用)共享 AIRR-seq 数据,是保证数据质量可靠、准确的重要途径。尽管越来越多同行评议期刊要求公开原始数据,但截至 2022 年,仅 38.1% 的 TCR 测序研究真正提供了原始数据,前路仍长。 元数据格式的标准化被视为鼓励研究者共享原始数据的“钥匙”——只有建立一套简洁统一的数据库生态,实现输入/输出的无缝读写,才能降低共享门槛。为此,AIRR-C 制定了数据标准(MiAIRR 与 AIRR 文件格式),确保数据在共享存储库中具备可重复性、统一质控与规范存取。该标准覆盖 AIRR-seq 数据及元数据的发布、整理与共享,元数据字段包括:研究信息、受试者信息、样本采集与处理、测序参数、原始序列、序列数据处理流程及最终 AIRR 序列等。 值得注意的是,符合 AIRR 文件格式标准的数据可直接提交至美国国立生物技术信息中心(NCBI)(参见《AIRR-seq 数据提交 NCBI 指南》)。为进一步便利共享,AIRR-C 还建立了“AIRR 数据共享体(ADC)”,它由地理上分散、遵循 AIRR 标准的存储库组成。ADC 提供基于 Web 的查询 API,使研究者能够轻松发现与获取 ADC 中的 AIRR-seq 研究及其配套注释序列数据。借助 MiAIRR 标准与 AIRR 文件格式,ADC 显著提升了数据的互操作性与重用率,促进可重复性并支持荟萃分析。用户可通过 iReceptor Gateway 网页界面交互式探索 ADC。 除大规模数据库(多为非抗原注释数据)外,还有若干小型数据库专门提供 TCR或 BCR的抗原注释信息(见补充表 3)。 AIRR-C 同样为 AIRR 软件工具制定了标准,确保数据标准能被无缝使用。凡符合上述标准的工具(标准细则见 AIRR-C 官网)均可被标记为“AIRR 兼容”。目前,已有 9 款软件通过 AIRR 兼容性认证。
6.局限与优化
过去几年,AIRR-seq 领域发展迅猛,极大提升了我们对疾病如何影响适应性免疫应答的理解。然而,当前仍存在若干技术层面的局限(图 6)。此处“局限”泛指数据生成与解读过程中广义的不足。下文“展望”部分将具体说明如何在 AIRR 领域逐一克服这些短板。
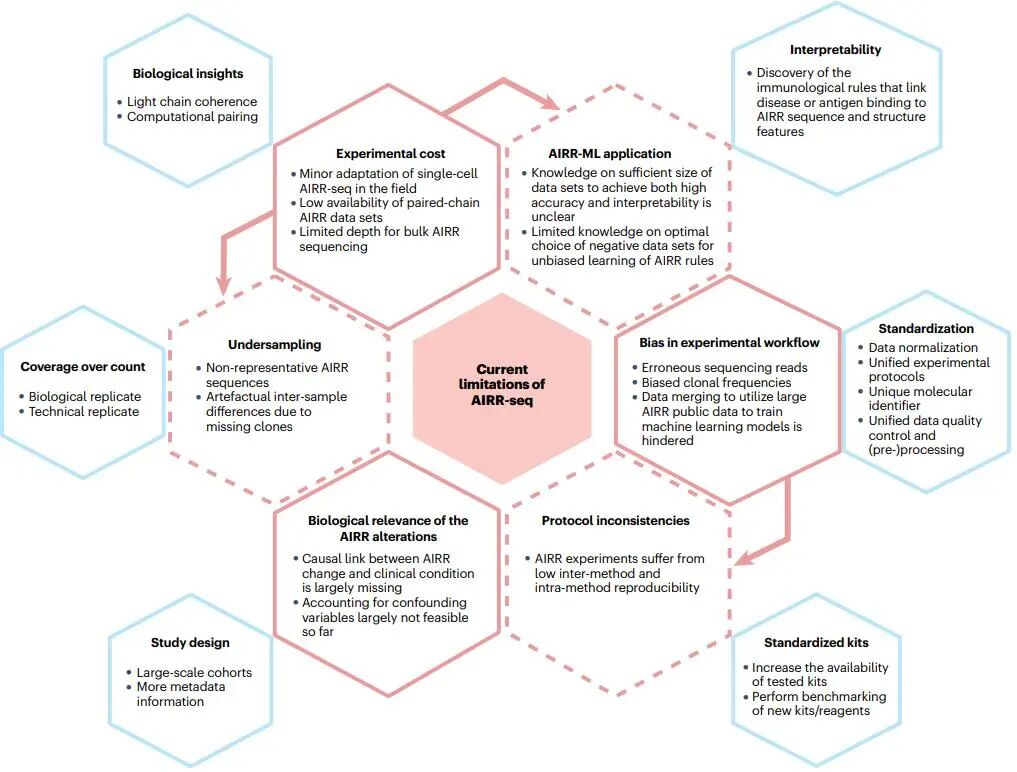
图6 | AIRR-seq领域的当前局限及其解决方法
6.1 实验偏倚与数据归一化
技术与生物偏倚使得不同 repertoires 之间的比较极具挑战;样本来源(不同个体、不同生物学区室、不同细胞亚群)本身就带来不可避免的生物学异质性。如“实验设计”节所述,测序流程各环节(图 3)均可引入实验偏倚,例如 RNA 提取、逆转录及 PCR 扩增步骤。逆转录和 PCR 过程中产生的核苷酸错配、扩增偏好等误差,会直接影响定量准确性及 AIR 多样性评估。为识别并减轻这些偏倚,可采取以下策略:在文库构建阶段掺入 spike-in 对照与 UMI(唯一分子标识符);采用 NoisET 等生物信息算法降低扩增偏倚,该算法已用于 AIRR-seq 实验。 不同实验方案会引入不同类型的偏倚,方案间不一致导致数据集内部与数据集间的可重复性低下,进而阻碍 AIRR 数据的比较与整合。优先使用商业化试剂盒而非自制“homebrew”流程,有助于减少实验不一致。偏倚亦可通过计算手段校正:iROAR 工具旨在生成可长期合并、跨研究比较的“常绿”数据集;AIRR-ML 方法需大样本量及高质量注释元数据方能获得最佳预测精度,因此能够校正批次效应、整合现有公共数据,对训练机器学习模型极为宝贵。目前,AIRR-seq 研究尚缺乏系统性的数学/统计方法来校正实验偏倚。已有研究提出基于 Shannon 指数过滤无信息 TCR 读段的方法,但未对读段计数进行校正。可借鉴其他高通量测序(HTS)领域的归一化策略,例如转录组或微生物组测序。事实上,与微生物组数据类似,AIRR 数据同样具有高物种多样性、数据稀疏性及样本间重叠度低的特点。
6.2 实验成本
单细胞 AIRR-seq 的高昂费用,导致其在学术研究中的普及度仍低,公共数据库中也罕见已配对链且已注释特异性的 AIRR 数据。为此,研究者正开发数学方法,通过克隆频率估计与半 bulk 测序中给定链对的观测概率,间接推断 TCR 配对信息。为进一步提升准确性,科学家还在探索链配对的内在规律),但迄今尚未发现普适的 AIR 配对规则。
在 bulk 测序中,RNA 法需更高测序深度,成本同样居高不下,易造成“采样不足”,即每份样本的读段覆盖度不够。简单加大深度并非良策——这会引入更多测序错误,导致克隆大小或 BCR 的 SHM 谱型失真,甚至因噪声改变小样本的克隆分布。目前最佳替代方案是设置技术性和/或生物学重复,以辅助检出稀有克隆并校正噪声。
预测 AIR 识别的抗原表位对免疫治疗开发极具价值。现有预测多基于序列,但公共数据库中带抗原注释的序列数量,与所有可能的受体–表位对之高多样性相比杯水车薪,成为训练精准机器学习模型的瓶颈。可能的应对策略包括:采用通用 AIR–表位识别模型,先揭示结合模式与规律;然而,表位单点突变即可改变结合范围与受体亲和力,需为每个可用表位单独训练模型,而数据库中表位分布极不均衡,使该路线困难重重。近年兴起的深度突变扫描(DMS)实验,通过系统突变表位并测量结合,可建模 paratope–epitope 互作,为解析复杂的 AIR–抗原结合景观提供新视角。
6.3 生物学因果关系的验证
AIRR-seq 分析已揭示与临床表型或遗传背景(参考文献 385–387)的诸多关联,但迄今未能建立因果链条。突破这一局限,可能需要:引入基于生物学的 AIRR 数据编码策略;构建因果推断模型。根本障碍在于缺乏具备完整元数据的大规模队列,导致难以控制样本选择及年龄、性别等混杂变量,从而限制了因果驱动的研究规模。
6.4 AIRR-ML 优化
无论是基于序列还是基于整个 repertoires 的机器学习基准测试都表明,当前方法仍需大幅优化:最小样本量下限尚未评估;需要开发适配 AIRR 生物学特性的算法,以捕捉更复杂的 repertoire 模式;最佳“负样本”集合仍待定义;必须设计无偏策略来估计模型性能。
此外,序列层面与 repertoire 层面的 AIRR-ML 结果都难以解释。高预测精度仅提示数据中存在能把两类标签(如健康/疾病、结合/非结合)分开的免疫信号,下一步必须弄清究竟是哪些 AIRR 特征贡献了这种精度(例如结合规则)。可解释性与数据编码方式及模型架构紧密相关:把序列切成 k-mer 可能破坏氨基酸间的相互作用,从而丢失生物学信息; 模型越复杂,越难把特征与标签建立直观联系。因此,为便于解释,有时应主动选择更简单的架构:仅使用具有不同生物物理化学特性的氨基酸 3-mer 加上富集的 V/J 基因,就足以区分乳糜泻患者与健康人;仅取 CDRH3 的特定子区域便可区分公共与私有克隆型;仅基于特定胚系 V 基因就能刻画 CDR3 在不同免疫状态下的变化,并按序列与某一免疫状态的相关概率排序。
研究表明,对可解释性结果的分析可能受到“确认偏倚”的干扰:研究者会不自觉地把先入为主的信念和假设带入分析过程。为避免这一问题,必须用“金标准”(通常是合成数据)来验证:从候选解释中提炼出的假设,是否真正反映了模型预期的逻辑
7. 展望
7.1 将抗体蛋白质组学整合进 AIRR 研究
利用质谱等蛋白质组学手段,可在血液或黏膜组织中直接分析抗体多样性。把 bulk 与单细胞 BCR 测序同抗体蛋白质谱结合,有望完整捕捉体液免疫全貌。得益于深度学习,新兴的“从头蛋白测序”技术可能颠覆传统抗体分析——它无需依赖潜在偏倚的 BCR 数据库去做谱图反卷积,即可直接获得抗体序列。
7.2 深入理解病理生理
高通量抗原注释与结合预测。当前公共 AIR 序列与结构数据库虽快速膨胀,但绝大多数条目缺乏抗原结合注释,这限制了我们认识:不同个体、不同免疫状态下抗原特异性的变化; 针对各类抗原的 AIR 出现频率。单细胞抗原特异性 AIR 测序、结构生物学技术,以及系统免疫学、统计与机器学习等交叉学科的进步,正开始提供破解这些难题的钥匙。尤其单细胞测序已帮助鉴定高达 20 % 的表达双 TCR 链 T 细胞,为阐明多重 TCR 链表达机制并预测表位特异性奠定基础。
大规模带抗原注释的 AIRR 数据可能很快会催生计算与机器学习方法,只需 AIR 序列或结构即可预测抗原特异性,从而把目前公共 AIRR 库中无法触及的抗原特异性信息解锁出来。重要的是,这些途径将进一步加深我们对 AIRR 交叉反应性的理解。
过去十年,已出现众多竞赛,旨在解决有关蛋白结构、相互作用与功能预测的悬而未决问题,例如结构预测关键评估(CASP)或相互作用预测关键评估(CAPRI),或基于人工智能的图像识别竞赛如 ImageNet 等。这些竞赛促成了 AlphaFold 等突破性发现,也可能帮助我们打磨目前仍显粗糙的抗原特异性适应性免疫预测工具。
将 AIRR 数据与转录组数据整合。近期已采用 AIRR 与转录组联合分析,以更好理解细胞的转录谱如何与 AIR-抗原结合相关联。为此,若干研究团队已报道了整合 AIRR 与转录组的方法。初步结果表明,抗原结合特异性与转录谱可能存在关联,且转录组信息可能提高 pMHC-TCR 表位预测精度。未来仍需阐明 MHC 背景在多大程度上影响转录组与 TCR 序列特异性之间的互作。
适应性免疫中的基因型-表型关联:最近,胚系基因库与体液免疫应答之间的关联已被深入探究。例如,能够激活特定胚系前体(这些前体有很大概率经过亲和力成熟后成为广谱中和抗体)的免疫原,已在针对主要人类病原体的精准疫苗研发中展现前景。针对胚系基因的疫苗所诱导的反应强度,在很大程度上可由不同免疫球蛋白基因型及其对应B细胞的频率来解释,而非免疫原剂量。因此,在临床试验中设计并测试靶向胚系的免疫原时,必须考虑免疫球蛋白等位基因的变异。从免疫学角度看,这些结果表明宿主的遗传变异可调节疫苗诱导的广谱中和抗体应答的强弱。为了设计更具针对性的疫苗,理解胚系基因变异的进化与选择将至关重要。 HLA基因型与多种疾病易感性相关。此外,HLA基因型会塑造个体的TCR库。近期观察到,HLA等位基因可影响TCRβ链的组成,且可基于某些独特TCR的存在来预测HLA类型。然而,我们最近发现,原本被认为受限于特定HLA类型的TCR,也能识别由不匹配的HLA所呈递的抗原。尽管转基因小鼠研究正在推进,但仍需增加更多配对的或非配对的TCRα和TCRβ链深度测序数据集,并结合HLA基因型信息,才能更准确地了解HLA基因型与TCR库之间的关联。
7.3 转化的角度
AIRR-seq 结合先进的统计与数学建模(包括机器学习),现已形成一套工具包,可用于识别与疾病、血清状态或治疗反应相关的 AIRR 特征。这一现象已在多种疾病(癌症、自身免疫病、移植和感染)以及不同治疗策略中得到验证。如今,该领域未来的挑战在于将这些以研究为导向的方法转化为临床应用。在 B 细胞和 T 细胞恶性肿瘤方面,EuroClonality 联盟已通过 AIRR-seq 在诊断与预后评估上取得重大进展。患者护理中的利益相关者支持以及跨学科协作,是取得成功的关键。
目前,bulk 和单细胞方法正被用于挖掘尽可能接近人类抗体库的抗原特异性抗体。最近,生成式机器学习方法——学习抗体“语言”及抗原特异性结合模式——被用于生成新型或优化抗体,从而用计算发现取代实验筛选。此外,过去十年中,过继性 T 细胞疗法(如 CAR-T 或工程化 T 细胞)在非实体瘤中已获成功,靶向 CD20 或 CD19 等淋巴瘤表面抗原的疗法已应用于临床。若将 AIRR-seq 与抗原预测结合,识别出可特异性引导工程化 T 细胞或 CAR-T 细胞攻击肿瘤的靶点,此类疗法有望扩展至实体瘤。单细胞 AIRR-seq 可追踪患者体内 CAR-T 细胞,解析疗效或失败机制。同样,用于治疗自身免疫病的调节性 CAR-T 细胞正在开发中,调节性 T 细胞疗法也将受益于旨在提高靶向效率的 AIRR-seq 分析。
总之,我们认为 AIRR 领域需超越当前主流的“抗原不可知”序列分析,下一前沿是完全带有抗原注释的 AIRR 数据分析。唯有达成此目标,我们才能真正理解健康与疾病状态下适应性免疫的特异性与功能。为此,必须在高通量 AIRR 数据生成与计算分析上取得新突破。此外,尽管 AIRR 领域已成功借鉴生态学等其他领域的多样性分析等成熟概念,但仍需更丰富的视角。例如,鉴于最新发现表明胚系基因多样性广泛且与免疫相关,理解进化如何塑造人类免疫系统,或将催生基于进化医学的 AIRR 诊疗新策略。
名称解释
Adaptive immune receptor repertoire (AIRR): 适应性免疫受体组库 单个个体在某一时间点的全部适应性免疫受体集合。
Adaptive immune receptors (AIRs): 适应性免疫受体 B细胞受体、抗体和T细胞受体的统称。
Class-switch recombinations: 类别转换重组 增殖中的B细胞通过重排免疫球蛋白重链恒定区基因,改变所产生抗体类别而不改变抗原特异性的过程。
Clonotypes: 克隆型 定义范围从CDR3氨基酸序列到序列簇或整个可变区序列,尚无统一标准。
Epitope: 表位 抗原中被适应性免疫受体识别并直接接触的具体部位。
Generation probability: 产生概率 观察到某一重组后适应性免疫受体序列的概率。
Germline alleles: 胚系等位基因 构成BCR/TCR可变区的V、D、J基因的不同等位变异。
Ground truth: 金标准 训练数据生成过程中的所有参数及其取值均已知且受控的环境。
Paratope: 互补位 适应性免疫受体中直接与表位接触并参与抗原/表位结合的氨基酸集合。
Peptide–MHC complex (pMHC): 肽-MHC复合物 MHC分子将抗原肽呈递于细胞表面,TCR识别该复合物;简称pMHC。
Private clonotypes: 私有克隆型 仅出现在单个个体适应性免疫受体组库中的序列。
Public clonotypes: 公共克隆型 在来自不同个体的多个组库中出现次数大于n(n>1)的适应性免疫受体序列。
Sequencing depth: 测序深度 对给定样本测得的读段总数。
Somatic hypermutations (SHMs): 体细胞高频突变 主要在生发中心内发生的、使已重组BCR可变区发生突变并可能提高抗原亲和力的过程。
Unique dual indexes: 唯一双索引 用于测序后过滤索引跳跃或错配读段的i5与i7索引引物唯一配对。
Unique molecular identifiers (UMIs): 唯一分子标识符 建库时加在DNA/RNA片段上的短序列,用于标识原始分子,减少PCR扩增带来的误差与定量偏倚。文献来源:
Mhanna, V., Bashour, H., Lê Quý, K. et al. Adaptive immune receptor repertoire analysis. Nat Rev Methods Primers 4, 6 (2024). https://doi.org/10.1038/s43586-023-00284-1
本文参与 腾讯云自媒体同步曝光计划,分享自微信公众号。
原始发表:2025-10-29,如有侵权请联系 cloudcommunity@tencent.com 删除
评论
登录后参与评论
推荐阅读
目录

