Nat. Struct. Mol. Biol. | AlphaFold 数据库正在“变老”: 为什么 2 亿结构模型也会过时?
Nat. Struct. Mol. Biol. | AlphaFold 数据库正在“变老”: 为什么 2 亿结构模型也会过时?

DrugAI
发布于 2026-01-06 14:01:36
发布于 2026-01-06 14:01:36
DRUGONE
自 2022 年 AlphaFold 数据库(AlphaFoldDB)公开以来,它已为超过 2 亿条 UniProt 蛋白序列提供了预测的三维结构。作为结构生物学与 AI 制药的关键基础设施,AlphaFoldDB 被广泛应用于结构解析、突变效应预测、分子设计等众多任务。

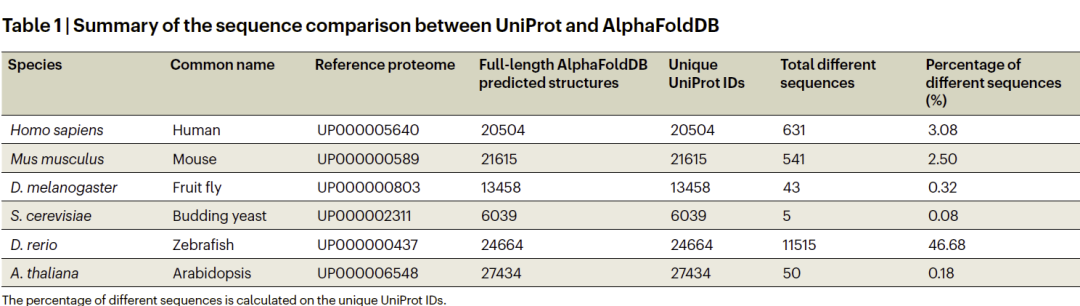
然而,随着研究人员深入分析,这个庞大的结构资源正在悄悄老化。尤其在人类蛋白组的 20,504 个 AlphaFold 结构中,有 631 条序列已经与 2025 年的最新 UniProt 数据不一致——意味着3.08% 的结构模型不再与当前的真实蛋白序列对应。
AlphaFoldDB 的“时间差”:为什么结构会变得过时?
AlphaFoldDB 的结构是基于 2021 年 4 月版 UniProt 序列生成的。但 UniProt 会持续升级数据库,包括:
- 纠正注释错误
- 合并冗余序列
- 更新基因信息、物种注释
- 根据新实验数据调整序列
两者更新频率不一致,使得 AlphaFoldDB 不可避免地出现“时效性问题”。
研究人员对六大模式生物进行比对后发现:
- 人类与小鼠序列更新最频繁(差异分别为 3.08% 和 2.5%),因为相关研究最活跃。
- 果蝇与拟南芥差异很小(0.32% 和 0.18%)。
- 酵母仅 0.08%,几乎不受影响。
- 斑马鱼差异竟高达 46.68%,原因是 UniProt 对其蛋白序列进行了大规模清理。
这说明:随着时间推移,AlphaFoldDB 与生物学“真实世界”的差距会越来越大。
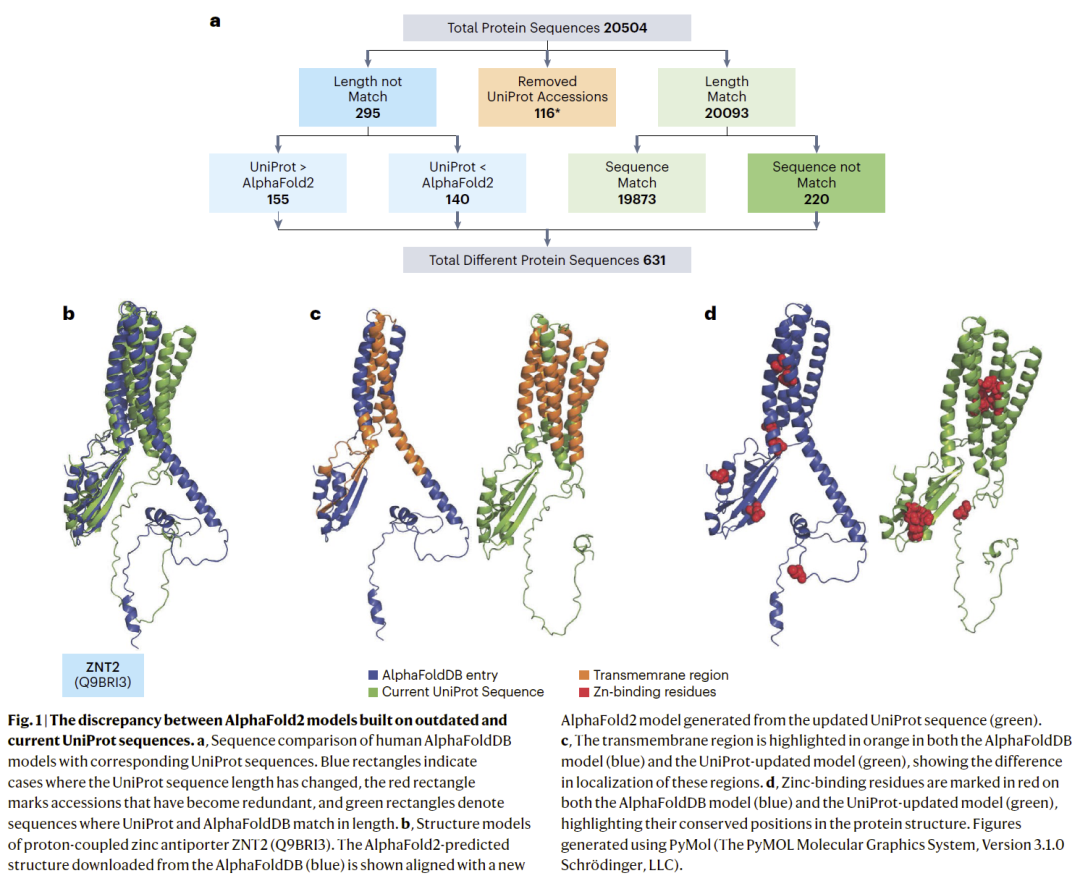
结构真的会变吗?以 ZNT2 为例
研究人员发现部分蛋白的序列不仅老化,还发生了实质性变化。
例如:
- ZNT2(Q9BRI3)是一种与新生儿短暂性锌缺乏相关的锌转运蛋白。
- 新版本 UniProt 在约 100 位点处新增了 50 个氨基酸插入片段。
- 当研究人员利用最新序列重新构建 AlphaFold2 结构时,发现:
跨膜区位置改变
金属结合位点也发生位移
这些变化足以影响功能解析、突变预测乃至药物靶点设计。
此外,有 221 条蛋白虽然序列长度未变,但氨基酸序列发生差异,如 serpin B11 在新版中出现 6 处残基替换。
更重要的是:406 条存在差异的蛋白具有高注释评分,512 条属于“已审核”蛋白,136 条已知与疾病相关。这意味着,模型老化影响的并不是无关紧要的蛋白,而是许多关键蛋白。
数据库“老化”是普遍现象,而非 AlphaFold 独有
斑马鱼的极端案例更突显该问题的普遍性:
- 由于 UniProt 近年大规模清理,斑马鱼的审核蛋白从原先的数千条减少到 3,355 条。
- 其中 93 条不在 AlphaFoldDB 中,一些甚至已在注释更新后获得“已审核”身份,但 AlphaFoldDB 仍停留在旧版本。
这意味着:结构数据库不仅序列会老化,注释也会老化。AlphaFoldDB 更新不够频繁,而基于其构建的各种工具、子数据库同样存在这一问题。
如何避免使用“过时结构”?研究人员给出的建议
为确保研究准确性,研究人员提出以下策略:
1. 始终以 UniProt 为主参考,检查序列是否仍然匹配
许多机构(如 UniProt、Swiss-Model)已经开始添加标记,专门提醒用户结构是否对应旧序列。
2. 必要时重新运行 AlphaFold2 生成模型
对于药物研发、功能位点预测、突变效应等关键场景,重新预测结构更可靠。
3. 使用同步工具,如 AlphaSync
这是目前用于更新 AlphaFold 模型的社区努力工具,正在对老旧模型进行重新建模。
4. 注意其他静态数据库也会老化
包括 missense 预测数据库(如 AlphaMissense),这些同样需要与最新序列比对。
最新进展:AlphaFoldDB 已发布更新版(2025)
研究人员在准备文章时,EMBL-EBI 宣布了 与 2025 年最新 UniProt 序列完全对应的 AlphaFoldDB 新版本。
这对社区是重大利好,但仍需注意:
- 各类衍生工具、模型库、第三方数据库可能 仍基于旧版本
- 这些资源 未必 会提示用户模型对应过期序列
- 风险在实际研究中依然存在
换句话说:AlphaFoldDB 更新了,但生态系统还未全部更新。
结语:时间在生物信息学中流逝得更快
AlphaFoldDB 是结构生物学史上革命性的资源,但它并非永恒不变。
- 序列会变
- 注释会变
- 生物学认识会变
在这个快速更新的时代,AI 结构模型也会“变老”。
保持数据同步、建立可持续更新的生态系统,将是未来 AI 生物信息学必须面对的重要挑战。
整理 | DrugOne团队
参考资料
Tsitsa, I., Conev, A., David, A. et al. The aging of the AlphaFold database. Nat Struct Mol Biol (2025).
https://doi.org/10.1038/s41594-025-01725-z
内容为【DrugOne】公众号原创|转载请注明来源
本文参与 腾讯云自媒体同步曝光计划,分享自微信公众号。
原始发表:2025-12-11,如有侵权请联系 cloudcommunity@tencent.com 删除
评论
登录后参与评论
推荐阅读

