J. Med. Chem. | CLaSP:对比学习引导的潜在评分平台
J. Med. Chem. | CLaSP:对比学习引导的潜在评分平台
用户1151118
发布于 2026-01-08 13:02:22
发布于 2026-01-08 13:02:22
Contrastive Learning-Guided Latent Scoring Platform for Comprehensive Drug-Likeness Evaluation
期刊: Journal of Medicinal Chemistry 链接: https://doi.org/10.1021/acs.jmedchem.5c01618 代码: https://github.com/beiweiClover/CLaSP 平台: https://lmmd.ecust.edu.cn/CLaSP 简介: 本文介绍了一种名为 CLaSP 的新型药物类药性评估框架,其创新点在于整合变分自编码与三元组对比学习,解决了现有方法泛化能力和可解释性不足的问题。该方法通过构建结构化潜空间,结合理化性质和 ADMET 相关特征生成连续、可解释的 CLaSP_Score 来评估分子成药性。实验使用了 FDA 批准药物、ZINC、ChEMBL、GDB17 等数据集,基准测试表明 CLaSP 在多个数据集和真实案例中表现优于 QED 和 DBPP-Predictor,且能有效捕捉药物优化轨迹,如在 Wee1 抑制剂的案例中得到体现。此外,研究还提供了用户友好的 Web 平台供早期药物设计使用,该论文为药物类药性评估提供了一种全面、可靠且实用的新工具。

在现代药物研发领域,早期化合物的类药性评估是提升研发效率、降低失败风险的关键环节。传统评估方法在泛化能力、可解释性及综合考量药物关键属性方面存在显著局限。近期,唐赟老师团队在《Journal of Medicinal Chemistry》发表的“CLaSP: A Contrastive Learning-Guided Latent Scoring Platform for Comprehensive Drug-Likeness Evaluation”一文,提出了一种融合变分自编码与三元组对比学习的创新框架,为药物类药性评估提供了全新解决方案。本文将系统剖析这一方法的技术架构、性能优势及实际应用价值。
研究背景:药物类药性评估的现状与挑战
药物研发过程耗时长、成本高,早期筛选阶段有效识别不合适的化合物至关重要。类药性研究通过评估化合物的安全性、有效性和药代动力学等关键属性,为后续优化提供指导,是提升药物发现效率的核心环节。
现有评估方法存在明显缺陷:
- • 经验规则类方法(如Lipinski的“五规则”、Veber规则等):基于经验观察,存在僵化性,定量区分能力有限,难以精准评估候选化合物的类药性差异。
- • 定量评分方法(如QED):虽能定量评估,但仅基于8项理化性质,缺乏对毒性等关键ADMET(吸收、分布、代谢、排泄、毒性)特征的考量。
- • 机器学习分类模型(如DBPP-Predictor):多采用二元分类法,受限于训练数据集的代表性,易出现过拟合,对新型化学实体的预测可靠性不足。
- • 无监督学习方法(如基于RNNs、VAEs的方法):通过潜在相似性间接评估类药性,可解释性差,难以用于结构差异较大的分子优化。
这些局限凸显了开发综合、可解释且稳健的类药性评分方法的迫切需求。
CLaSP的技术架构与工作原理
CLaSP(对比学习引导的潜在评分平台)通过整合对比学习与变分自编码技术,构建了一个结构化的潜在空间,实现了对分子成药性的连续、可解释评估。其核心工作流程包括四个关键步骤:
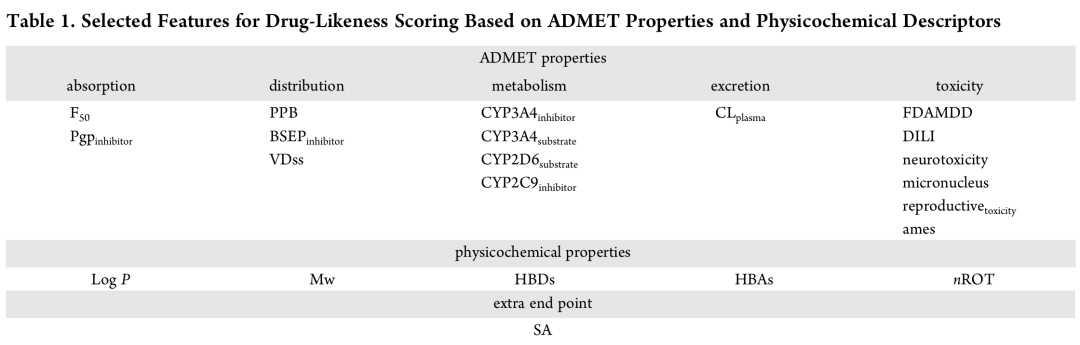
1. ADMET相关特征的筛选与伪标签生成
- • 利用ADMETlab 3.0和admetSAR 3.0获取化合物的ADMET相关特征,生成伪标签。
- • 结合随机森林特征重要性分析和互信息分析进行特征筛选,确保所选特征与类药性高度相关。筛选规则为:在ADMETlab 3.0或admetSAR 3.0的四次比较中至少被选中三次,或在两个平台各两次比较中均被选中,以保证特征的稳健性和跨平台一致性。
- • 最终选定16个代表性ADMET特征(涵盖吸收、分布、代谢、排泄、毒性五大类),并补充关键理化性质(如分子量、氢键供体/受体数量等)和合成可及性评分,形成综合特征集。
2. 集成预测平台的构建
- • 多任务Uni-Mol模型:基于预训练的Uni-Mol分子表征模型,构建多任务学习框架,同时预测16个ADMET相关属性。该模型采用Transformer架构,通过多头自注意力机制捕捉分子特征与属性间的复杂关系,在15个分类任务上的性能优于GAT、GCN、ChemBERTa等基线模型。
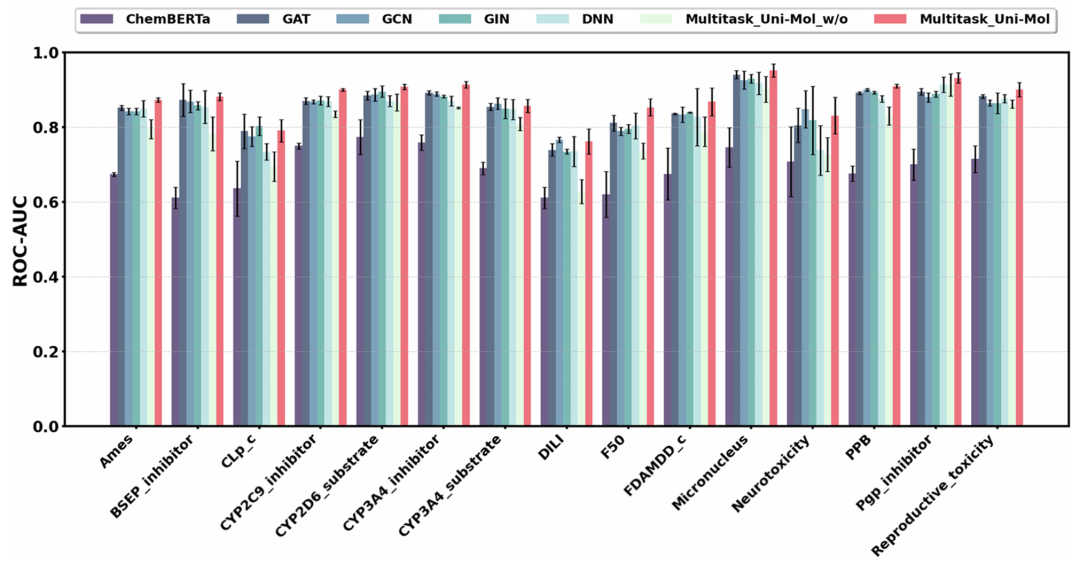
通过 ROC-AUC 指标比较了不同模型在 15 个分类终点上的性能。结果显示,Multitask_Uni-Mol 在大多数任务上优于 ChemBERTa、GAT、GCN、GIN、DNN 及其基于 MLP 的消融模型(Multitask_Uni-Mol_w/o),表明其稳健且一致的预测能力。
通过 ROC-AUC 指标比较了不同模型在 15 个分类终点上的性能。结果显示,Multitask_Uni-Mol 在大多数任务上优于 ChemBERTa、GAT、GCN、GIN、DNN 及其基于 MLP 的消融模型(Multitask_Uni-Mol_w/o),表明其稳健且一致的预测能力。
- • 理化性质与合成可及性计算:利用RDKit工具包计算分子的理化性质(如LogP、分子量等)和合成可及性(SA)评分,SA评分采用Ertl和Schuffenhauer提出的方法,基于分子复杂性和片段贡献评估合成难易程度。
3. 对比学习变分自编码器(CLVAE)的设计
- • 基础架构:在变分自编码器(VAE)基础上引入三元组对比损失,形成CLVAE。VAE通过编码器将高维分子数据映射到潜在空间,再通过解码器重构原始数据,其目标函数为证据下界(ELBO),包含重构损失和KL散度正则项。
- • 对比损失引入:三元组损失函数通过锚点(FDA批准药物)、正样本(另一FDA批准药物)和负样本(非FDA分子)的设计,促使潜在空间中类药分子聚集、非类药分子分离,损失函数为:
- • 总损失函数:融合重构损失、KL散度和三元组损失,实现潜在空间的结构化构建: 其中,权重系数λ₁、λ₂、λ₃平衡各损失项贡献,最优配置为三元组损失权重0.5,兼顾重构连续性与类药区分性。
4. CLaSP_Score的构建
- • 潜在空间分析:CLVAE构建的潜在空间中,FDA批准药物的分布中心代表理想药物的原型特征,候选分子与该中心的距离反映其与理想药物的相似度,周围药物的局部密度则体现其在已知类药区域的覆盖度。
- • 评分公式:CLaSP_Score通过归一化密度得分(ρₙₒᵣₘ)和归一化距离得分(Dₙₒᵣₘ)的加权求和得到: 其中,α为权重系数(0≤α≤1),ρₙₒᵣₘ基于核密度估计(KDE)计算,Dₙₒᵣₘ为欧氏距离的归一化结果。
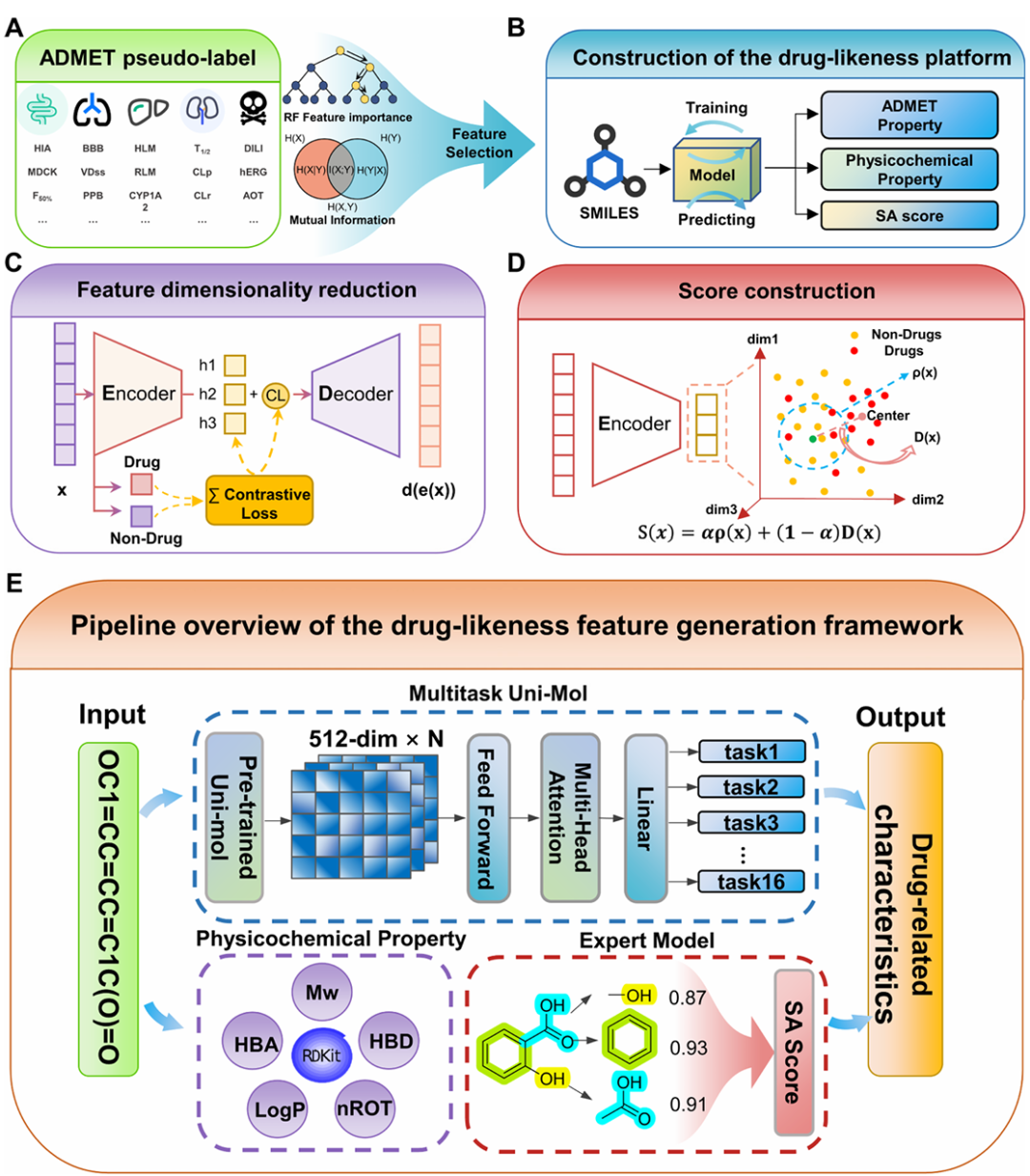
该图展示了 CLaSP 的整体构建流程,包含五个部分。(A)基于随机森林(RF)特征重要性和互信息生成 ADMET 伪标签并进行特征选择;(B)构建多任务预测平台,从 SMILES 表征中预测 ADMET 属性、理化性质和合成可及性(SA)评分;(C)通过基于对比学习的变分自编码器(VAE)提取类药潜空间;(D)基于提取的潜空间构建成药性评分;(E)展示(B)中预测平台的详细结构,整合了基于 Uni-Mol 的多任务 ADMET 预测、RDKit 衍生的理化描述符和基于片段的 SA 评分估计。
该图展示了 CLaSP 的整体构建流程,包含五个部分。(A)基于随机森林(RF)特征重要性和互信息生成 ADMET 伪标签并进行特征选择;(B)构建多任务预测平台,从 SMILES 表征中预测 ADMET 属性、理化性质和合成可及性(SA)评分;(C)通过基于对比学习的变分自编码器(VAE)提取类药潜空间;(D)基于提取的潜空间构建成药性评分;(E)展示(B)中预测平台的详细结构,整合了基于 Uni-Mol 的多任务 ADMET 预测、RDKit 衍生的理化描述符和基于片段的 SA 评分估计。
性能评估与优势分析
1. 潜在空间质量评估
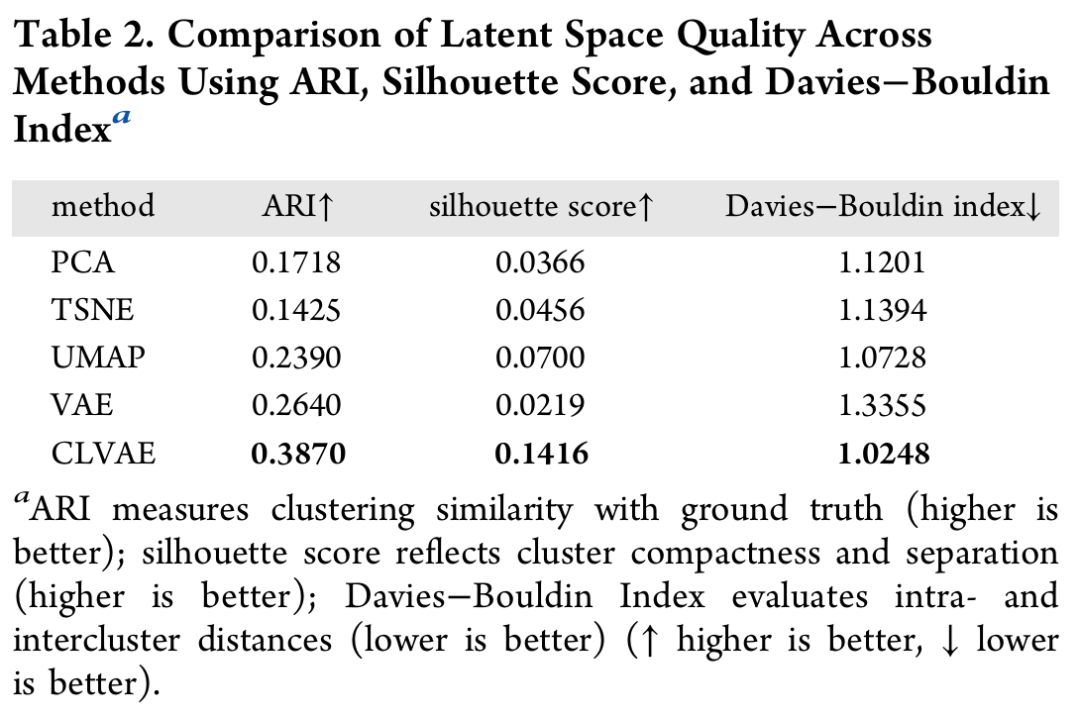
与PCA、t-SNE、UMAP等降维方法相比,CLVAE生成的潜在空间在三个关键指标上表现最优:
- • 调整兰德指数(ARI):0.3870,高于VAE的0.2640,表明与真实标签的聚类一致性更强;
- • 轮廓系数:0.1416,反映聚类紧凑性和分离度更优;
- • 戴维斯-布尔丁指数:1.0248,低于其他方法,说明类内与类间距离平衡更佳。
可视化结果显示,CLVAE能形成从非类药到类药分子的平滑过渡分布,而PCA分离效果差,t-SNE和UMAP仅体现细微理化差异,无法明确区分类药性。
(A)和(B)分别为 CLVAE 和标准 VAE 的潜空间;(C)-(E)为 PCA、t-SNE 和 UMAP 的结果,每个点代表一个化合物,按数据源(FDA、ChEMBL、ZINC、GDB17)着色。CLVAE(A)呈现更结构化且类药感知的分布,形成从非类药到类药化合物的平滑过渡;PCA(C)无法实现有效分离,t-SNE(D)和 UMAP(E)仅部分聚类且与类药性无明确关联,标准 VAE(B)的类别分离弱于 CLVAE。
(A)和(B)分别为 CLVAE 和标准 VAE 的潜空间;(C)-(E)为 PCA、t-SNE 和 UMAP 的结果,每个点代表一个化合物,按数据源(FDA、ChEMBL、ZINC、GDB17)着色。CLVAE(A)呈现更结构化且类药感知的分布,形成从非类药到类药化合物的平滑过渡;PCA(C)无法实现有效分离,t-SNE(D)和 UMAP(E)仅部分聚类且与类药性无明确关联,标准 VAE(B)的类别分离弱于 CLVAE。
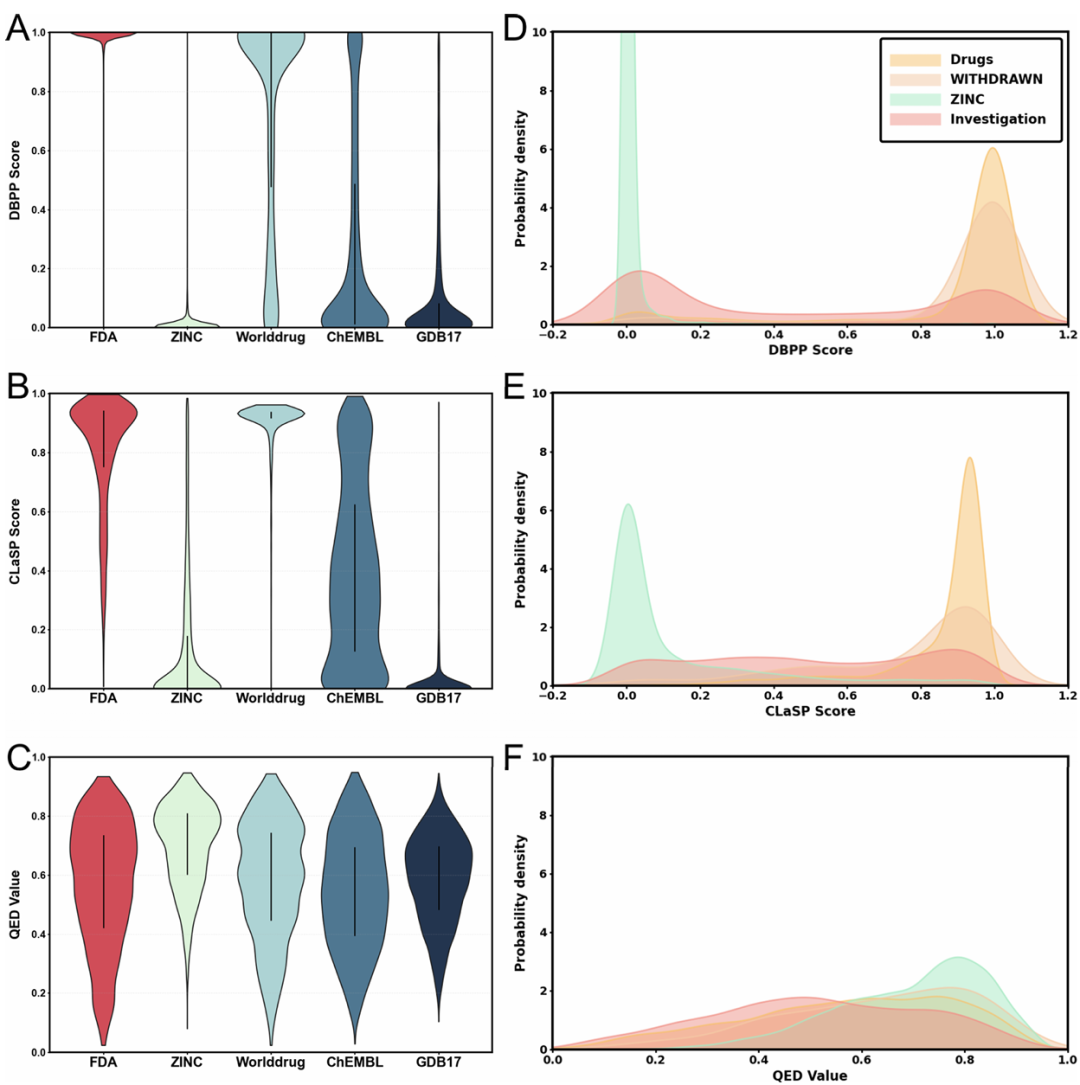
2. 评分性能对比
- • 数据集层面评估:在7个不同来源的数据集上,CLaSP_Score的排名(Drugs > WITHDRAWN > TCMSP > Investigation > ChEMBL > ZINC > GDB17)与药物研发的实际阶段高度吻合。相比之下,QED对药物数据集的评分低于计算机生成的GDB17分子,DBPP_Score则高估撤回药物的评分(甚至高于批准药物)。
- • 真实世界样本测试:对1751个研究阶段化合物和266个撤回药物的分析显示,CLaSP_Score呈现从ZINC到Investigation、Withdrawn再到Drugs的平滑梯度,能有效区分批准药物与撤回药物(CLaSP_Score分别为0.853和0.757),而DBPP_Score难以区分(0.872和0.904)。
- • 样本依赖性分析:Mann-Whitney U检验表明,CLaSP_Score在药物类和非药物类数据集间存在统计显著性差异,即使训练中未涉及非药物分子,仍表现出低样本依赖性,泛化能力优于DBPP_Score(受限于监督训练的样本偏差)和QED(受数据筛选实践影响)。
3. 案例研究:Wee1抑制剂的优化轨迹
在Wee1激酶抑制剂的优化案例中,CLaSP_Score与抑制剂的活性(IC₅₀)呈正相关。例如,化合物34和32的CLaSP_Score较高(0.539、0.526),其WEE1抑制活性也优于母体化合物AZD1775(IC₅₀分别为134.2 nM、14 nM vs 41 nM);而具有血液毒性的ZN-c3(化合物2)评分较低(0.294),无不良事件报告的SC0191(化合物3)评分较高(0.537),表明CLaSP能有效捕捉药物优化过程中的类药性提升。
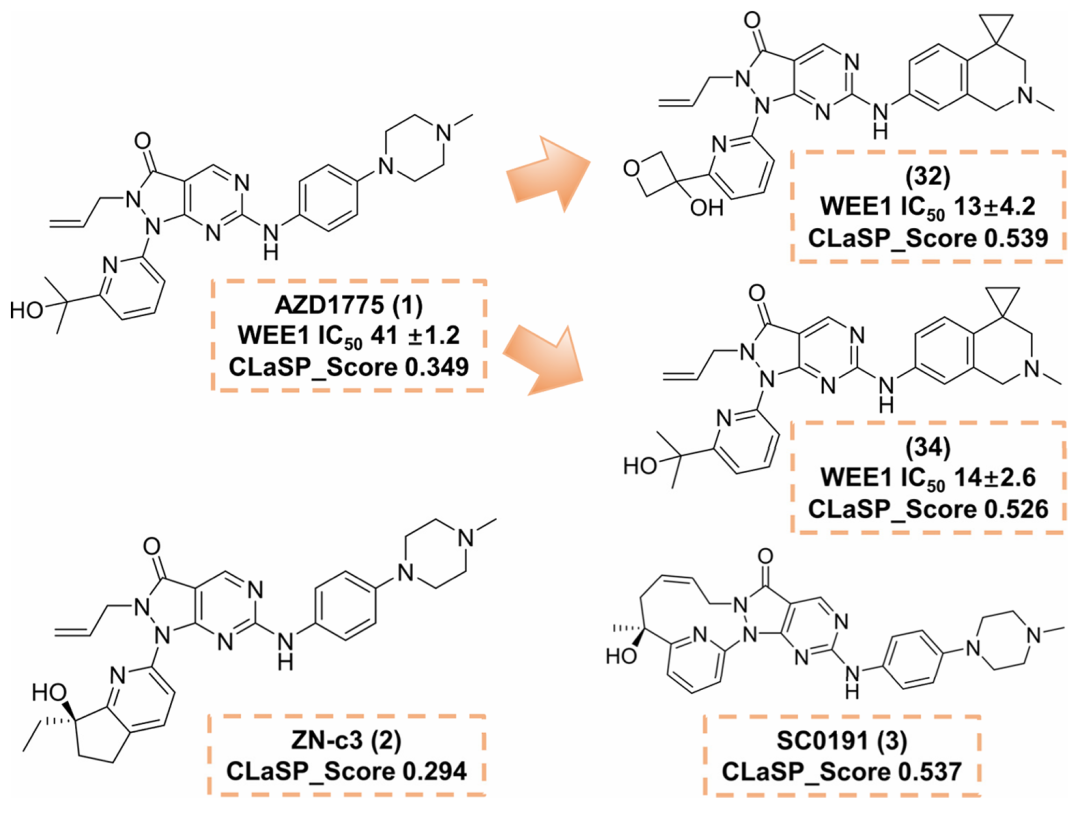
基于 AZD1775(1)的 WEE1 抑制剂结构优化展示,标注了关键化合物的 CLaSP 评分和 WEE1 抑制活性(IC₅₀)。化合物 32 和 34 表现出更高的效力和 CLaSP 评分,SC0191(3)相比 ZN-c3(2)具有更优的特性,与 CLaSP 评分一致。
基于 AZD1775(1)的 WEE1 抑制剂结构优化展示,标注了关键化合物的 CLaSP 评分和 WEE1 抑制活性(IC₅₀)。化合物 32 和 34 表现出更高的效力和 CLaSP 评分,SC0191(3)相比 ZN-c3(2)具有更优的特性,与 CLaSP 评分一致。
应用价值与Web平台
1. 综合优势
- • 可解释性:通过潜在空间的维度分析,明确各维度对应的分子属性(如z₁与分子复杂性和脂溶性相关,z₂与亲水性和蛋白结合特性相关,z₃与安全性和代谢特征相关),结合SHAP分析增强解释性;
- • 适应性:半监督训练策略减少对标注数据的依赖,支持迁移学习,适用于新型化学实体的评估;
- • 全面性:整合ADMET、理化性质和合成可及性,相比QED(仅理化性质)、ADMET-score(仅ADMET)更全面;
- • 实用性:提供代码和Web平台,支持单样本和批量分析,无需本地安装。

2. Web平台功能
CLaSP Web平台(https://lmmd.ecust.edu.cn/CLaSP)设计用户友好,支持:
- • 输入方式:SMILES字符串或CSV文件上传(需包含“SMILES”列);
- • 输出结果:CLaSP_Score及22个类药性相关终点(ADMET、理化性质等);
- • 技术支持:前端基于HTML/CSS/JavaScript,后端采用Django框架,集成PyTorch、RDKit等工具。
局限与展望
CLaSP目前尚未整合明确的结构描述符,在靶点特异性任务(如结合亲和力预测、骨架优化)中存在局限。未来可通过以下方向拓展:
- • 引入结构条件生成模型,增强对分子-靶点相互作用的捕捉;
- • 采用基于图的编码器,提升对分子结构特征的表征能力;
- • 扩展特征集,纳入更多与特定疾病相关的生物活性指标。
结语
CLaSP通过对比学习与变分自编码的创新融合,构建了一个兼具泛化能力和可解释性的类药性评估框架。其在多数据集上的卓越表现、对药物优化轨迹的精准捕捉,以及便捷的Web平台,使其成为早期药物研发中化合物筛选和优先级排序的有力工具。对于 medicinal chemists 和药物研发人员而言,CLaSP不仅提供了量化的类药性评分,更通过潜在空间的结构化分析,为分子优化提供了明确的方向指导,有望加速药物发现进程。
(●'◡'●) 需要进一步讨论的同学欢迎留言交流!
本文参与 腾讯云自媒体同步曝光计划,分享自微信公众号。
原始发表:2025-08-03,如有侵权请联系 cloudcommunity@tencent.com 删除
评论
登录后参与评论
推荐阅读
目录

