国内多所顶尖机构联合研发 AuroBind:突破虚拟筛选瓶颈,解锁海量未被开发蛋白质的成药潜力
国内多所顶尖机构联合研发 AuroBind:突破虚拟筛选瓶颈,解锁海量未被开发蛋白质的成药潜力
用户1151118
发布于 2026-01-08 13:04:45
发布于 2026-01-08 13:04:45
Fitness Aligned Structural Modeling Enables Scalable Virtual Screening with AuroBind
期刊: arxiv 链接: https://www.arxiv.org/abs/2508.02137 代码: https://github.com/GENTEL-lab/AuroBind 简介: 该论文提出了 AuroBind,一种可扩展的虚拟筛选框架,其创新点在于通过适配性对齐的结构建模,结合直接偏好优化、自蒸馏和师生加速策略,实现了配体结合结构和结合适配性的联合预测。该方法基于 AlphaFold 3 架构进行优化,分两阶段训练:先在 PDB 和自蒸馏的蛋白质 - 配体复合物上训练结构预测能力,再在化学基因组数据上微调适配性预测。实验中,AuroBind 在 DAVIS、BindingDB 和 LIT-PCBA 等数据集上表现优于现有模型,对 10 个疾病相关靶点的前瞻性筛选命中率达 7%-69%,顶级化合物 potency 达亚纳摩尔至皮摩尔水平,尤其在孤儿 GPCRs 上成功识别出活性配体。该论文证明了 AuroBind 在结构 - 功能学习和高通量分子筛选中的有效性,为治疗发现提供了强大工具。

研究背景与科学问题
在当代药物研发领域,人类蛋白质组中超过96%的蛋白质仍未被获批治疗药物所靶向,这一“未被药物开发”的巨大空白凸显了传统药物发现方法的局限性。结构基于虚拟筛选技术虽被寄予厚望,但其在原子级精度与结合适配性预测方面的固有缺陷,严重制约了其转化应用价值。现有方法中,物理基于的对接工具如Glide和AutoDock Vina在凸面或高极性结合位点表现不佳,而深度学习模型则在实际虚拟筛选场景中的效能尚未得到充分验证。
正是在这一背景下,郑双佳团队开发了AuroBind——一个兼具可扩展性与高精度的虚拟筛选框架,旨在通过适配性对齐的结构建模,突破现有技术瓶颈,实现对超大化合物库的高效筛选。
AuroBind框架的核心设计
AuroBind的架构设计植根于AlphaFold 3的基础架构,但通过三项关键扩展实现了功能跃升。其核心创新在于将蛋白质序列与配体SMILES作为输入,通过48层PairFormer捕获蛋白质-配体的联合表征,进而通过扩散模块生成原子分辨率的复合物结构,同时配备适配性预测头以输出残基水平的适配性图谱和全局结合适配性分数。这种设计使其能够在单次前向传播中同时预测原子级结合构象与适配性,为后续筛选奠定了基础。
为实现规模化筛选,团队提出了师生蒸馏策略,开发了轻量级学生模型AuroFast。该模型专注于结合适配性预测,较AlphaFold 3实现了100,000倍的推理速度提升,使得在标准GPU集群上每小时筛选2000-3000万个化合物成为可能。这种效率提升并未以精度为代价,通过保留结构嵌入与适配性预测能力,AuroFast在保持筛选质量的同时,彻底改变了大规模虚拟筛选的可行性。
训练过程采用两阶段策略:第一阶段针对蛋白质-配体结构预测,使用约10万个PDB来源的复合物进行初始训练,随后通过自蒸馏策略从BindingDB数据集生成23万个高置信度复合物结构进行优化;第二阶段则在约127万个化学基因组对数据集上进行微调,先采用均方误差目标建立基础适配性预测能力,再通过直接偏好优化(DPO)进一步提升模型区分细微适配性差异的能力。这种训练范式确保了模型在结构准确性与功能适配性之间的精准平衡。
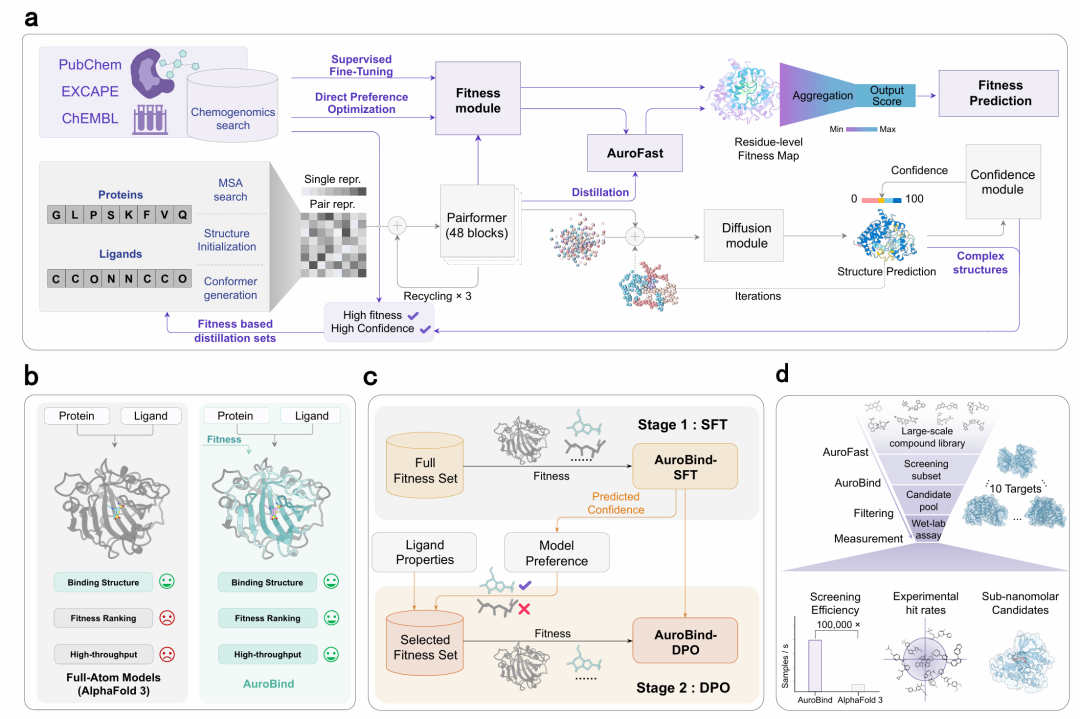
该图展示了 AuroBind 的整体框架,包括模型架构、与 AlphaFold 3 的对比、两阶段训练策略及高通量实验验证流程。模型以蛋白质序列和配体 SMILES 为输入,通过 PairFormer 捕获相互作用并生成复合物结构,同时预测适配性;AuroFast 作为蒸馏出的学生模型实现超快速筛选。训练分为监督微调与直接偏好优化两个阶段,最终通过对 10 个靶点的筛选验证,展现出高命中率、强富集能力和亚纳摩尔级先导化合物的发现能力。
该图展示了 AuroBind 的整体框架,包括模型架构、与 AlphaFold 3 的对比、两阶段训练策略及高通量实验验证流程。模型以蛋白质序列和配体 SMILES 为输入,通过 PairFormer 捕获相互作用并生成复合物结构,同时预测适配性;AuroFast 作为蒸馏出的学生模型实现超快速筛选。训练分为监督微调与直接偏好优化两个阶段,最终通过对 10 个靶点的筛选验证,展现出高命中率、强富集能力和亚纳摩尔级先导化合物的发现能力。
计算性能与基准测试
在结合适配性排序基准测试中,AuroBind表现出显著优势。在DAVIS和BindingDB数据集上,经过精细调优的AuroBind-FT版本的精确率-召回率曲线下面积(AUPR)分别达到0.61和0.70,较最佳基线模型ConPLex分别提升33.8%和11.6%。值得注意的是,即使是零样本变体AuroBind-ZS也优于AlphaFold 3的结构置信度分数作为适配性代理的表现,显示出其在未见过的数据上的强大泛化能力。
在大规模虚拟筛选评估中,AuroFast在LIT-PCBA基准测试中实现了7.58的1%富集因子,较次优方法提升37.6%。这一结果尤为重要,因为该基准包含超过260万个蛋白质-配体复合物,涵盖15个靶点,且测试过程严格排除了训练集中出现的任何复合物,确保了评估的公正性。计算效率方面,AuroFast每化合物平均耗时仅0.0012秒,较AutoDock Vina快25,000倍,为超大规模筛选提供了技术支撑。
结构预测精度验证中,AuroBind在PoseBuster V1和V2基准上的成功率分别达到79.1%和81.7%,超过AlphaFold 3和Protenix等方法,并较传统对接工具提升22%-28%。在7OFF复合物案例中,AuroBind准确恢复了晶体结构中观察到的关键极性相互作用,包括GLU106、ASP141和ARG139之间的氢键网络,而AlphaFold 3的预测则缺失了多个关键相互作用,凸显了AuroBind在捕捉精细结构特征方面的优势。
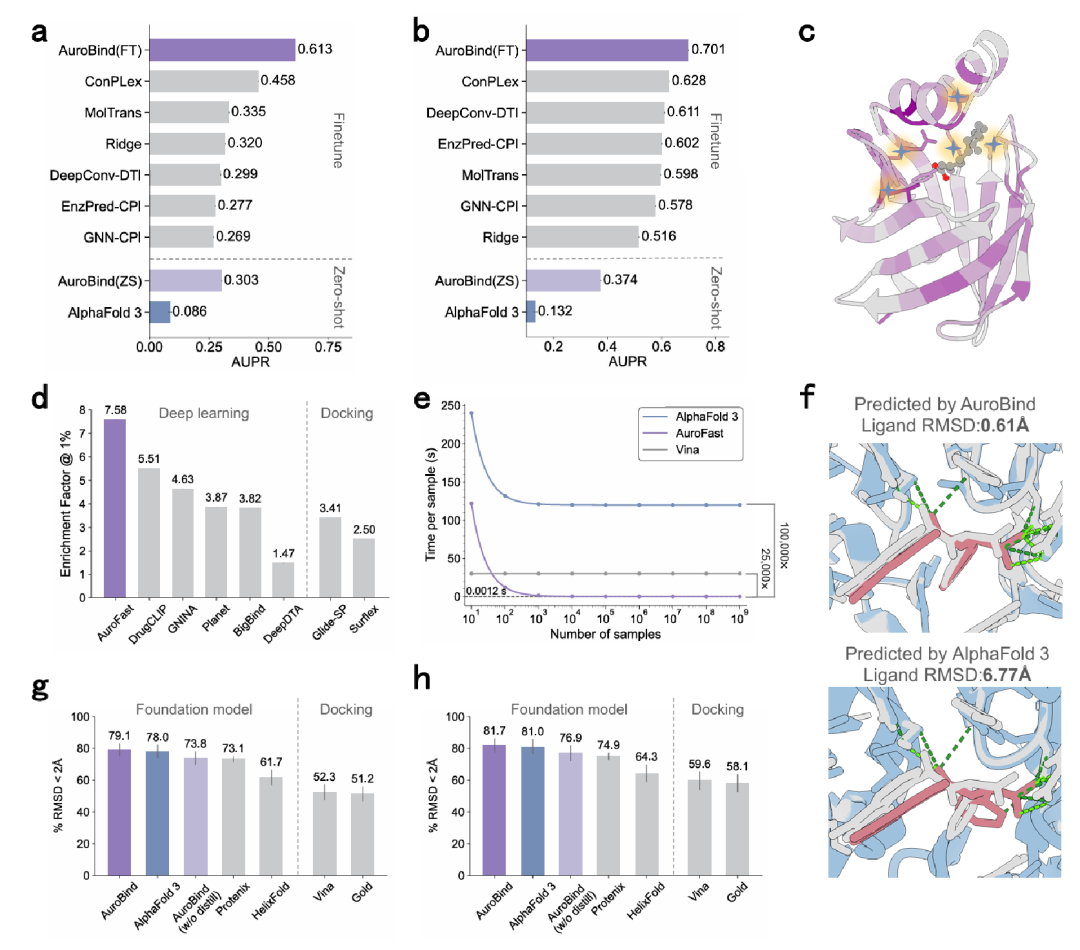
该图呈现了 AuroBind 在多项基准测试中的表现。在 DAVIS 和 BindingDB 数据集上,其精确率 - 召回率曲线下面积(AUPR)优于其他模型;LIT-PCBA 基准中,AuroFast 的 1% 富集因子(EF1%)领先于深度学习和对接方法;计算效率上,AuroFast 较 AlphaFold 3 快 100,000 倍,较 AutoDock Vina 快 25,000 倍。结构预测方面,AuroBind 在 PoseBuster V1 和 V2 基准上的成功率超过 AlphaFold 3 等工具,且在 7OFF 复合物案例中展示出更准确的构象预测。
该图呈现了 AuroBind 在多项基准测试中的表现。在 DAVIS 和 BindingDB 数据集上,其精确率 - 召回率曲线下面积(AUPR)优于其他模型;LIT-PCBA 基准中,AuroFast 的 1% 富集因子(EF1%)领先于深度学习和对接方法;计算效率上,AuroFast 较 AlphaFold 3 快 100,000 倍,较 AutoDock Vina 快 25,000 倍。结构预测方面,AuroBind 在 PoseBuster V1 和 V2 基准上的成功率超过 AlphaFold 3 等工具,且在 7OFF 复合物案例中展示出更准确的构象预测。
实验验证与药物发现应用
为验证AuroBind的实际应用价值,研究团队针对10个治疗相关靶点开展了系统性虚拟筛选 campaign,涵盖受体酪氨酸激酶、表观遗传调控因子和G蛋白偶联受体(GPCR)等多个家族。筛选流程采用分层策略:先用AuroFast从3000万个可购买化合物中筛选出10,000个候选,再经AuroBind重新评分得到500个化合物,最终经药物 likeness 过滤后选择30-50个进行实验验证。
实验结果显示,AuroBind的命中率范围为7%-69%,其中8个靶点的命中率超过10%,5个超过20%,4个超过30%。尤为值得关注的是,在孤儿GPCRs(GPR151和GPR160)上,尽管缺乏已知活性化合物或晶体结构,且与训练集中蛋白质的序列一致性低于30%,AuroBind仍成功识别出激动剂和拮抗剂,成功率达16%-30%。功能测定证实,这些化合物能在肝癌和前列腺癌模型中调控GPR160的活性,展示了AuroBind在探索未知靶点方面的独特价值。
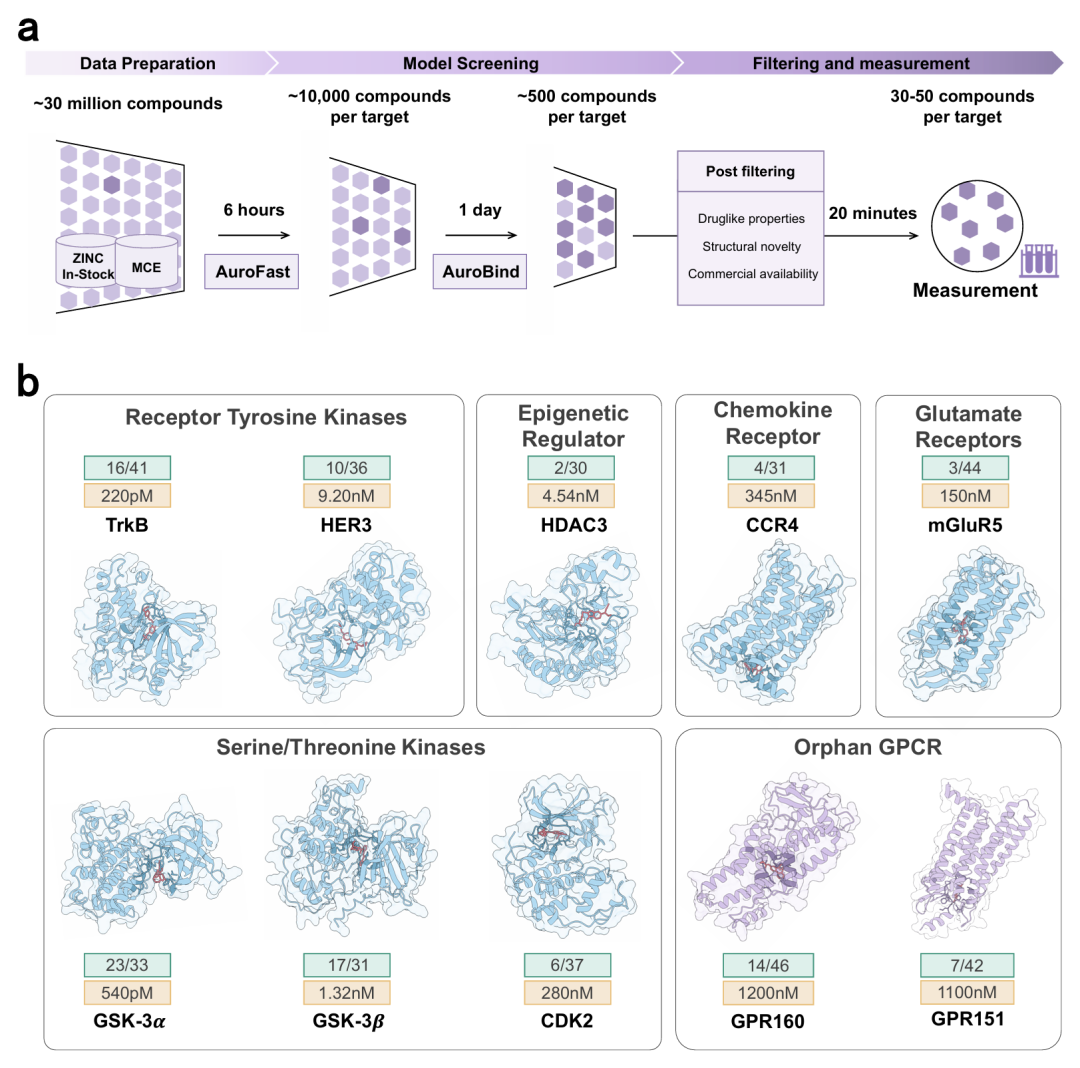
该图展示了虚拟筛选和验证工作流程,以及 10 个不同蛋白质靶点的实验结果。筛选从约 3000 万个化合物开始,经 AuroFast 和 AuroBind 逐步筛选至 30-50 个化合物进行湿实验验证。实验结果显示,各靶点的命中率在 7%-69% 之间,顶级化合物的结合亲和力(IC₅₀)从皮摩尔到纳摩尔不等,涵盖受体酪氨酸激酶、表观遗传调控因子、GPCR 等多个家族,验证了 AuroBind 的广泛适用性。
该图展示了虚拟筛选和验证工作流程,以及 10 个不同蛋白质靶点的实验结果。筛选从约 3000 万个化合物开始,经 AuroFast 和 AuroBind 逐步筛选至 30-50 个化合物进行湿实验验证。实验结果显示,各靶点的命中率在 7%-69% 之间,顶级化合物的结合亲和力(IC₅₀)从皮摩尔到纳摩尔不等,涵盖受体酪氨酸激酶、表观遗传调控因子、GPCR 等多个家族,验证了 AuroBind 的广泛适用性。
在化合物 potency 方面,AuroBind筛选出的顶级化合物达到亚纳摩尔至皮摩尔水平。其中TrkB靶向化合物的IC50低至220 pM,GSK3α的最佳化合物IC50为540 pM。与先前报道的筛选方法相比,AuroBind在多个靶点上的效能提升显著:mGluR5(15.3倍)、HER3(435倍)、TrkB(818倍)和GSK3α(4185倍),即使与经湿实验优化的化合物相比仍具优势。
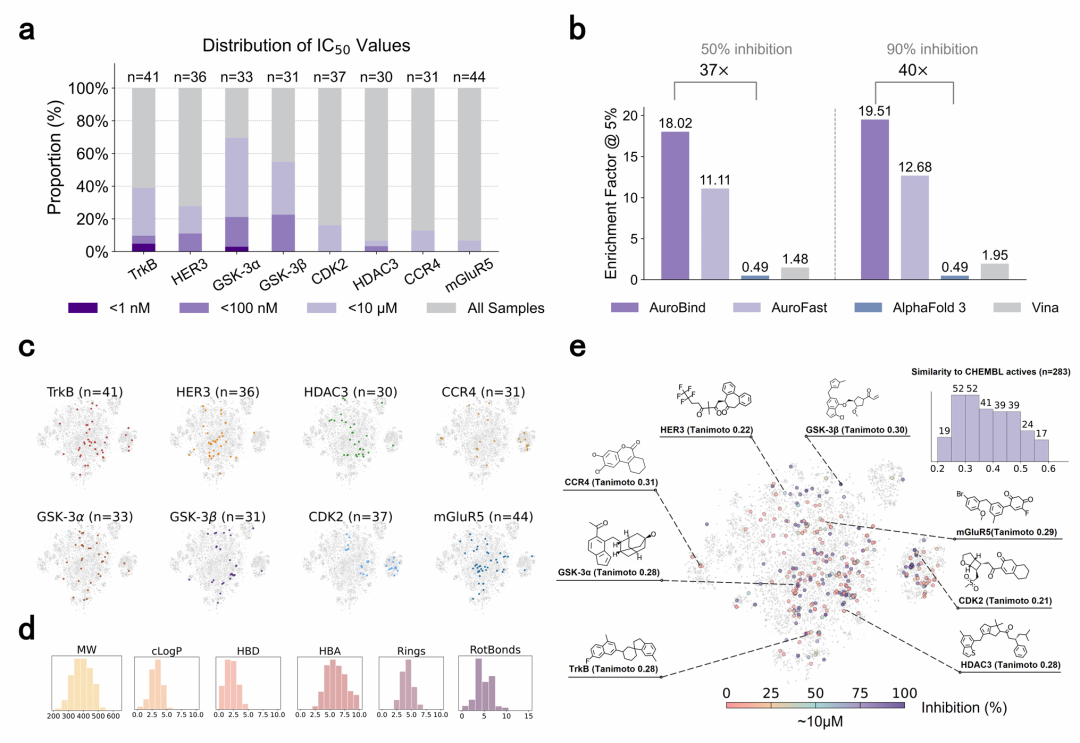
该图分析了 AuroBind 筛选化合物的效能、富集能力、化学多样性和新颖性。效能分布显示多个靶点的化合物达到亚纳摩尔级;富集因子(EF1%)表明 AuroBind 在筛选高活性化合物方面优于其他方法;t-SNE 图和理化性质分析显示命中化合物覆盖广泛的化学空间;结构新颖性分析发现 25% 的命中化合物与已知活性分子相似度低,证明其发现新骨架的能力。
该图分析了 AuroBind 筛选化合物的效能、富集能力、化学多样性和新颖性。效能分布显示多个靶点的化合物达到亚纳摩尔级;富集因子(EF1%)表明 AuroBind 在筛选高活性化合物方面优于其他方法;t-SNE 图和理化性质分析显示命中化合物覆盖广泛的化学空间;结构新颖性分析发现 25% 的命中化合物与已知活性分子相似度低,证明其发现新骨架的能力。
化学多样性分析显示,25%的命中化合物与ChEMBL中已知活性分子的Tanimoto相似度低于0.3,表明AuroBind能够发现超越传统方法范围的结构多样分子。这种能力在孤儿GPCRs上表现得尤为突出,成功识别出此前无任何活性小分子报道的靶点的有效配体,证明了其在开拓全新化学空间方面的潜力。
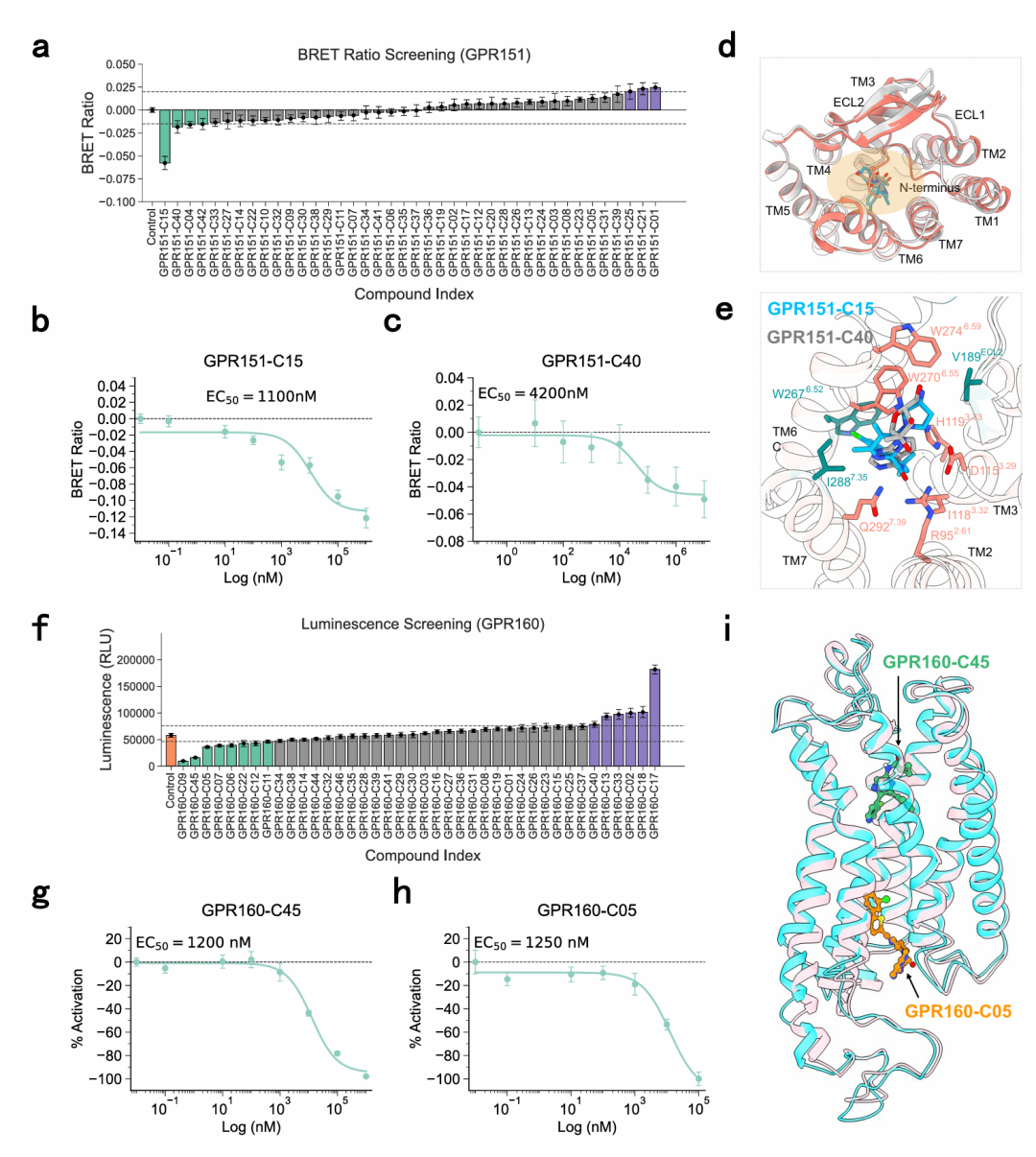
该图展示了针对孤儿 GPCR(GPR151 和 GPR160)的功能验证结果。GPR151 的 BRET2 测定发现 7 个活性化合物,其中 GPR151-C15 和 GPR160-C40 的 EC₅₀分别为 1100 nM 和 4200 nM,结构建模显示它们与受体的正交口袋结合并形成关键相互作用。GPR160 的 GloSensor cAMP 测定发现 14 个活性化合物,GPR160-C45 和 GPR160-C05 的 EC₅₀约为 1200 nM 和 1250 nM,且结合模式不同,证明 AuroBind 对未表征靶点的筛选能力。
该图展示了针对孤儿 GPCR(GPR151 和 GPR160)的功能验证结果。GPR151 的 BRET2 测定发现 7 个活性化合物,其中 GPR151-C15 和 GPR160-C40 的 EC₅₀分别为 1100 nM 和 4200 nM,结构建模显示它们与受体的正交口袋结合并形成关键相互作用。GPR160 的 GloSensor cAMP 测定发现 14 个活性化合物,GPR160-C45 和 GPR160-C05 的 EC₅₀约为 1200 nM 和 1250 nM,且结合模式不同,证明 AuroBind 对未表征靶点的筛选能力。
讨论与展望
AuroBind的成功在于其从单纯几何相互作用建模向功能感知结构学习的范式转变,通过整合结构表征学习与化学基因组适配性数据,实现了从构象预测到结构-功能关系捕获的跨越。这种转变使其能够在缺乏实验解析复合物结构的情况下依然保持高性能,如在GPR160这类构象灵活的孤儿GPCR上的成功应用所示。
与同期发表的Boltz-2等方法相比,AuroBind的独特优势体现在三个方面:支持超高通量虚拟筛选的规模化能力、从计算预测到前瞻性实验验证的闭环体系,以及针对未知结构靶点的发现能力。这些优势使其不仅在性能指标上领先,更在实际药物研发流程中展现出实用价值。
当然,AuroBind仍存在局限性:对高度动态或无序靶点(如KRAS或c-Myc)的性能有待评估,在数据稀疏场景中适配性分数的准确性仍有提升空间,针对极度稀疏蛋白质家族的泛化策略需要进一步探索。这些方向将成为未来研究的重点。
总体而言,AuroBind展示了功能微调的结构基础模型在连接几何结构与生物学功能方面的巨大潜力。随着生成建模技术和结构数据库的持续发展,这类框架有望成为自主化、高通量、功能导向的药物发现平台的基础,推动药物研发迈向AI驱动的端到端设计新时代。
本文参与 腾讯云自媒体同步曝光计划,分享自微信公众号。
原始发表:2025-08-06,如有侵权请联系 cloudcommunity@tencent.com 删除
评论
登录后参与评论
推荐阅读
目录

