日本筑波大学打造Boltz-sample:通过对表征缩放来引导 Boltz-2 进行蛋白多构象采样
日本筑波大学打造Boltz-sample:通过对表征缩放来引导 Boltz-2 进行蛋白多构象采样

DrugIntel
发布于 2026-01-27 14:44:08
发布于 2026-01-27 14:44:08

在结构生物学领域,深度学习驱动的蛋白质结构预测技术已实现革命性突破,以AlphaFold系列、Boltz系列为代表的模型将预测精度推向接近实验水平。然而,生物体内的蛋白质并非静态单一结构,而是通过在多个功能构象间动态转换实现生理功能——从膜转运蛋白的内外向构象切换,到酶的激活/抑制状态转变,再到折叠转换蛋白的拓扑结构重排,构象多样性是蛋白质功能实现的核心基础。
当前主流预测模型多输出单一优势构象,难以捕捉这种动态多样性。传统解决方案如分子动力学模拟、MSA(多序列比对)扰动等,或面临计算成本高昂的问题,或依赖经验性参数调整,缺乏系统性和可解释性。近日,日本筑波大学Suzuki和Amagasa在bioRxiv发表的研究,提出了一种名为Boltz-sample的创新性构象采样策略,通过对Boltz-2模型隐空间对表征(latent pair representation)的系统性缩放,实现了高效、可控的多构象预测,为解决这一核心难题提供了全新思路。
一、技术核心:隐空间对表征缩放的创新设计
Boltz-sample的核心创新在于避开传统的输入扰动思路,直接对模型内部的潜空间表征进行调控,其技术设计兼具简洁性与深刻的生物学机制关联性。
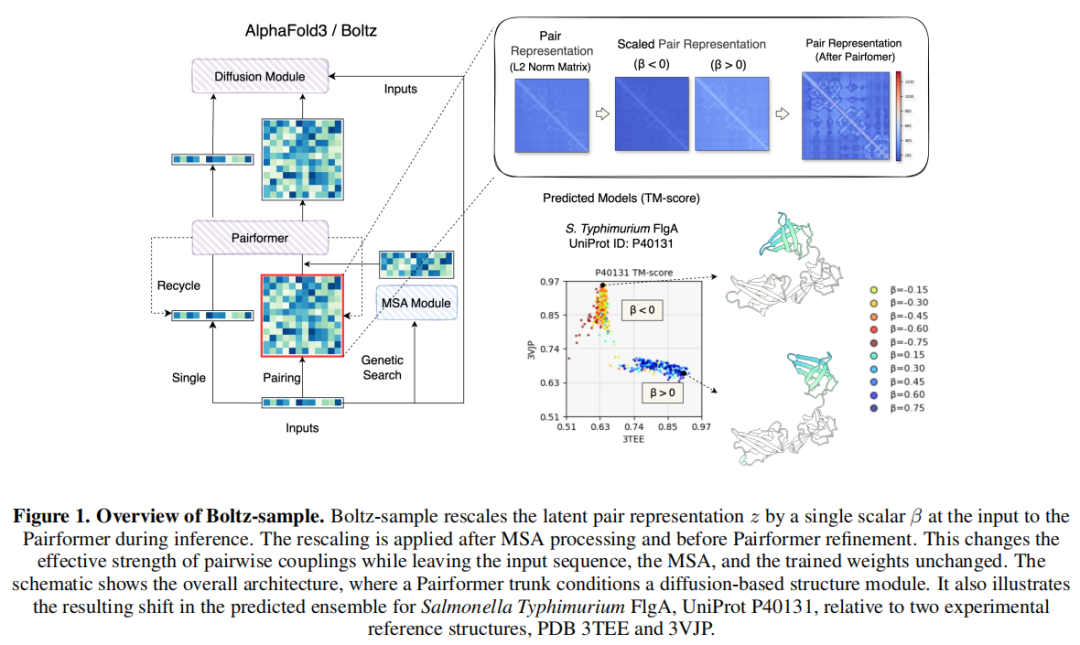
1. 调控对象与数学表达
Boltz-2模型采用AlphaFold3风格的架构设计,通过Pairformer模块整合序列特征与共进化信息,生成隐空间对表征张量(N为残基数,为表征维度),该张量本质上是介导进化约束向结构几何转化的残基-残基有效耦合场。
Boltz-sample在推理阶段的每个循环步骤中,对Pairformer输入的隐空间对表征施加全局标量缩放:
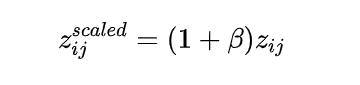
其中为核心调控参数,取值范围为(-1, 1)。这一操作不改变输入序列、MSA数据及模型训练权重,仅通过调整对耦合的有效强度,系统性调控扩散过程的信号噪声比。
2. 调控机制的物理意义
- 当时:增强共进化约束与几何约束的权重,使模型更聚焦于优势构象,提升预测精度;
- 当时:放松上述约束,降低优势构象的信号压制,引导模型探索潜在的替代构象;
- 与无分类器引导(classifier-free guidance)不同,该策略无需额外分类器或无条件分支,避免了二次前向传播的计算开销,同时保持了调控的透明性与可解释性。
3. 与传统方法的本质区别
传统MSA扰动方法(子采样、随机掩码、聚类等)通过修改输入进化信息诱导构象多样性,需依赖经验性参数选择(如子采样比例、聚类数量),且结果具有不确定性。而Boltz-sample直接作用于模型内部整合后的结构信息表征,通过单一参数实现对构象采样方向的系统性调控,将构象探索从 输入扰动的随机搜索 转变为 隐空间的定向导航 。
二、实证性能:多维度基准测试
研究团队在三类具有代表性的基准数据集上,从构象覆盖度、预测精度、计算效率等维度,对Boltz-sample进行了全面验证,其性能表现显著优于传统方法。
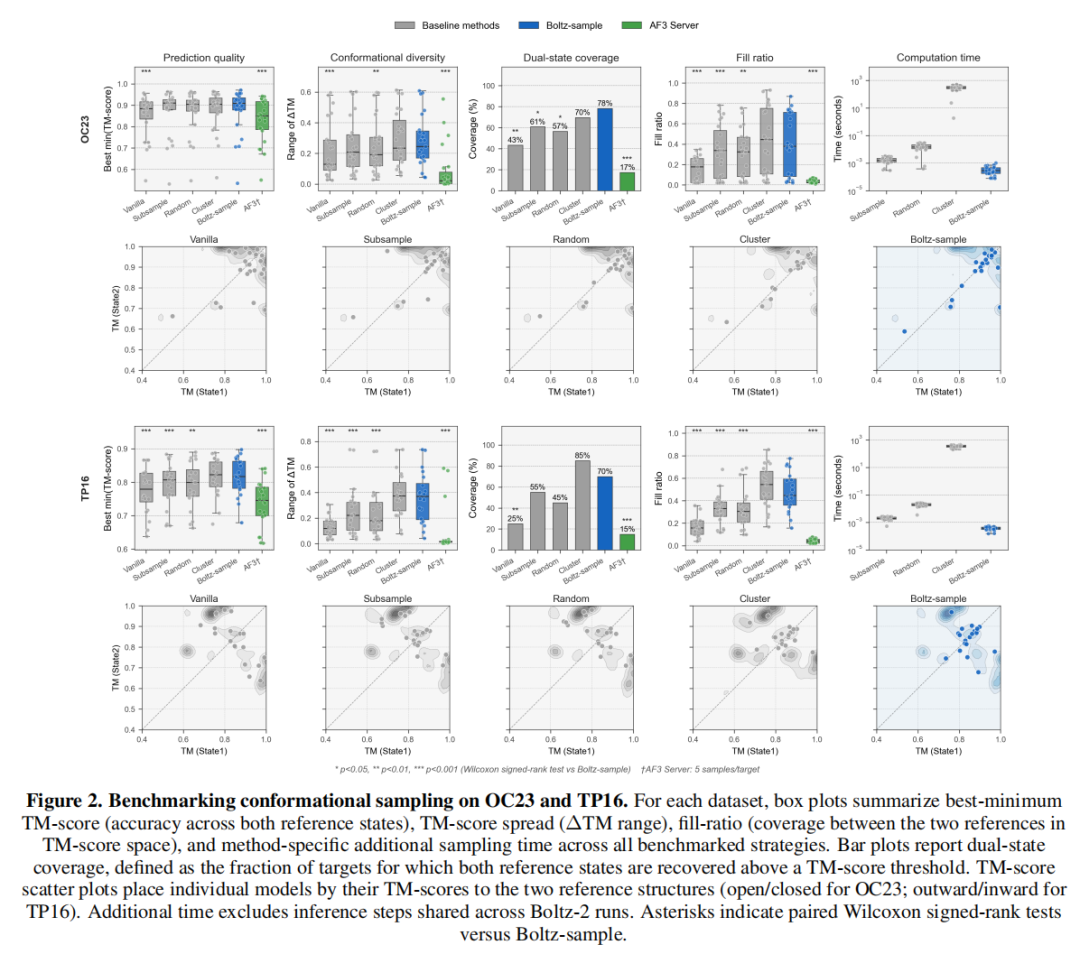
1. 标准构象转换数据集(OC23+TP16)
OC23包含23个具有开放/闭合构象的蛋白质(成对TM-score ≤ 0.85),TP16则聚焦膜转运蛋白的向内/向外构象(排除超长序列SPF1后共20个靶点),两类数据集均来自AFsample2基准,是评估构象采样能力的经典测试集。
- 双态覆盖率大幅提升:OC23数据集上,Boltz-sample将双态覆盖率(两构象TM-score均≥0.8的靶点比例)从常规推理的43%提升至78%,超越MSA聚类的70%;TP16数据集上从25%提升至70%,接近聚类方法的85%;
- 构象过渡空间覆盖更完整:fill-ratio指标(量化构象过渡路径覆盖度,对两端状态赋予更高权重)显著优于常规推理及MSA子采样、随机掩码方法,表明其不仅能捕捉端点构象,还能覆盖中间过渡态;
- 计算效率极致优化:每个预测的额外耗时不足秒,远低于MSA聚类(需处理多个聚类子集的编码与推理),实现了高精度与高效率的统一。
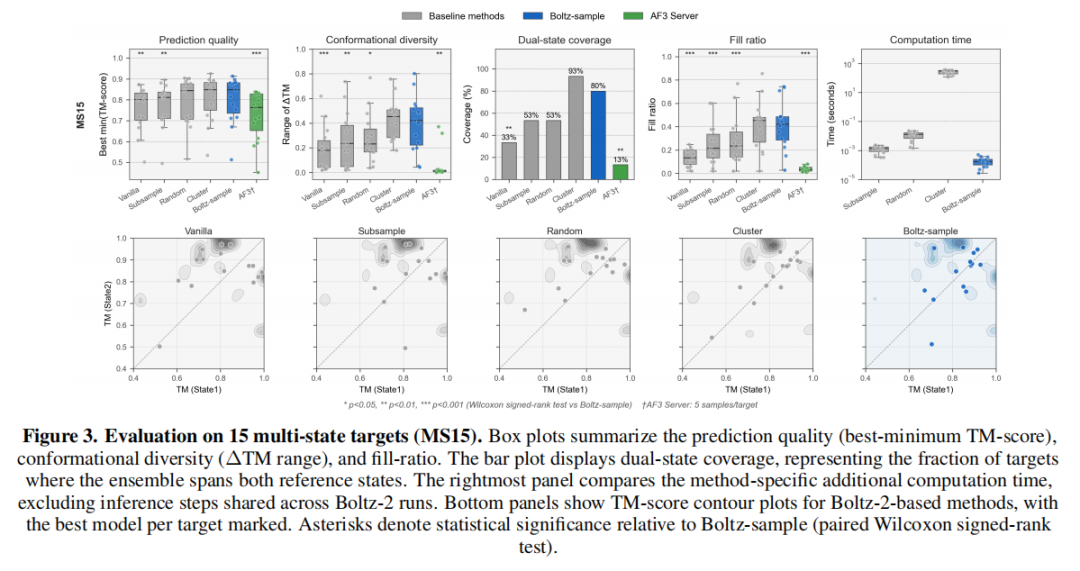
2. 多样化多态靶点数据集(MS15)
为验证方法的普适性,研究团队构建了MS15数据集,包含15个跨6大功能类别的蛋白质(激酶、GTP酶、折叠转换蛋白、膜转运蛋白等),涵盖铰链式结构闭合、电梯式转运、拓扑折叠转换等多种构象变化类型,构象多样性更丰富。
- 双态覆盖率达80%:较常规推理的33%实现质的飞跃,仅略低于MSA聚类的93%,但无需承担聚类带来的高额计算成本;
- 构象预测精度提升:最佳最小TM-score(两构象中较差者的最高TM-score)分布显著向高值偏移(p<0.01),表明其在探索多样性的同时未牺牲预测准确性;
- 适配多种构象变化类型:对腺苷酸激酶(P69441)的连续构象过渡、EF-Tu(P0CE48)的离散模式切换、RfaH(P0AFW0)的α-螺旋向β-桶折叠转换均能有效捕捉,验证了方法对不同构象变化机制的适配性。
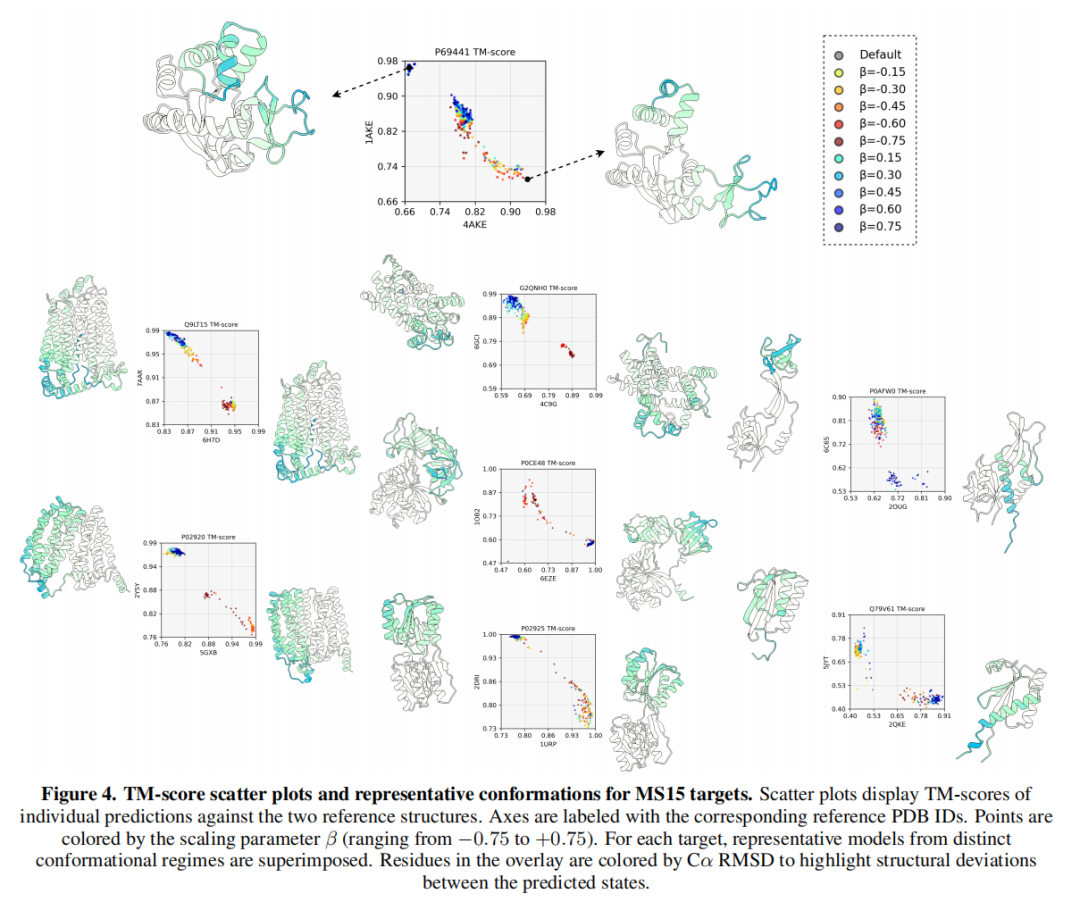
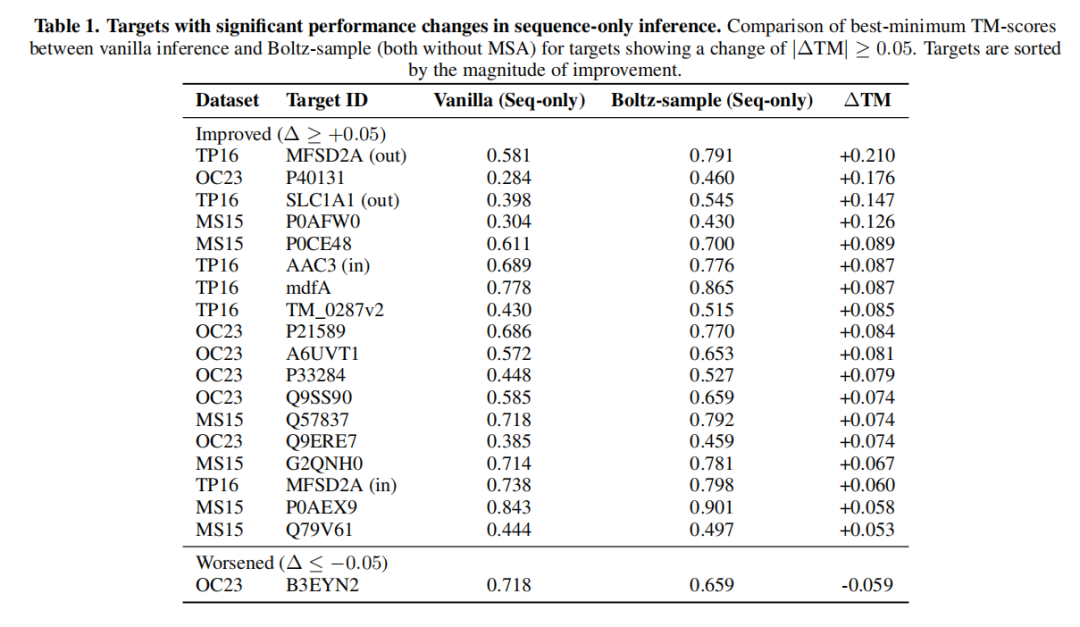
3. 序列仅输入场景的性能验证
关键发现在于,Boltz-sample的优势并非依赖MSA提供的共进化信号。在无MSA输入的序列仅推理场景下:
- 31%的靶点(18个)最佳最小TM-score提升≥0.05,仅1.7%(1个)出现下降;
- 代表性靶点性能提升显著:MFSD2A(TP16)的TM-score从0.58提升至0.79(+0.21),P40131(OC23)从0.28提升至0.46(+0.18);
- 这一结果证明,Boltz-sample能够激活模型从PDB训练集中内化的结构先验知识,即使缺乏明确的共进化信号,仍能高效挖掘序列本身的多构象潜力。
三、机制解析:隐空间调控的深层生物学意义
通过系列消融实验与机制探究,研究团队揭示了Boltz-sample实现高效构象采样的核心机制,为理解深度学习模型的构象表征提供了重要 insights。
1. 结构先验的激活机制
Boltz-2等先进模型在训练过程中,不仅学习了序列与结构的映射关系,还内化了丰富的蛋白质结构先验(如折叠模式、结构域相互作用规则等)。在常规推理中,这些先验被优势构象的强信号压制,而 缩放通过调整约束强度,起到了 解锁 这些潜在先验的作用,使模型能够访问被优势构象掩盖的替代状态。
这一机制挑战了 替代构象仅由共进化信号编码 的传统认知,表明深度学习模型具备捕捉序列内在结构可塑性的能力,而Boltz-sample提供了激活这种能力的有效工具。
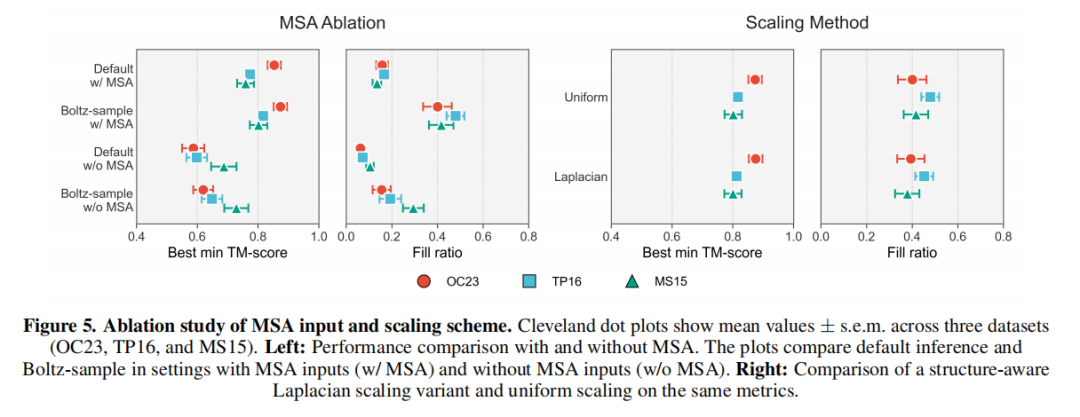
2. 全局缩放vs空间结构化缩放
研究团队对比了全局均匀缩放与基于图拉普拉斯的空间结构化缩放(根据残基相互作用网络的连通性赋予位置依赖权重):
- 两类方法在最佳最小TM-score、fill-ratio等核心指标上无显著差异;
- 这表明,调控构象转换的核心是对表征的全局强度,而非局部空间结构的精细调控,进一步验证了全局标量缩放的合理性与高效性。
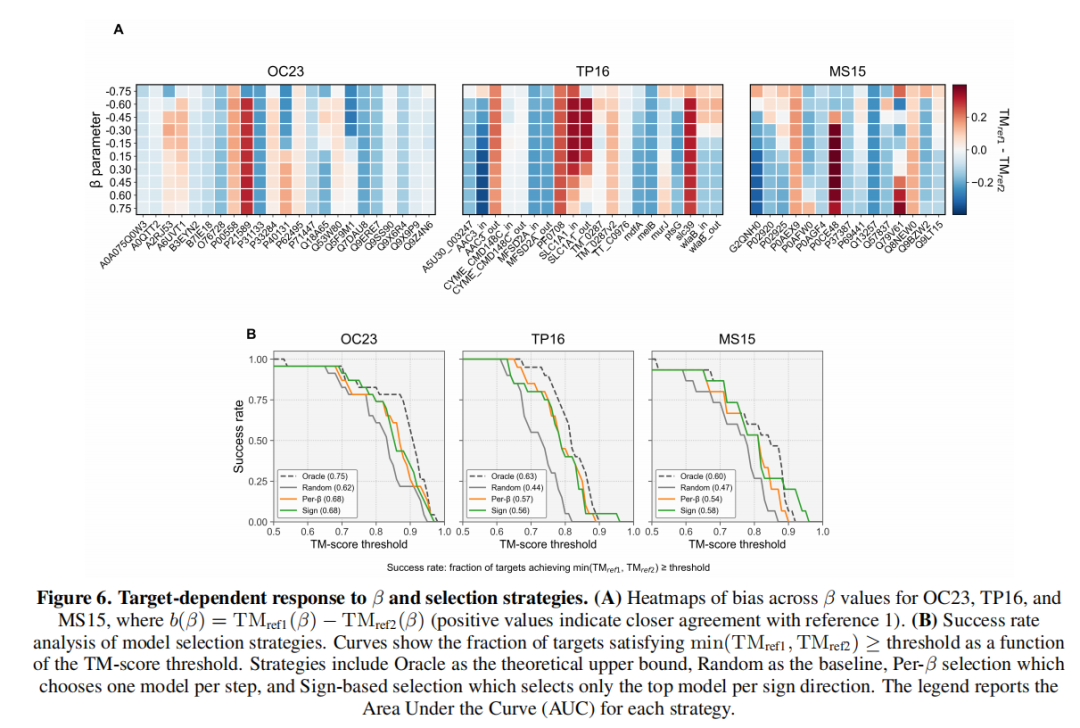
3. 参数响应特性与实用筛选策略
- 靶点依赖性响应: 对构象的调控方向具有靶点依赖性,不同蛋白质对 的响应模式不同(连续过渡或离散切换),但 的符号始终定义了不同的潜空间搜索方向;
- 高效筛选策略:针对无参考结构的实际应用场景,提出两种基于置信度(pLDDT分数)的筛选策略:
- 逐β选择:保留每个β值对应的最高置信度模型(约10个模型);
- 符号选择:仅保留 <0和 >0集合中最高置信度的2个模型;
- 符号选择策略在MS15上的AUC达0.58,接近理论上限(0.60),显著优于随机基线(0.47),无需PCA或聚类等复杂后处理,即可高效获取高质量多构象集合。
4. 方法局限性
Boltz-sample在MAD2(Q13257)等靶点上未能成功恢复替代构象,这一局限性源于其全局调控机制:当替代构象的信号被优势构象强烈压制(即 minority signal被平均化)时,全局缩放无法有效分离弱信号,而MSA聚类等方法通过明确拆分多模态共进化信号,可能在这类靶点上表现更优。
四、小结
Boltz-sample通过对Boltz-2模型隐空间对表征的全局标量缩放,实现了蛋白质构象采样的系统性调控。其证实,先进深度学习模型的隐空间对表征中蕴含着丰富的构象多样性信息,这些信息可通过简单的标量调控被激活。这一发现为理解模型如何编码蛋白质动态特性提供了关键线索,推动了蛋白动态构象探索的理论认知升级。
参考文献:Shosuke Suzuki, Toshiyuki Amagasa, Steering Conformational Sampling in Boltz-2 via Pair Representation Scaling, bioRxiv 2026.01.23.701250
本文参与 腾讯云自媒体同步曝光计划,分享自微信公众号。
原始发表:2026-01-26,如有侵权请联系 cloudcommunity@tencent.com 删除
评论
登录后参与评论
推荐阅读
目录

