初探单细胞下游
我以后会在本专辑每期学习推文开头放上第一期提出的学习希冀:
这个新专辑有以下几点希冀:
- 带着像我一样的单细胞小白,一步步利用我们生信技能树、生信菜鸟团、单细胞天地的资源,掌握基本的scRNAseq流程
- 在学习的过程中,探索出合适的学习路径,帮助大家更好地利用已有资源
- 对过往推文中出现的错误、更新的软件进行审查,推陈出新
- 在过去的基本内容上深入挖掘影响小白学习的障碍,提炼总结,拓宽深度宽度
- 和大家讨论我在从零开始学习过程中遇到的问题,老师们在评论区指出我的不足提出建议
而我在将自己的学习笔记排版成推文时也会遵循以下行文特点:
- 务必详实逐步复现,如展示原推文中没展示的过程结果,添加参考资料帮助理解
- 重点推陈出新,如果原推文足够详细且我没遇到其他问题,可能会直接带过这篇学习推文,只在推文中展示结果,但是仍会告诉大家我看了啥,以便梳理小白学习路径
提醒自己整理笔记推陈出新的同时,告诉中途了解到的老师同学这个专辑的学习性质,避免水文之嫌
Seurat - Guided Clustering Tutorial
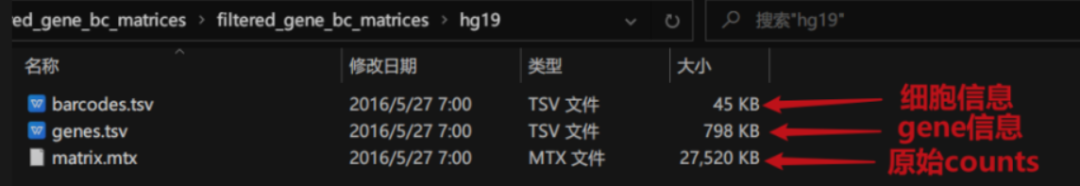
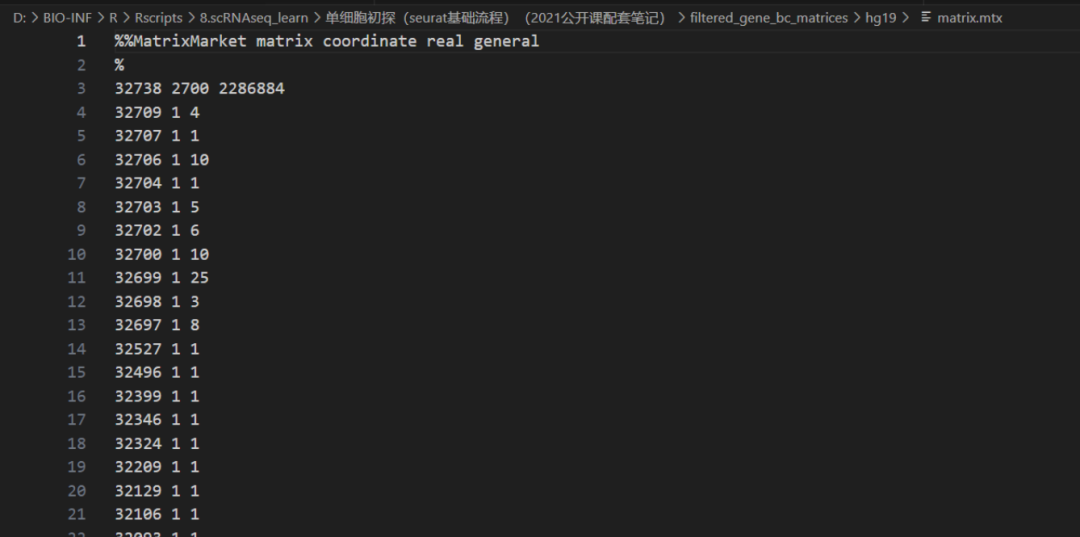
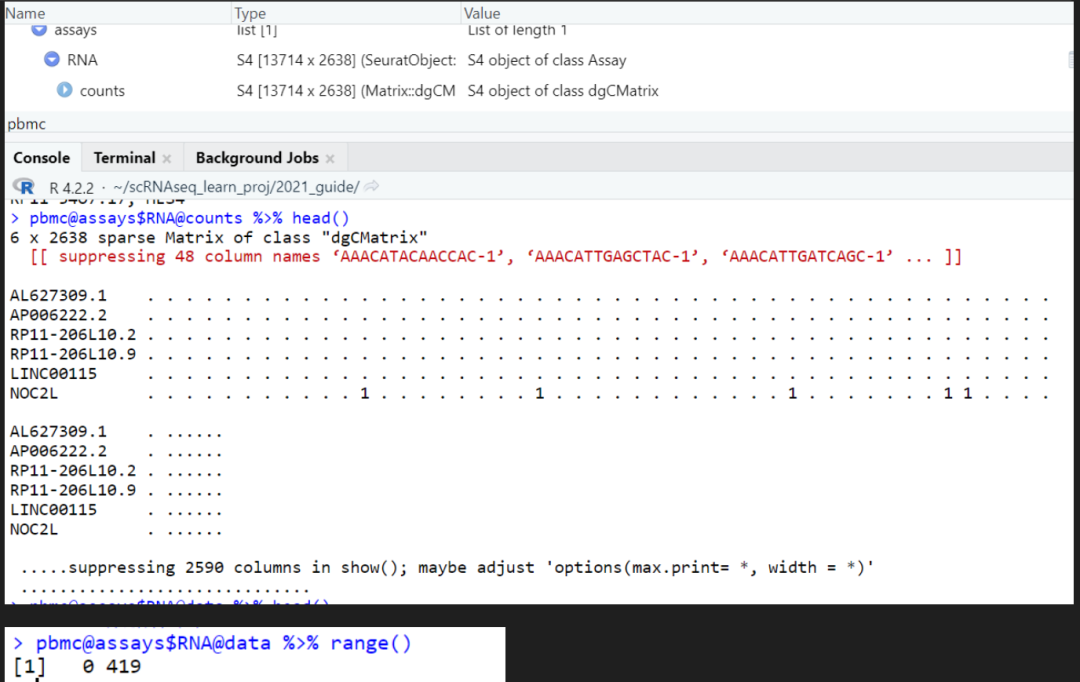
看看数据
原始counts
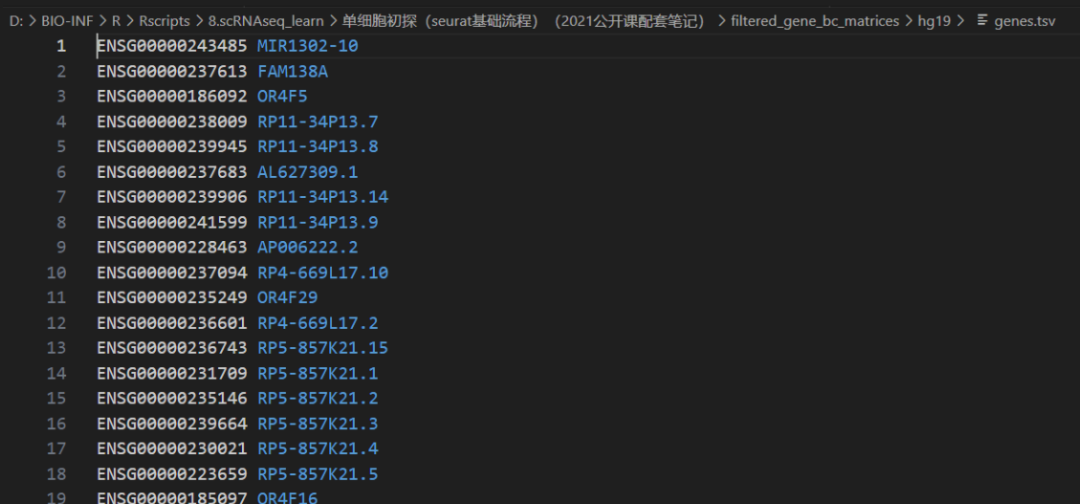
gene信息
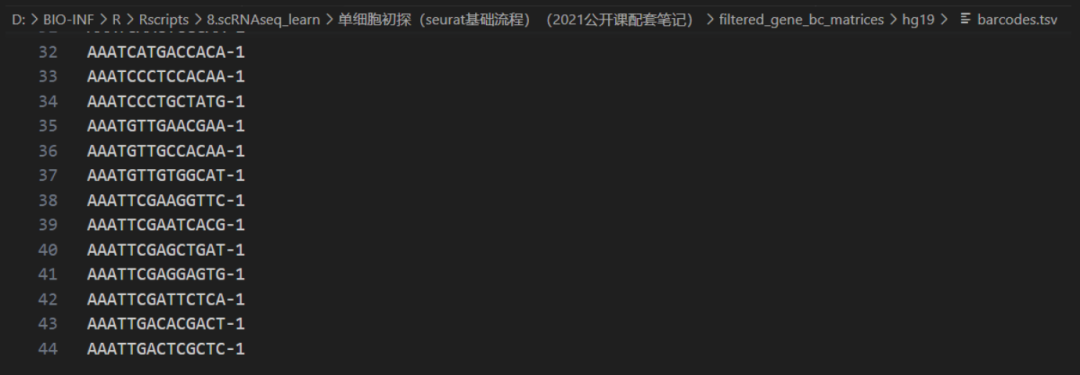
细胞信息barcode
可以记住这个10X技术输出文件目录和格式,以后使用
Read10X函数读取Seurat对象时可以检查一下
rm(list=ls())
## 加载R包
library(dplyr)
library(Seurat)
library(patchwork)
## 读取数据
pbmc.data <- Read10X(data.dir = "data/pbmc3k_filtered_gene_bc_matrices/filtered_gene_bc_matrices/hg19")
## 创建Seruat对象
pbmc <- CreateSeuratObject(counts = pbmc.data,
project = "pbmc3k",
min.cells = 3, # min.cell:每个feature至少在多少个细胞中表达(feature=gene)
min.features = 200) # min.features:每个细胞中至少有多少个feature被检测到
project = "pbmc3k"参数用于指定创建的Seurat对象的项目名称
当前激活ident:

这个因子变量需要注意,一开始创建的时候和orig.ident 因子水平一致,在后面我们会频繁修改这个变量指向的不同分组水平,在不同维度分组上探索数据
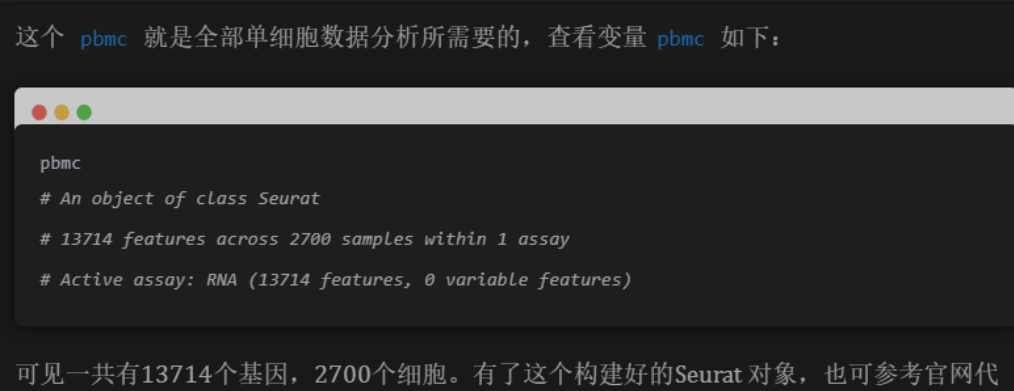
数据质控

## 计算细胞中线粒体基因比例
pbmc[["percent.mt"]] <- PercentageFeatureSet(pbmc, pattern = "^MT-")
## 使用小提琴图可视化QC指标
VlnPlot(pbmc, features = c("nFeature_RNA", "nCount_RNA", "percent.mt"), ncol = 3)
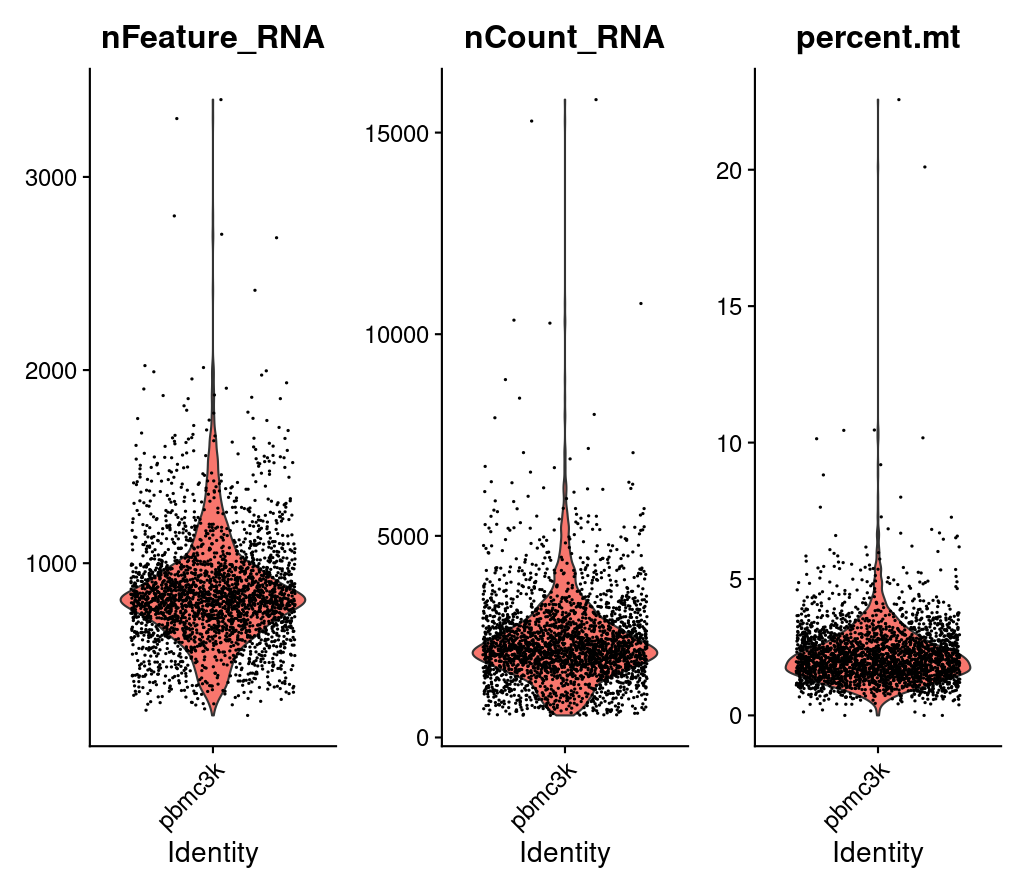
这些数据其实在metadata里
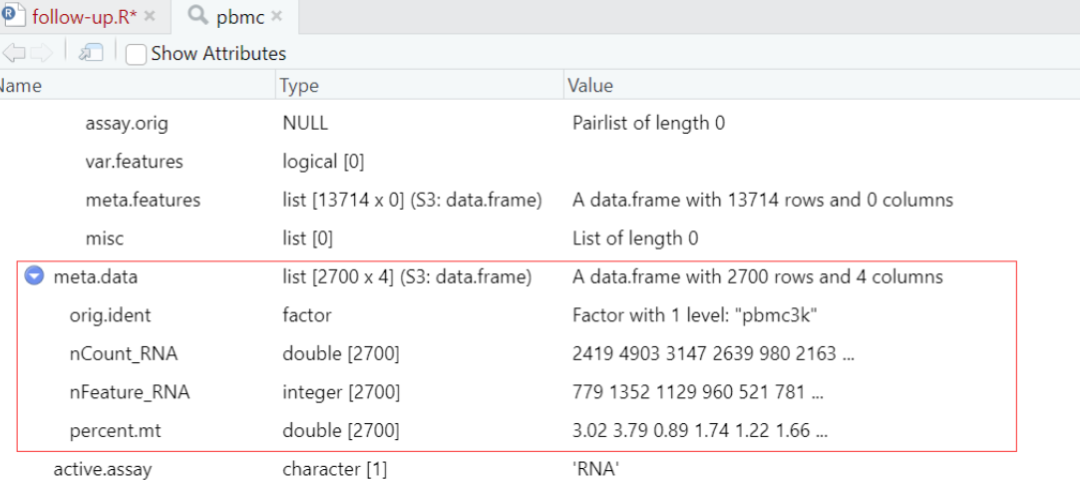
## FeatureScatter于可视化特征-特征关系
plot1 <- FeatureScatter(pbmc, feature1 = "nCount_RNA", feature2 = "percent.mt")
plot2 <- FeatureScatter(pbmc, feature1 = "nCount_RNA", feature2 = "nFeature_RNA")
plot1 + plot2
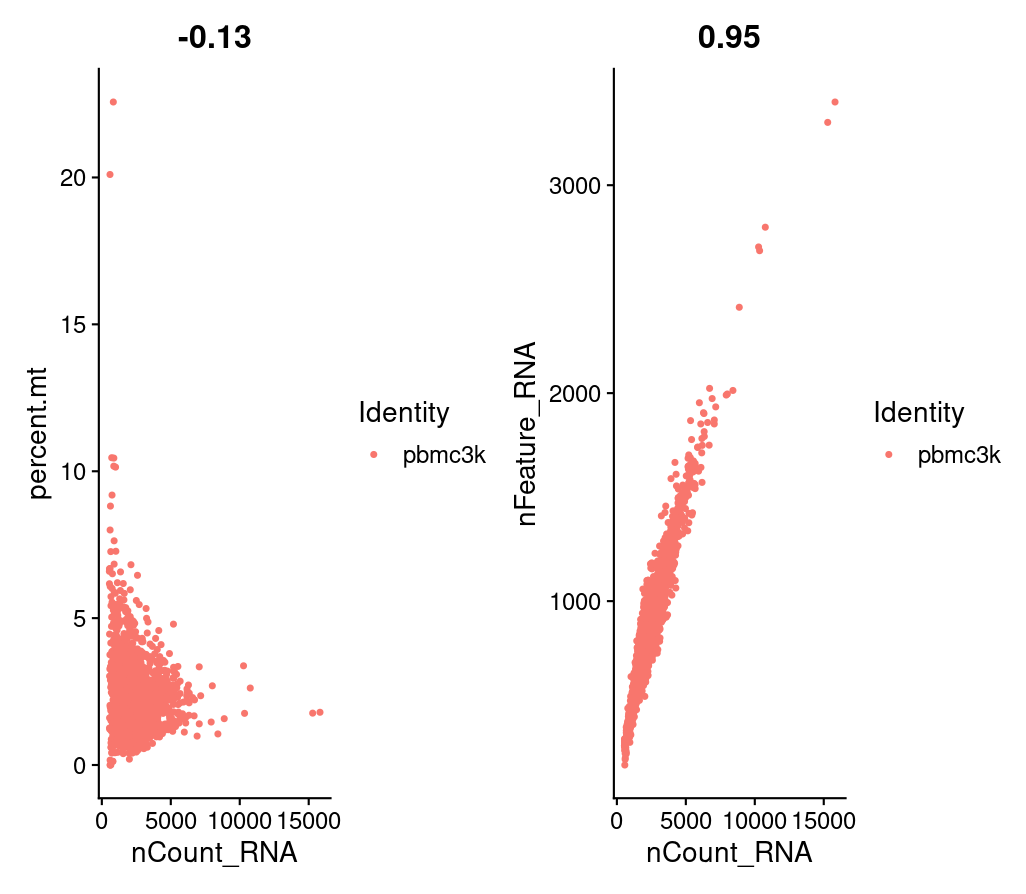
最上面的数字是相关系数
## 过滤
## 官方推荐过滤掉独特特征计数超过2500或小于200的细胞,或者过滤掉超过5%线粒体基因比例的细胞
pbmc <- subset(pbmc, subset = nFeature_RNA > 200 & nFeature_RNA < 2500 & percent.mt < 5)
pbmc
# An object of class Seurat
# 13714 features across 2638 samples within 1 assay
# Active assay: RNA (13714 features, 0 variable features)
可见过滤掉了62个细胞,继续后续分析
数据标准化
各种组学的原始数据普遍存在数据量不统一,数据变化范围过大,数据变化幅度不统一等问题。各种测序数据的分析流程都要对原始数据进行“标准化”,以符合下游分析的需求,单细胞数据也不例外。
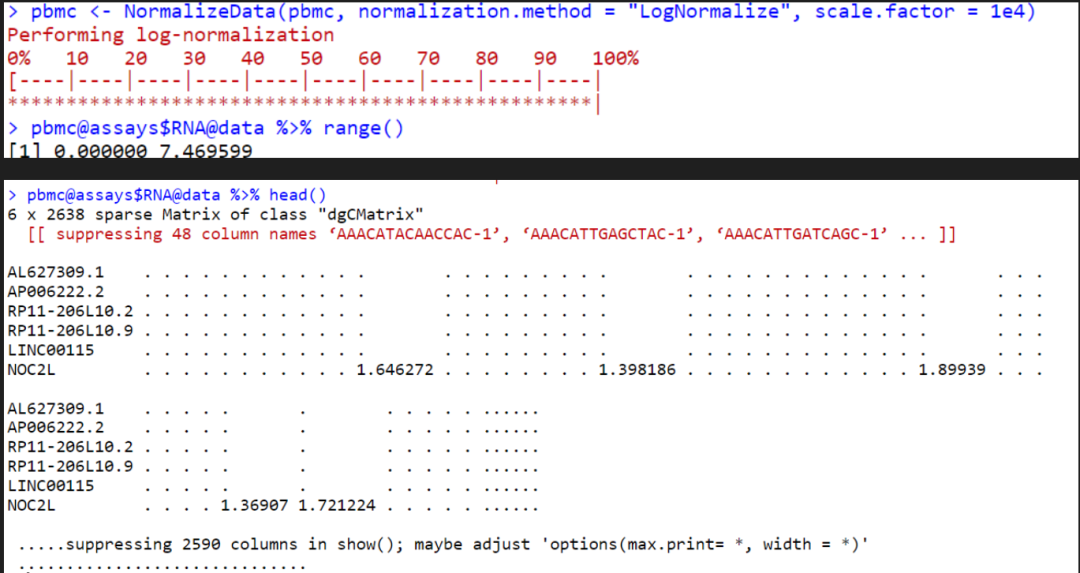
## 标准化
pbmc <- NormalizeData(pbmc, normalization.method = "LogNormalize", scale.factor = 1e4)
标准化方法:

识别高变基因
高变基因:在一些细胞中表达高,另一些细胞中表达低的基因。单细胞表达矩阵为稀疏矩阵(很多0,且为了压缩文件大小,0用.表示),选高变基因可以找到包含信息最多的基因
######识别高变基因#######
pbmc <- FindVariableFeatures(pbmc, selection.method = "vst", nfeatures = 2000) #返回两千个高变基因
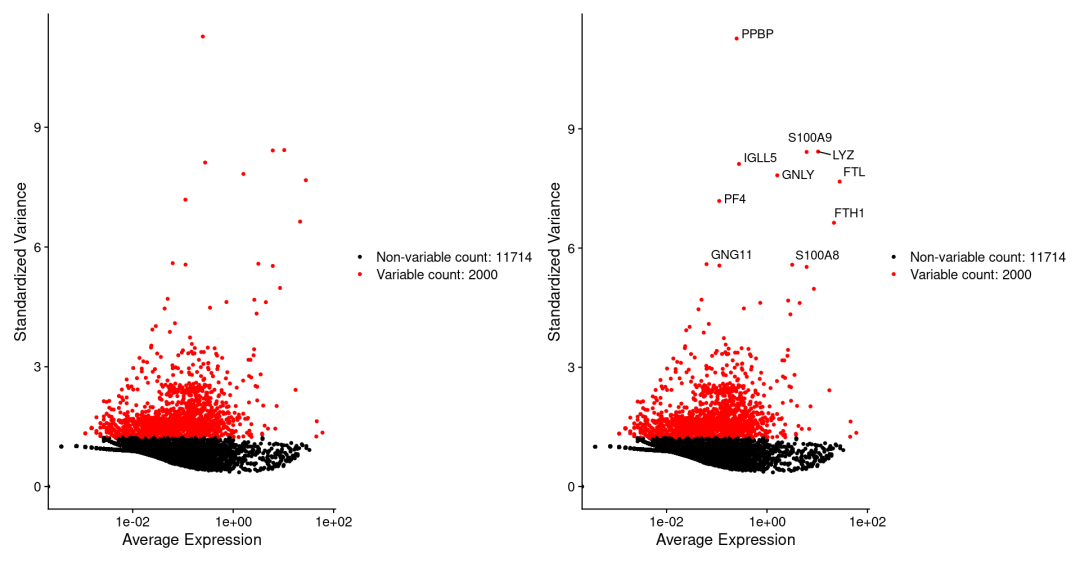
## 提取前10的高变基因
top10 <- head(VariableFeatures(pbmc), 10)
top10
# [1] "PPBP" "LYZ" "S100A9" "IGLL5" "GNLY" "FTL" "PF4" "FTH1" "GNG11" "S100A8"
## 展示高变基因
plot1 <- VariableFeaturePlot(pbmc)
plot2 <- LabelPoints(plot = plot1, points = top10, repel = TRUE)
plot1 + plot2
寻找高变特征使用的方法:

vst方法是一种用于选择具有变异性的高级别变量的方法
- 首先,通过使用局部多项式回归(loess)对log(方差)和log(均值)之间的关系进行拟合。该回归线被用来估计每个特征的期望方差。
- 然后,使用观察到的均值和估计的方差(由拟合的回归线给出)对特征值进行标准化。标准化后,特征的值将根据其所属的期望方差进行调整。
- 标准化后,计算特征的方差。在计算方差之前,可以对标准化值进行截断,限制其最大值(使用clip.max参数)。
通过使用vst方法,我们可以选择具有很高变异性的变量,总的来说就是,先标准化,再根据方差判断变异性。
这里识别高变基因使用的是Seurat包自带的
FindVariableFeatures函数,现在已经有了许多其它方法来探索单细胞数据集的高变基因,如COSG包
数据归一化
细心的同学可能会发现,在这里,我想相较于原推文删掉了其关于归一化的表述,我认为是存在问题的 目前国内对normalize翻译为归一化还是标准化各有说法 因为我前段时间学习了张敬信老师那本tidyverse的书,这里贴出书中的解释以供参考

所以这里的归一化需要区别于前面的标准化,其实就我个人理解前面认为是标准化主要是因为存在log处理
#######数据归一化##########
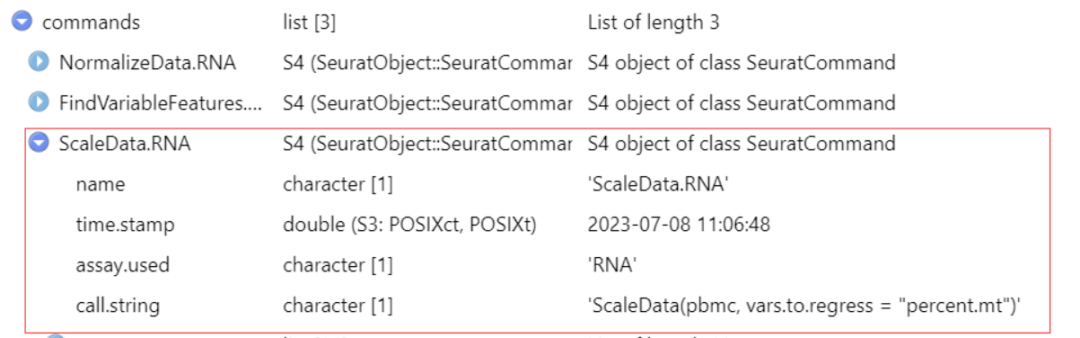
pbmc <- ScaleData(pbmc, vars.to.regress = "percent.mt")
ScaleData()函数将使用vars.to.regress参数指定要回归处理的变量,此处为"percent.mt",即细胞中的MT基因百分比,以消除MT基因的表达量对单细胞数据的影响。

手动查看pbmc内部变化,发现在commands中多了一项ScaleData.RNA
后阅读资料发现,scale.data保存在 pbmc[["RNA"]]@data / pbmc@assays$RNA@data中
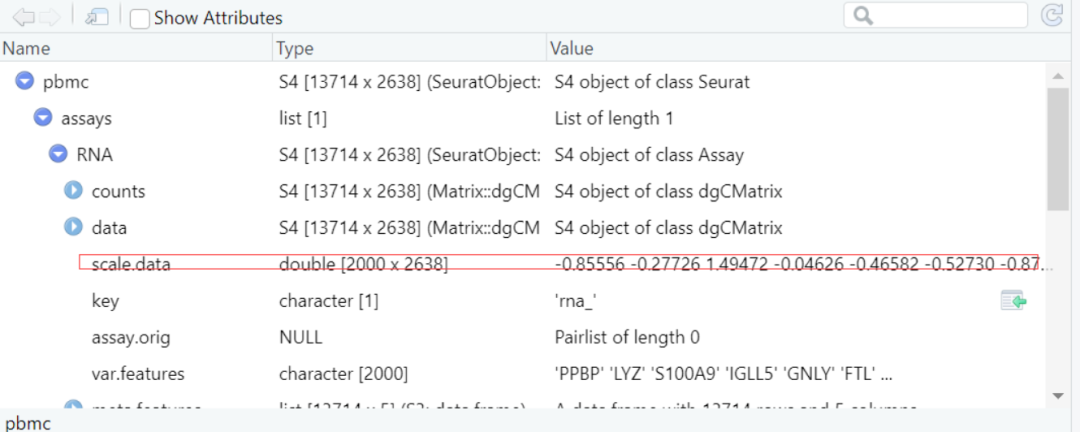
Seurat对象内部结构不清楚,找数据都不好找,基础不牢地动山摇
为了更好地理解数据,同时保证更好地展现,下期我们将一起来看看”Seurat对象内部结构“,通过本文流程跑完得到的最终Seurat对象,对Seurat对象内部结构和工作流程知识进行补全【flag】
降维
PCA降维,默认使用前面2000个高变基因的scale矩阵用于降维。Run开头的函数降维,Find开头的函数聚类。resolution参数表达聚类的分辨率,值越大得到的cluster越多,对于3K细胞的单细胞数据0.4-1.2 通常会得到较好的结果。
pbmc <- RunPCA(pbmc, features = VariableFeatures(object = pbmc)) ##默认会输出5个主成分
PC_ 1
Positive: CST3, TYROBP, LST1, AIF1, FTL, FTH1, LYZ, FCN1, S100A9, TYMP
FCER1G, CFD, LGALS1, LGALS2, SERPINA1, S100A8, CTSS, IFITM3, SPI1, CFP
PSAP, IFI30, COTL1, SAT1, S100A11, NPC2, GRN, LGALS3, GSTP1, PYCARD
Negative: MALAT1, LTB, IL32, IL7R, CD2, B2M, ACAP1, CTSW, STK17A, CD27
CD247, CCL5, GIMAP5, GZMA, AQP3, CST7, TRAF3IP3, SELL, GZMK, HOPX
MAL, MYC, ITM2A, ETS1, LYAR, GIMAP7, KLRG1, NKG7, ZAP70, BEX2
PC_ 2
Positive: CD79A, MS4A1, TCL1A, HLA-DQA1, HLA-DQB1, HLA-DRA, LINC00926, CD79B, HLA-DRB1, CD74
HLA-DMA, HLA-DPB1, HLA-DQA2, CD37, HLA-DRB5, HLA-DMB, HLA-DPA1, FCRLA, HVCN1, LTB
BLNK, P2RX5, IGLL5, IRF8, SWAP70, ARHGAP24, FCGR2B, SMIM14, PPP1R14A, C16orf74
Negative: NKG7, PRF1, CST7, GZMA, GZMB, FGFBP2, CTSW, GNLY, B2M, SPON2
CCL4, GZMH, FCGR3A, CCL5, CD247, XCL2, CLIC3, AKR1C3, SRGN, HOPX
TTC38, CTSC, APMAP, S100A4, IGFBP7, ANXA1, ID2, IL32, XCL1, RHOC
PC_ 3
Positive: HLA-DQA1, CD79A, CD79B, HLA-DQB1, HLA-DPA1, HLA-DPB1, CD74, MS4A1, HLA-DRB1, HLA-DRA
HLA-DRB5, HLA-DQA2, TCL1A, LINC00926, HLA-DMB, HLA-DMA, CD37, HVCN1, FCRLA, IRF8
PLAC8, BLNK, MALAT1, SMIM14, PLD4, IGLL5, SWAP70, P2RX5, LAT2, FCGR3A
Negative: PPBP, PF4, SDPR, SPARC, GNG11, NRGN, GP9, RGS18, TUBB1, CLU
HIST1H2AC, AP001189.4, ITGA2B, CD9, TMEM40, PTCRA, CA2, ACRBP, MMD, TREML1
NGFRAP1, F13A1, SEPT5, RUFY1, TSC22D1, CMTM5, MPP1, MYL9, RP11-367G6.3, GP1BA
PC_ 4
Positive: HLA-DQA1, CD79B, CD79A, MS4A1, HLA-DQB1, CD74, HLA-DPB1, HIST1H2AC, HLA-DPA1, HLA-DRB1
TCL1A, PF4, HLA-DQA2, SDPR, HLA-DRA, LINC00926, PPBP, GNG11, HLA-DRB5, SPARC
GP9, PTCRA, CA2, AP001189.4, CD9, NRGN, RGS18, GZMB, CLU, TUBB1
Negative: VIM, IL7R, S100A6, S100A8, IL32, S100A4, GIMAP7, S100A10, S100A9, MAL
AQP3, CD14, CD2, LGALS2, FYB, GIMAP4, ANXA1, RBP7, CD27, FCN1
LYZ, S100A12, MS4A6A, GIMAP5, S100A11, FOLR3, TRABD2A, AIF1, IL8, TMSB4X
PC_ 5
Positive: GZMB, FGFBP2, S100A8, NKG7, GNLY, CCL4, PRF1, CST7, SPON2, GZMA
GZMH, LGALS2, S100A9, CCL3, XCL2, CD14, CLIC3, CTSW, MS4A6A, GSTP1
S100A12, RBP7, IGFBP7, FOLR3, AKR1C3, TYROBP, CCL5, TTC38, XCL1, APMAP
Negative: LTB, IL7R, CKB, MS4A7, RP11-290F20.3, AQP3, SIGLEC10, VIM, CYTIP, HMOX1
LILRB2, PTGES3, HN1, CD2, FAM110A, CD27, ANXA5, CTD-2006K23.1, MAL, VMO1
CORO1B, TUBA1B, LILRA3, GDI2, TRADD, ATP1A1, IL32, ABRACL, CCDC109B, PPA1
pbmc <- FindNeighbors(pbmc, dims = 1:10) # 使用维度非输出维度
pbmc <- FindClusters(pbmc, resolution = 0.5)
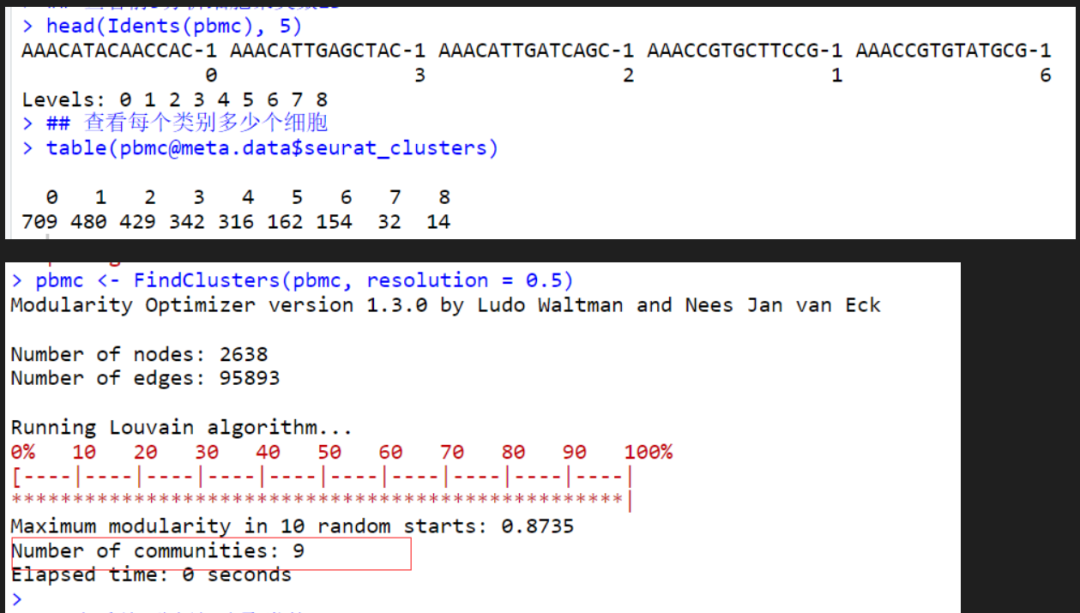
发现,我们得到的0.5分辨率下cluster数量(9)和原推文(10)对不上
原推文:
## 查看前5分析细胞聚类数ID
head(Idents(pbmc), 5)
# AAACATACAACCAC-1 AAACATTGAGCTAC-1 AAACATTGATCAGC-1 AAACCGTGCTTCCG-1 AAACCGTGTATGCG-1
# 3 2 8 4 3
# Levels: 0 1 2 3 4 5 6 7 8 9
## 查看每个类别多少个细胞
table(pbmc@meta.data$seurat_clusters)
# 0 1 2 3 4 5 6 7 8 9
# 598 483 345 324 300 214 159 106 96 13
# 上述结果可见,分为10类,由0-9,细胞数依次递减
查看帮助文档:
FindNeighbors函数通过计算数据集中每个细胞之间的相似性,找到每个细胞的k个最近邻,并可选地计算共享最近邻图。这些最近邻关系可以用来构建细胞之间的连接,用于后续的聚类分析、可视化和其他细胞间关系的研究
通过调用 FindClusters函数,可以根据细胞之间的共享最近邻关系,在数据集中识别出具有相似性的细胞聚类
提高分辨率resolution到0.8,使clusters分组达到10,与原代码结果仍不完全一样,怀疑是R包更新的问题?
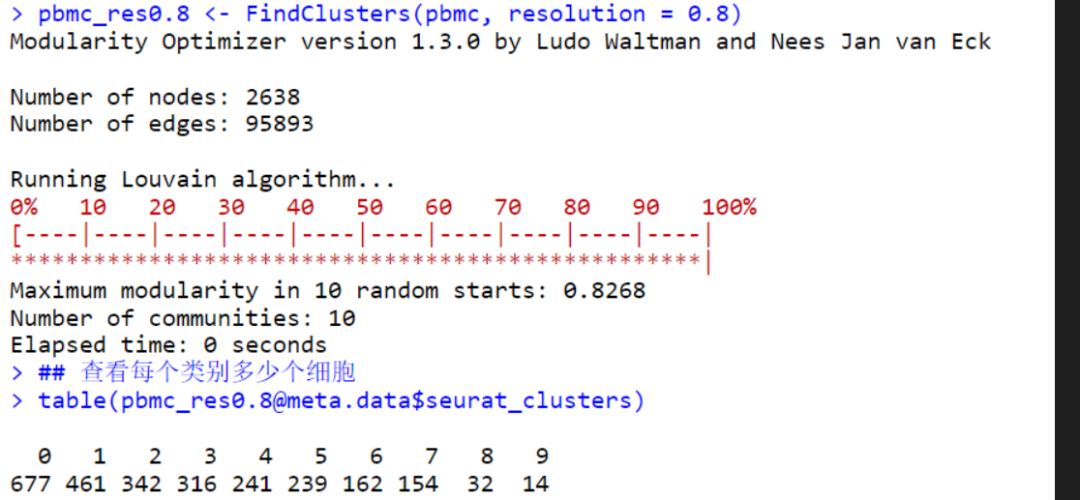
UMAP/tSNE可视化
降维可以将相似的细胞放置在低维度的空间
pbmc <- RunUMAP(pbmc, dims = 1:10) # Which dimensions to use as input features, used only if features is NULL
p1 <- DimPlot(pbmc, reduction = "umap", label = T, label.size = 5)
pbmc <- RunTSNE(pbmc, dims = 1:10)
p2 <- DimPlot(pbmc, reduction = "tsne", label = T, label.size = 5)
p1+p2
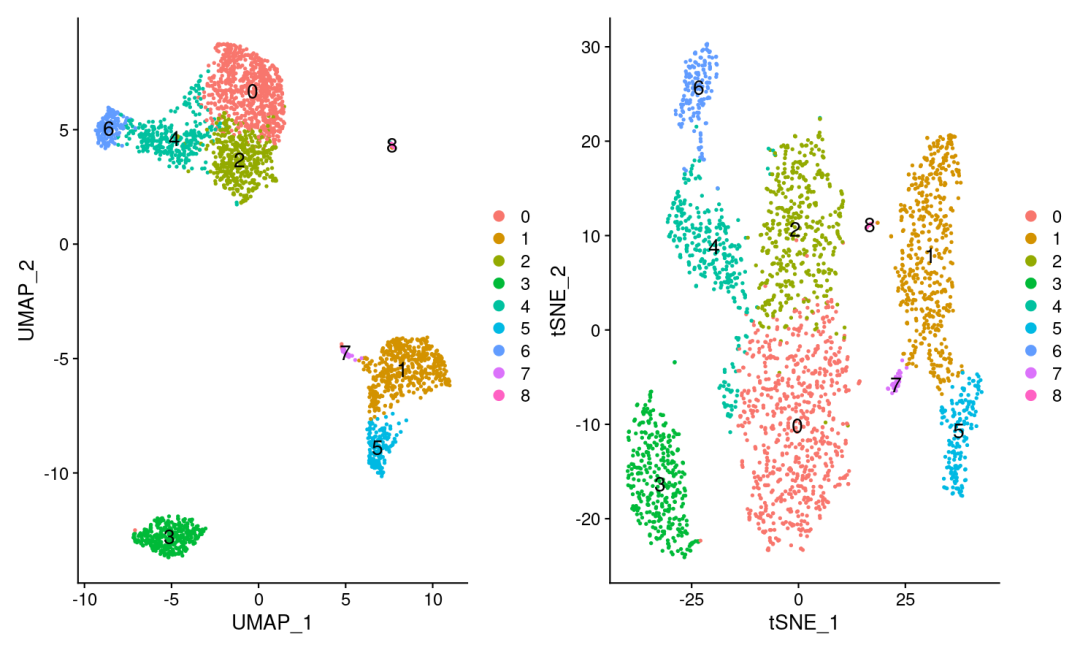
这是原推文降维分组可视化情况:
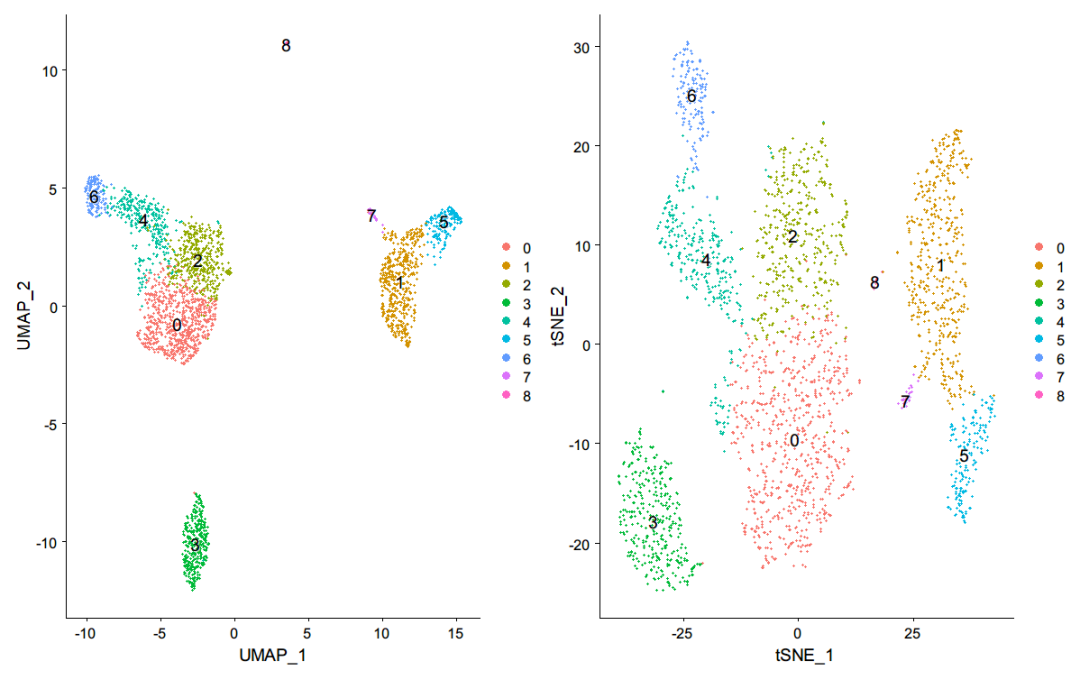
可以发现也是9个分组,与原文前面10个分组不一致,和我们0.5分辨率下分组一致,所以原文降维处应有问题
在umap图中,cluster之间的距离更明显,图形也更美观
Marker鉴定细胞
根据生物学背景知识,将表达相应Marker基因的各个单细胞亚群的重命名
自定义
#查看B细胞marker基因在不同聚类中表达情况
VlnPlot(pbmc, features = c("MS4A1", "CD79A"))
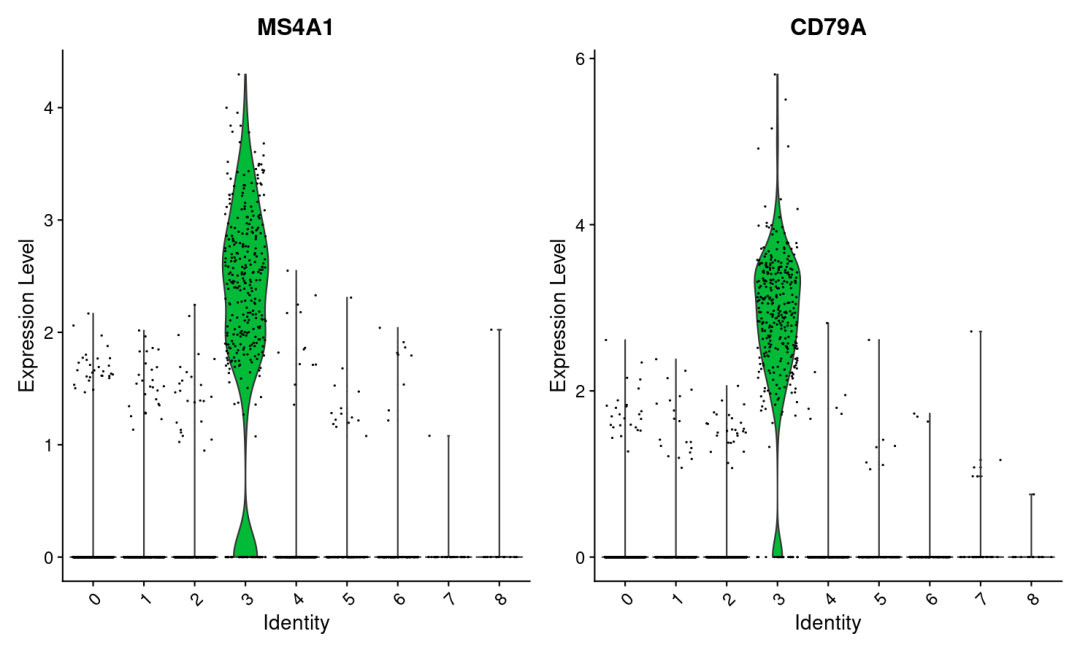
可视化展示marker基因的坐标映射分布
FeaturePlot(pbmc, features = c("MS4A1", "GNLY", "CD3E",
"CD14", "FCER1A", "FCGR3A",
"LYZ", "PPBP", "CD8A"))
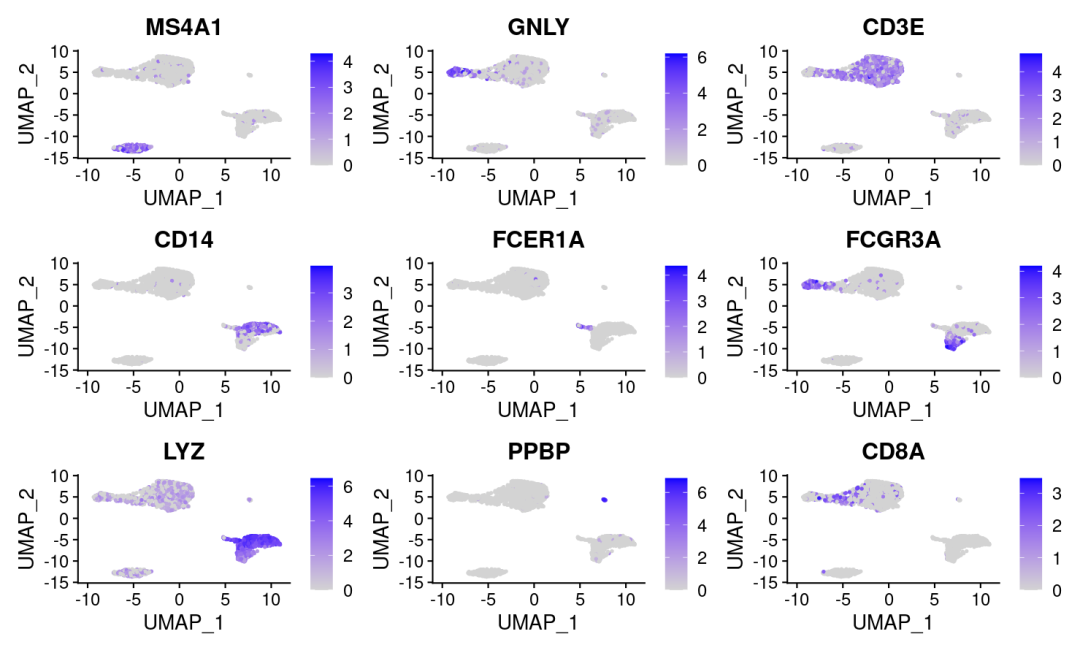
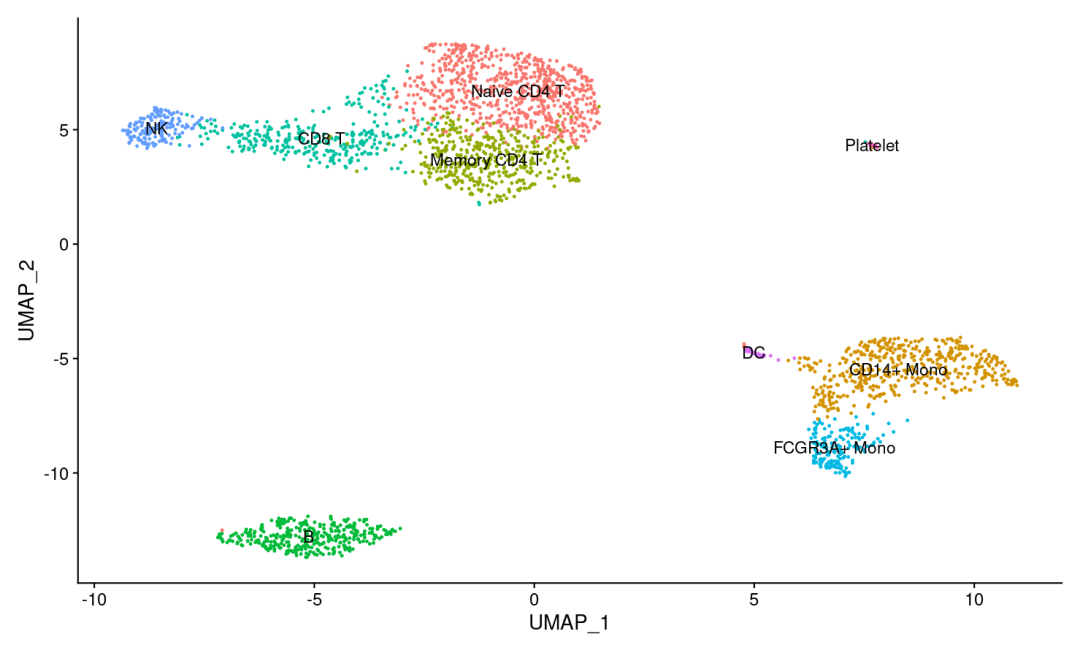
上图结合UMAP可视化中的聚类对聚类重命名(根据marker自定义),会得到如下所示的分群情况
new.cluster.ids <- c("Naive CD4 T", "CD14+ Mono", "Memory CD4 T",
"B", "CD8 T", "FCGR3A+ Mono", "NK", "DC", "Platelet")
names(new.cluster.ids) <- levels(pbmc)
pbmc <- RenameIdents(pbmc, new.cluster.ids)
DimPlot(pbmc, reduction = 'umap',
label = TRUE, pt.size = 0.5) + NoLegend()
检查各分组内基因表达情况
这个时候有5个可视化方法选择,分别是:小提琴图,坐标映射图,峰峦图,气泡图,热图。代码参见可视化单细胞亚群的标记基因的5个方法 其实这个部分和前面通过marker鉴定细胞类型可视化方法一致
- 小提琴图
VlnPlot(pbmc, features = c("MS4A1", "CD79A"))
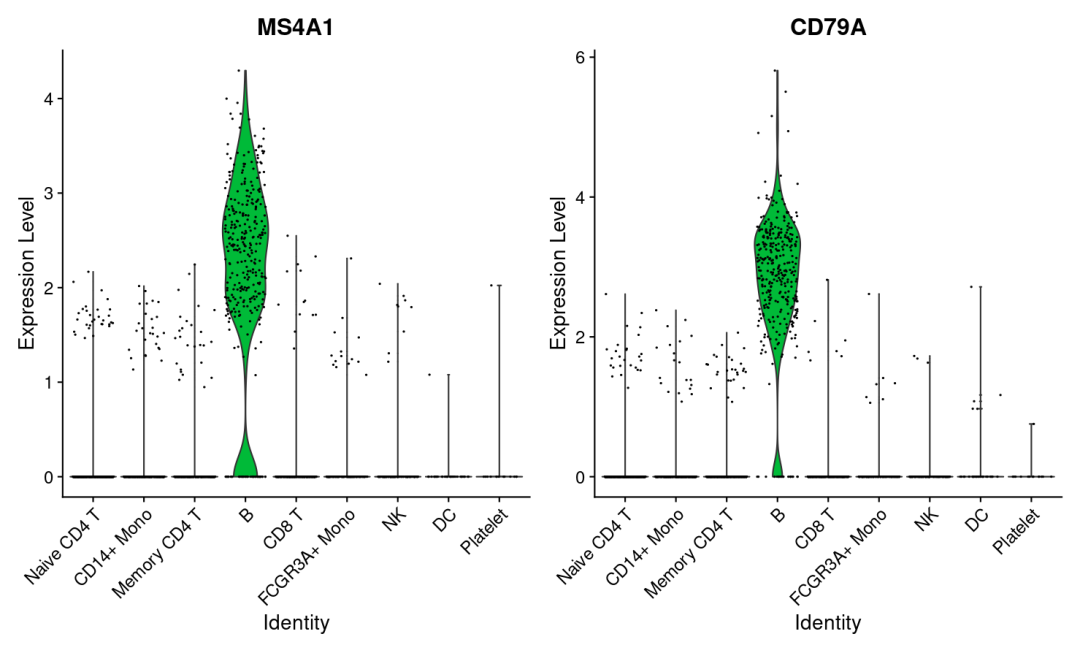
- 坐标映射图
FeaturePlot(pbmc, features = c("MS4A1", "CD79A"))
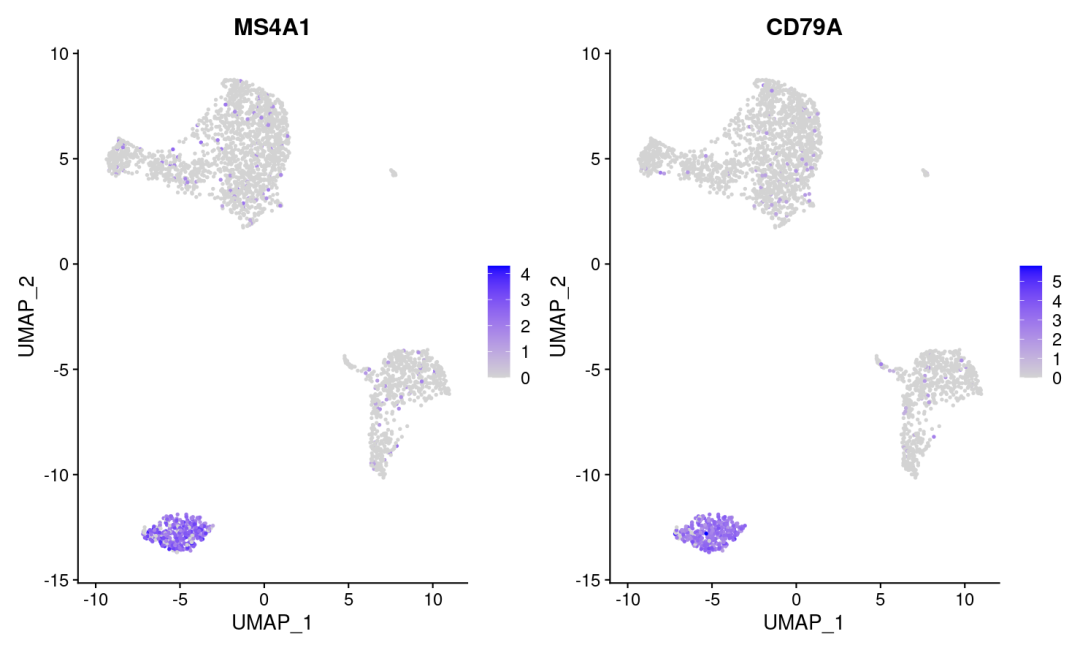
- 峰峦图
RidgePlot(pbmc, features = c("MS4A1", "CD79A"), ncol = 1)
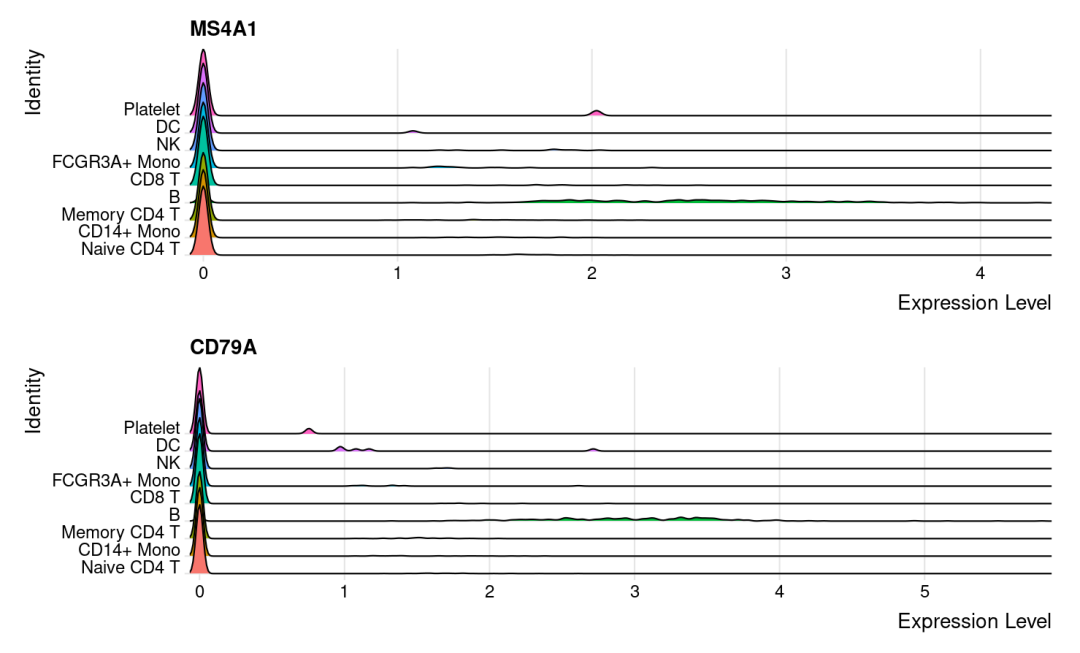
- 气泡图
features= c('IL7R', 'CCR7','CD14', 'LYZ', 'IL7R', 'S100A4',"MS4A1", "CD8A",'FOXP3',
'FCGR3A', 'MS4A7', 'GNLY', 'NKG7',
'FCER1A', 'CST3','PPBP')
DotPlot(pbmc, features = unique(features)) + RotatedAxis()
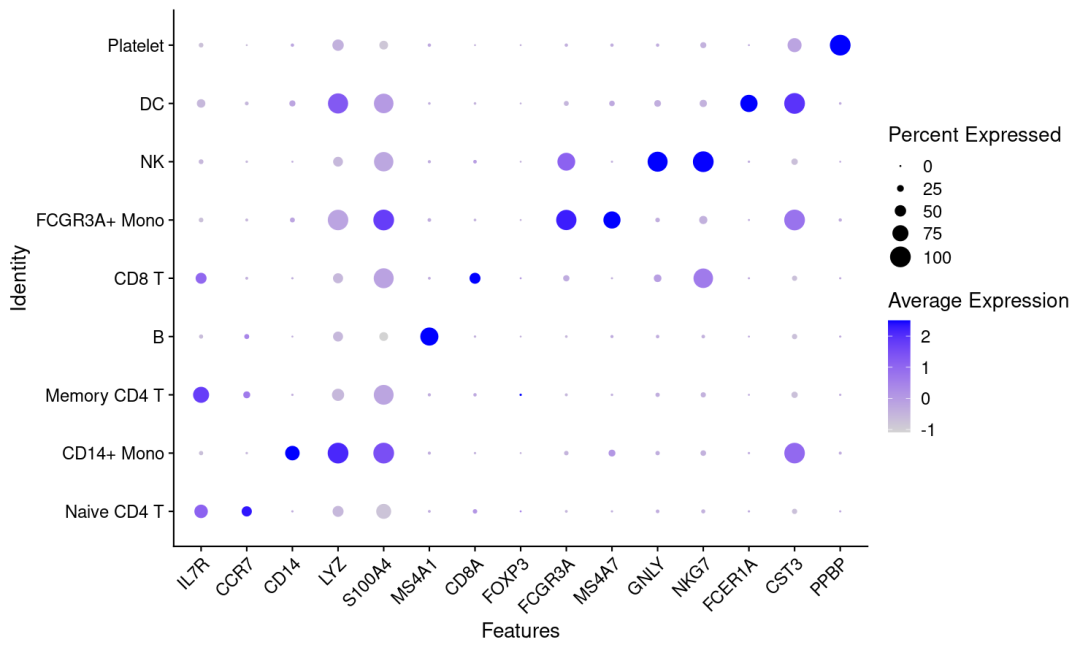
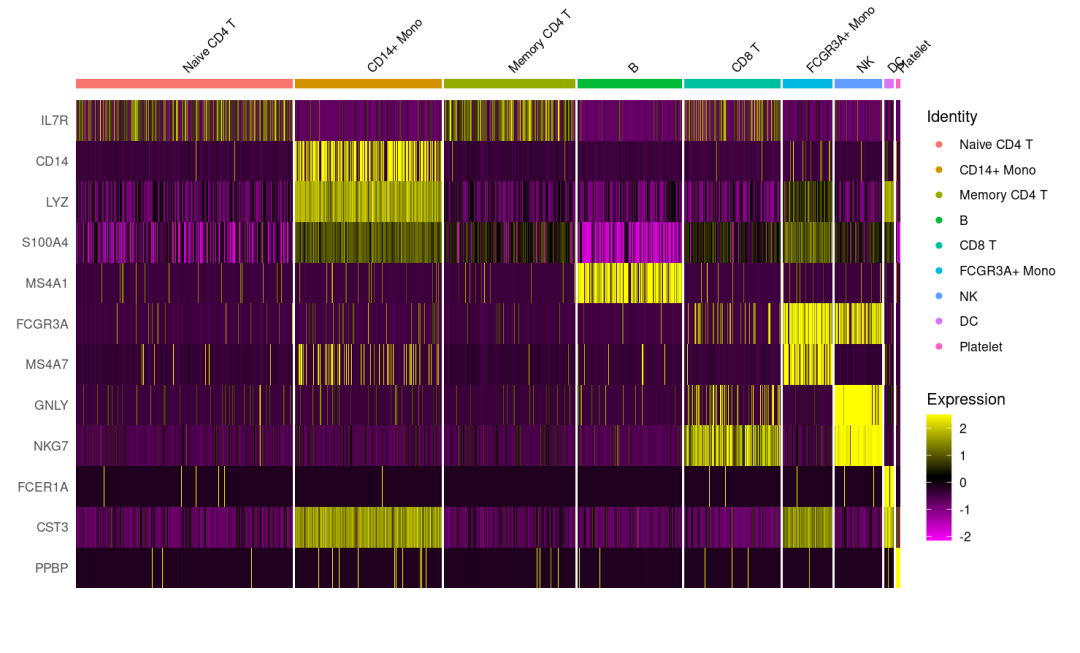
- 热图
DoHeatmap(subset(pbmc ), features = features, size = 3)
本文参与 腾讯云自媒体同步曝光计划,分享自微信公众号。
原始发表:2023-07-26,如有侵权请联系 cloudcommunity@tencent.com 删除
评论
登录后参与评论
推荐阅读
目录

