SCI ADV | 用POLARIS进行细胞组成推断和层特异性空间转录谱鉴定
SCI ADV | 用POLARIS进行细胞组成推断和层特异性空间转录谱鉴定

基于测序的ST技术提供全转录组图谱,但受到非单细胞水平分辨率的限制,对每个点的细胞数量或细胞类型组成缺乏了解会导致下游分析无效,这是ST数据分析中公认的关键问题。目前开发的分析方法往往未充分利用组织学图像信息。近日,《Science Advances》发表了一种多功能的ST分析方法:POLARIS,可以执行细胞类型去卷积,识别解剖学或功能性分层差异表达 (LDE) 基因,并能够从组织学图像推断细胞组成。

POLARIS是什么?

POLARIS是一种基于概率的推断方法,其集成scRNA-seq参考和ST数据与注释层信息。通过检查组织学图像和协同表达谱信息,合理地推断对应不同生物学功能的层或子区域(例如肿瘤活检组织中的癌症区域与非癌症区域,大脑皮层样本中的不同层,以及心脏中的心室和心房区域)。通过明确地允许和建模层特异性基因表达模式,POLARIS不仅能够高精度地识别细胞类型组成,而且还可以识别LDE基因,同时校正差异细胞组成。POLARIS的另一个关键特征是它可以灵活地利用组织学图像。该功能还使POLARIS能够根据没有基因表达测量的区域图像(即斑点之间的区域)推断超分辨率细胞组成,以及仅根据原始H&E染色图像预测细胞组成。
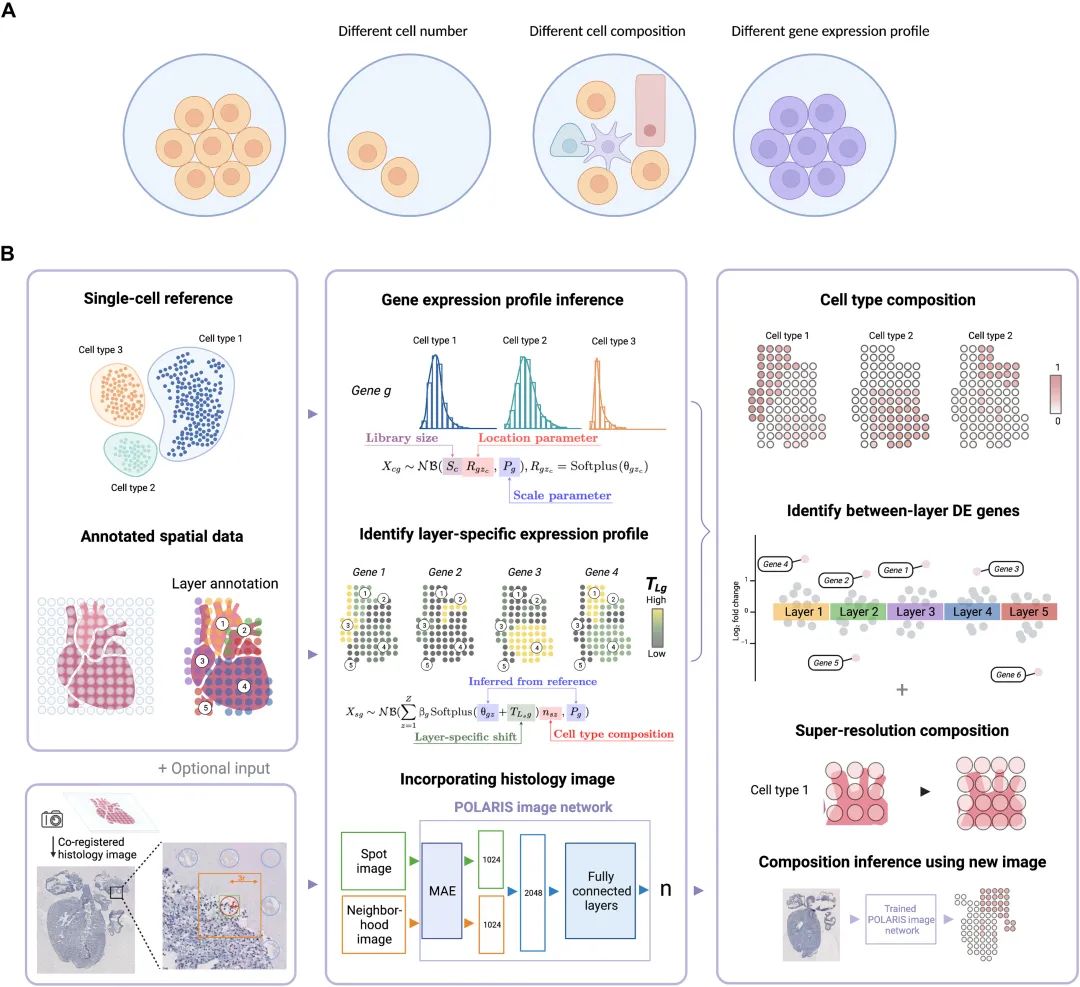
POLARIS概述
POLARIS将单细胞参考和注释空间数据作为输入,推断细胞类型特异性基因表达谱,并识别层特异性表达谱。当提供组织学图像(作为可选输入)时,POLARIS将另外训练图像网络。POLARIS的输出包括推断的点水平细胞类型组成、识别的LDE基因和可应用于独立图像的预训练POLARIS图像网络。
POLARIS的性能测试

根据来自多个组织的数据对POLARIS的性能进行评估,这些组织包括小鼠皮层、发育中的人类心脏和HER2+乳腺癌样本。
与其他去卷积方法相比,POLARIS有力地证明了跨组织的高去卷积精度,仅根据图像准确预测细胞组成,并识别具有生物学相关性/意义的LDE基因。性能测试的结果展示了POLARIS在以下三个方面的优势:去卷积精度、LDE基因识别和图像预测。
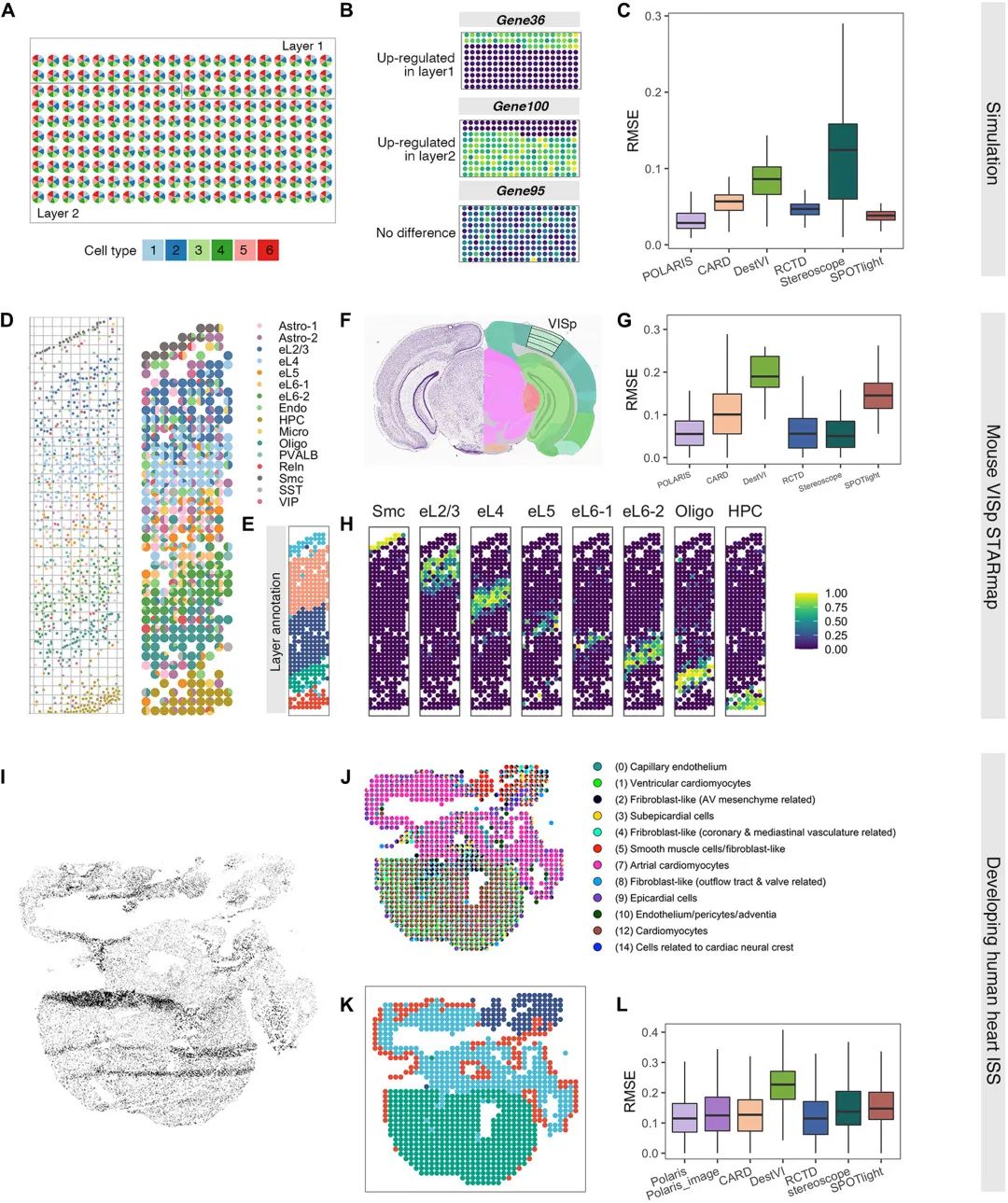
POLARIS的去卷积精度
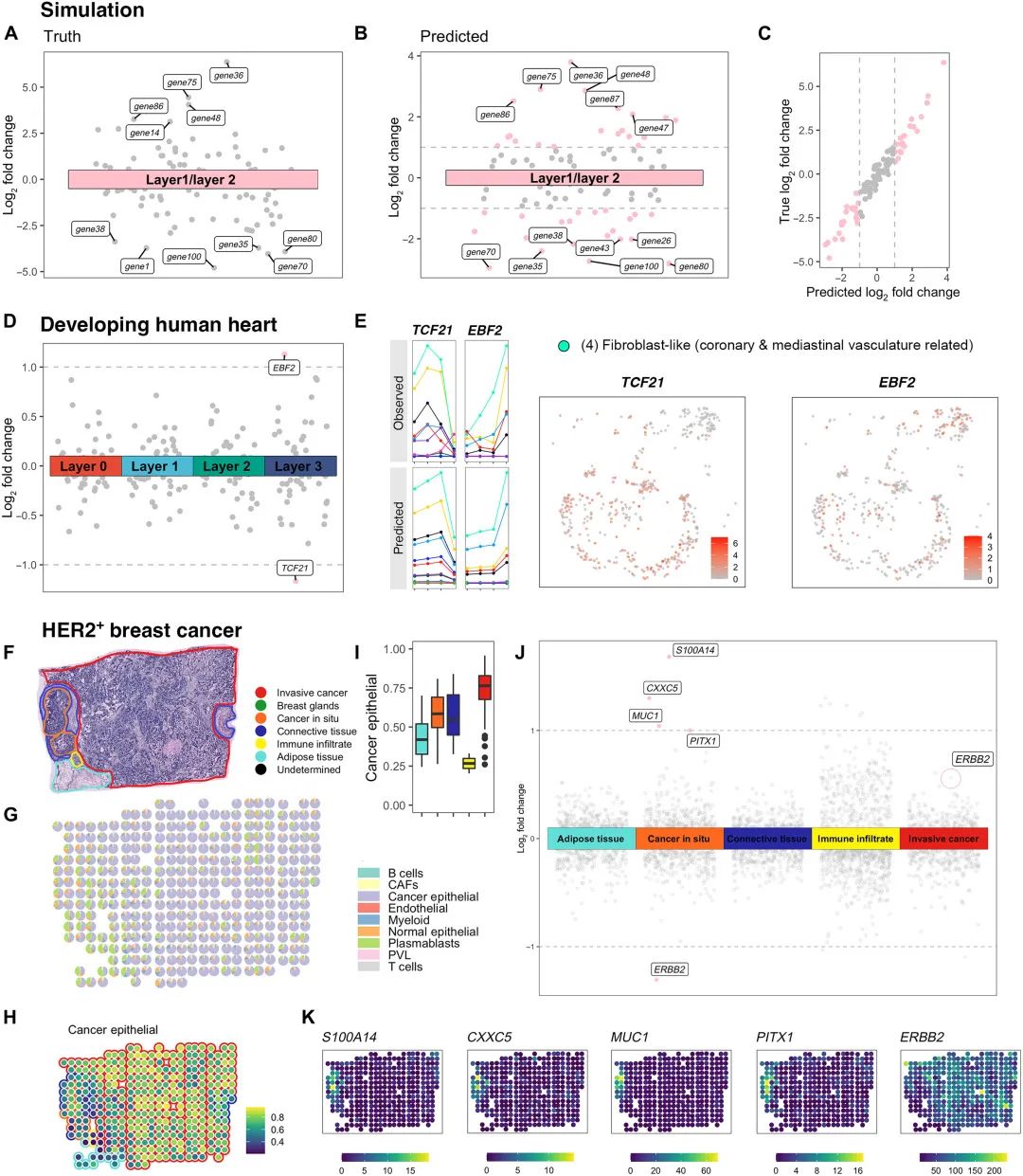
POLARIS 在三个数据集上识别的 LDE 基因
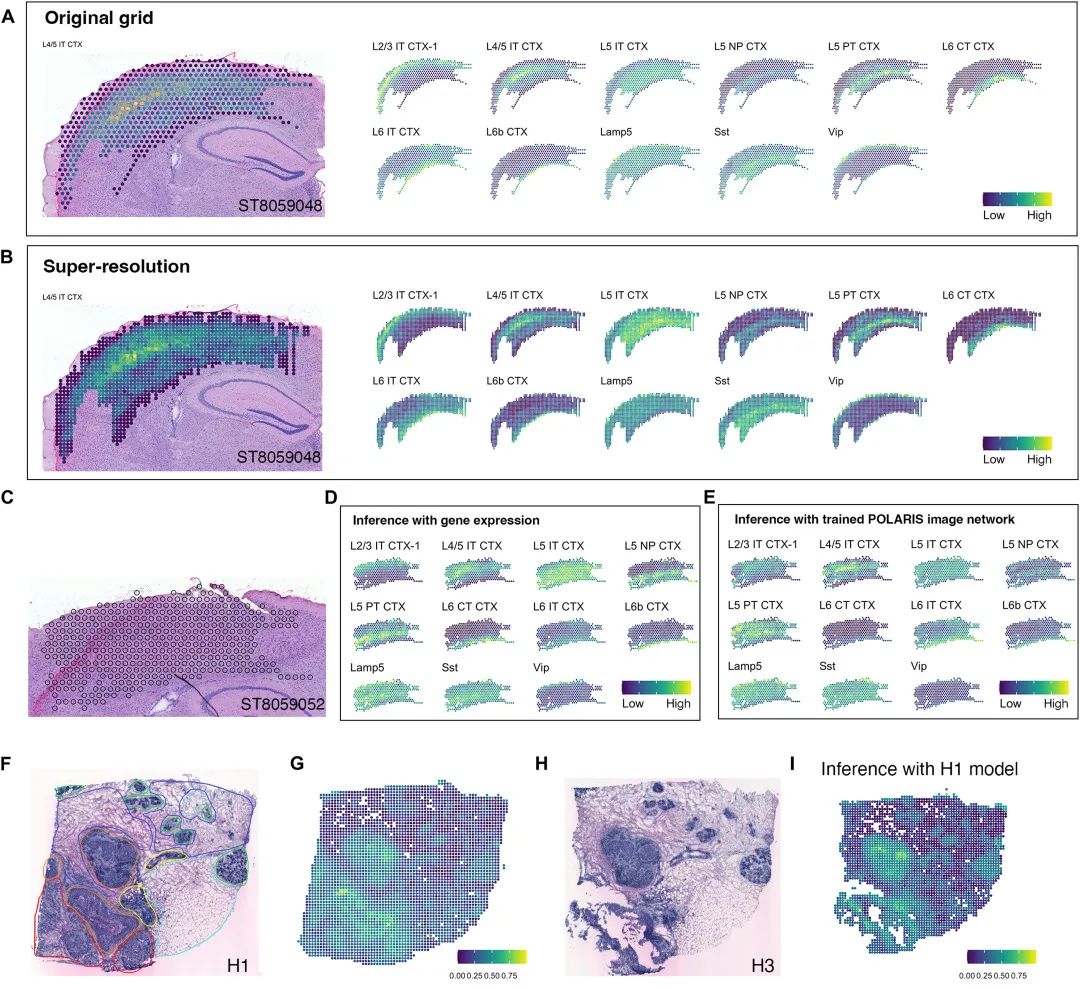
当使用组织图像作为输入时,POLARIS实现了超分辨率的细胞成分推断
POLARIS引入了一种全新的方法,可以纯粹从组织学图像推断细胞组成,相信POLARIS 结合组织学图像以阐明层特异性基因表达模式的多功能能力将推动空间生物学的发现。
POLARIS的源代码可在如下链接获取(👉 可点击阅读原文访问):
https://doi.org/10.5281/zenodo.7302022
