一些辅助划分单细胞亚型的工具
示例数据
基本上每个人开始学习单细胞,都是从这个文档开始:https://satijalab.org/seurat/articles/pbmc3k_tutorial.html
这个数据集之前用过:初探单细胞下游
rm(list=ls())
library(Seurat)
library(patchwork)
library(tidyverse)
library(AUCell)
# Load the PBMC dataset
# https://cf.10xgenomics.com/samples/cell/pbmc3k/pbmc3k_filtered_gene_bc_matrices.tar.gz
pbmc.data <- Read10X(data.dir = "filtered_gene_bc_matrices/hg19/")
# Initialize the Seurat object with the raw (non-normalized data).
pbmc <- CreateSeuratObject(counts = pbmc.data, project = "pbmc3k", min.cells = 3, min.features = 200)
pbmc

单细胞亚群标记基因列表
Bcells = c('MS4A1','SDC1','CD27','CD38','CD19', 'CD79A')
counts <- GetAssayData(object = pbmc, slot = "counts")
head(counts)
cell_rankings <- AUCell_buildRankings(counts)
cell_rankings
cells_AUC <- AUCell_calcAUC(Bcells, cell_rankings)
cells_AUC
cells_assignment <- AUCell_exploreThresholds(cells_AUC, plotHist = TRUE, assign=TRUE)
thr = cells_assignment$geneSet$aucThr$selected;thr
new_cells <- names(which(getAUC(cells_AUC)["geneSet",]> thr))
pbmc$is_list <- ifelse(colnames(pbmc) %in% new_cells, "list", "non_list")
colnames(pbmc@meta.data)
# DimPlot(object = pbmc, group.by = "is_list", label = TRUE) +
# DimPlot(object = pbmc, group.by = "seurat_annotations", label = TRUE)
代码拆解:

img
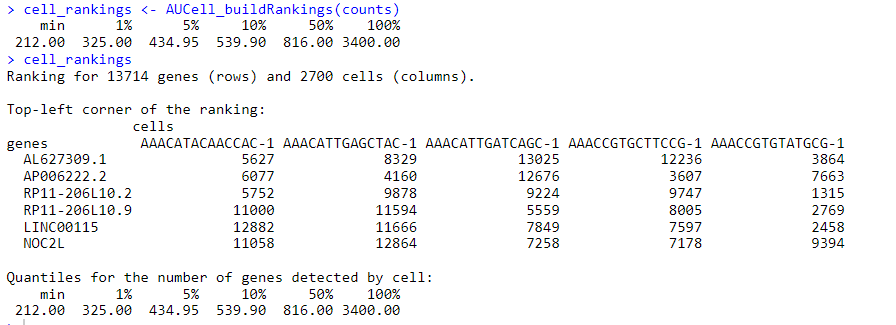
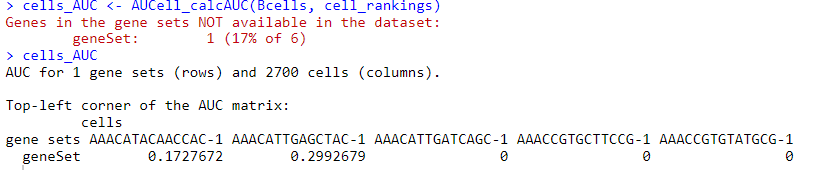
我们前面Bcells输入了6个基因,有一个不在该数据集features中,感觉计算AUC类似计算一个分数,后面就要找分数显著阈值


标记Bcells marker 列表 AUC得分显著显著的细胞

使用初探单细胞下游推文代码实现一下基本流程:
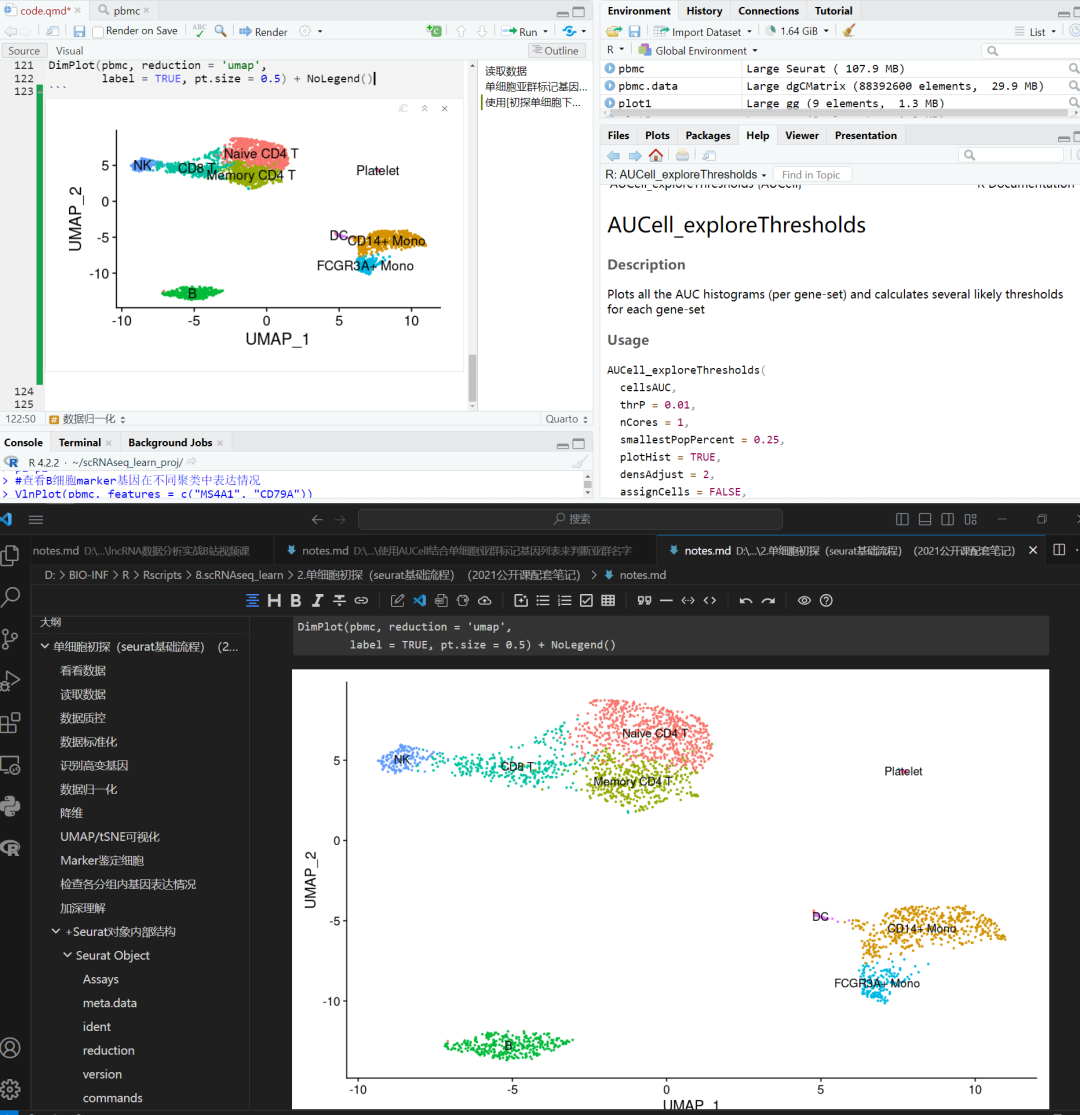
可视化AUCells结果
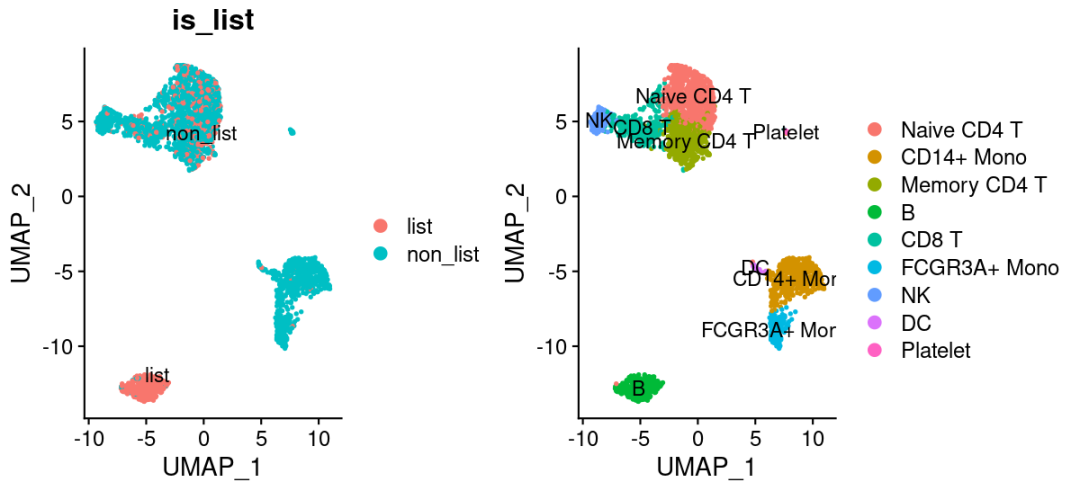
DimPlot(object = pbmc, group.by = "is_list", label = TRUE) +
DimPlot(object = pbmc, label = TRUE)
局限性:
这里测试了多种单细胞亚群的标记基因列表,发现如果是比较过于类似的细胞是没办法区分的,比如 CD14+ Mono 和 FCGR3A+ Mono,以及 Memory CD4 T和 Naive CD4 T ,或者说是需要首先区分了它们后找到了它们的特异性基因然后再使用这些基因才有可能区分它们,就陷入了一个鸡生蛋蛋生鸡的死循环。。。。 但是,对b细胞来说,就没有这个问题, 因为它和t细胞是截然不同,而且也不可能去跟髓系免疫细胞混淆。。。
可以看到基本上是近乎完美的区分了b细胞和其它细胞,就需要实现知道所有的单细胞亚群的标记基因然后去一个个测试。所以这种方法并不是单细胞亚群命名的主流操作,仅仅是特殊情况下,需要去确定某个未知的单细胞亚群或者说想搞清楚某个状态的单细胞亚群或者某个通路激活的单细胞才会这样的操作 更多的用处可以是针对某个亚群显示多个基因的打分
scGate: marker-based purification of cell types from heterogeneous single-cell RNA-seq datasets(https://github.com/carmonalab/scGate)
scGate 是一个 R 软件包,它将典型的基于人工标记的细胞类型注释方法自动化,从而能够从复杂的 scRNA-seq 数据集中准确、直观地纯化出感兴趣的细胞群,而无需参考基因表达谱或训练数据。
scGate 基于 UCell 进行稳健的单细胞特征评分,并基于 Seurat 这一全面而强大的单细胞 omics 分析框架。
简而言之,scGate 的输入包括:i) 存储在 Seurat 对象中的基因表达矩阵;ii) 一个 "门控模型"(GM),由一组定义相关细胞群的标记基因组成。基因门控模型可以是简单的单一标记基因,也可以是阳性和阴性标记基因的组合。更复杂的 GM 可以分级方式构建,类似于流式细胞仪中使用的门控策略。
scGate 使用基于等级的方法 UCell 评估每个细胞中特征标记表达的强度,然后通过计算相邻细胞的平均 UCell 分数来执行 k 近邻(kNN)平滑。最后,根据用户提供的门控模型生成的二元决策树中,会对 kNN 平滑特征得分应用一个通用阈值,从而将细胞注释为 "纯 "或 "不纯",与感兴趣的细胞群相关。
Testing the package
使用scGate使用手动定义的标记基因纯化感兴趣的细胞群体
library(scGate)
#Get a test scRNA-seq dataset (as a list of Seurat objects)
sample.data.seurat.list <- scGate::get_testing_data()
seurat_object <- sample.data.seurat.list$Satija
#Manually define a simple scGate gating model to purify eg. Natural Killer (NK) cells, using a positive marker KLRD1 and negative marker CD3D
my_scGate_model <- gating_model(name = "NK", signature = c("KLRD1","CD3D-"))
#scGate it!
seurat_object <- scGate(data = seurat_object, model = my_scGate_model)
#Use Seurat to visualize "Pure" and "Impure" cells
DimPlot(seurat_object, group.by = "is.pure")
#Use Seurat to subset pure cells
seurat_object_purified <- subset(seurat_object, subset = `is.pure` == "Pure" )
这段代码使用的测试数据集需要魔法否则无法获取,所以这里只作示例看看用法,我们还是具体到使用上面pbmc的数据集
可以发现所需输入和前面AUCell类似:seurat对象和标记基因列表
> seurat_object <- pbmc
> my_scGate_model <- gating_model(name="Bcells",signature = Bcells)
> Bcells
[1] "MS4A1" "SDC1" "CD27" "CD38" "CD19" "CD79A"
>
> seurat_object <- scGate(data = seurat_object, model = my_scGate_model)
Warning: The following genes were not found and will be
imputed to exp=0:
* SDC1
### Detected a total of 337 pure 'Target' cells (12.77% of total)
> DimPlot(seurat_object, group.by = "is.pure")
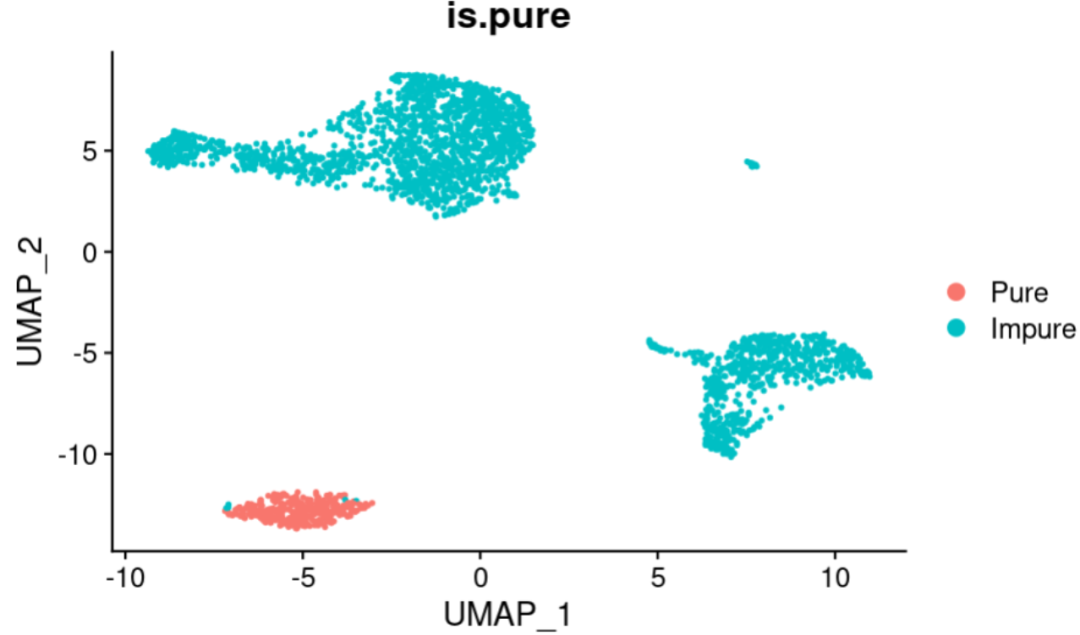
感觉效果比AUCell好呢
Pre-defined Gating Models
A database of gating models for scGate is available on scGate_models and can be loaded using get_scGateDB()
https://github.com/carmonalab/scGate_models
#Get scGate database of pre-defined gating models
scGate_models_DB <- get_scGateDB()
#For example, filter abT cells using one of scGate pre-defined gating models
seurat_object <- scGate(seurat_object, model = scGate_models_DB$human$generic$Tcell.alphabeta)
DimPlot(seurat_object)
The first time you run get_scGateDB() the database will be downloaded from the repository. On successive calls it will load your local version of the DB.
这个包的仓库还提供了一些定义好的scGate_model,也就是细胞亚型的markers
我们根据提供的代码进行修改,将model参数使用的markers改为Bcells的,看看
> #Get scGate database of pre-defined gating models
> scGate_models_DB <- get_scGateDB()
trying URL 'https://github.com/carmonalab/scGate_models/archive/master.zip'
downloaded 27 KB
>
> #For example, filter abT cells using one of scGate pre-defined gating models
> seurat_object <- scGate(seurat_object, model = scGate_models_DB$human$generic$Bcell)
Warning: The following genes were not found and will be
imputed to exp=0:
* COL1A1,RAMP2,SFTPB,SLPI,WFDC2,PIGR,SLC34A2,SFTA2,AGR3,ELF3,KRT19,COL1A2,COL3A1,DCN,LUM,THY1,MSR1Warning: pseudoinverse used at -2.2368Warning: neighborhood radius 0.30103Warning: reciprocal condition number 1.9598e-16
### Detected a total of 345 pure 'Target' cells (13.08% of total)
>
> DimPlot(seurat_object)
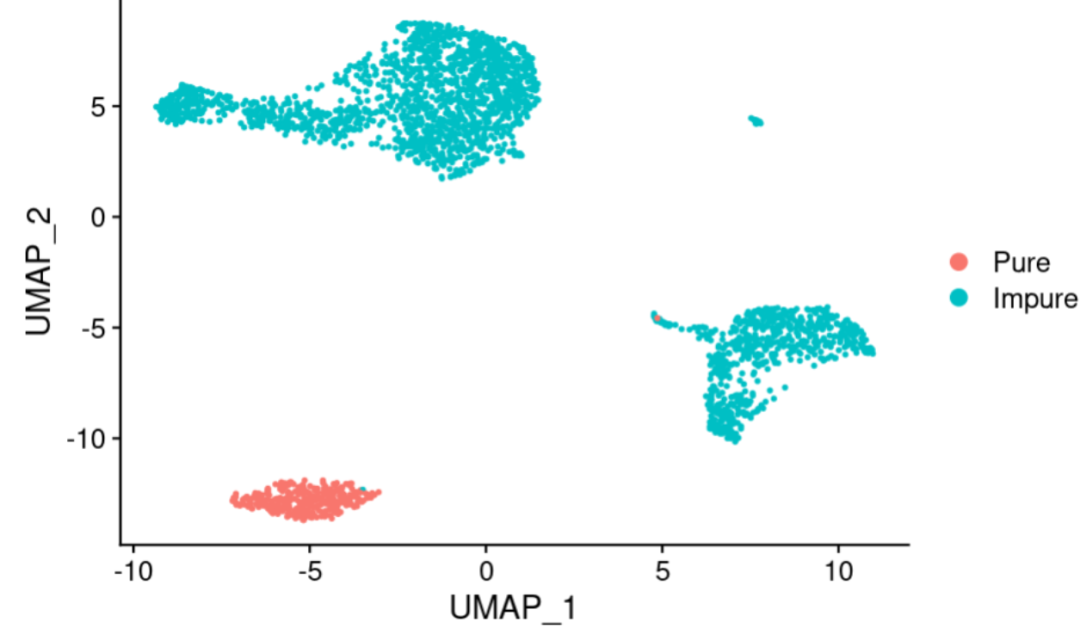
效果也很好
第二次调用Pre-defined Gating Models,会使用本地下载好的,并提示可以更新:
> scGate_models_DB <- get_scGateDB()
Using local version of repo scGate_models-master. If you want update it, set option force_update = TRUE
用来分型很方便
可以使用plot_tree函数来可视化某个模型的层次结构(需要ggparty)
install.packages("ggparty")
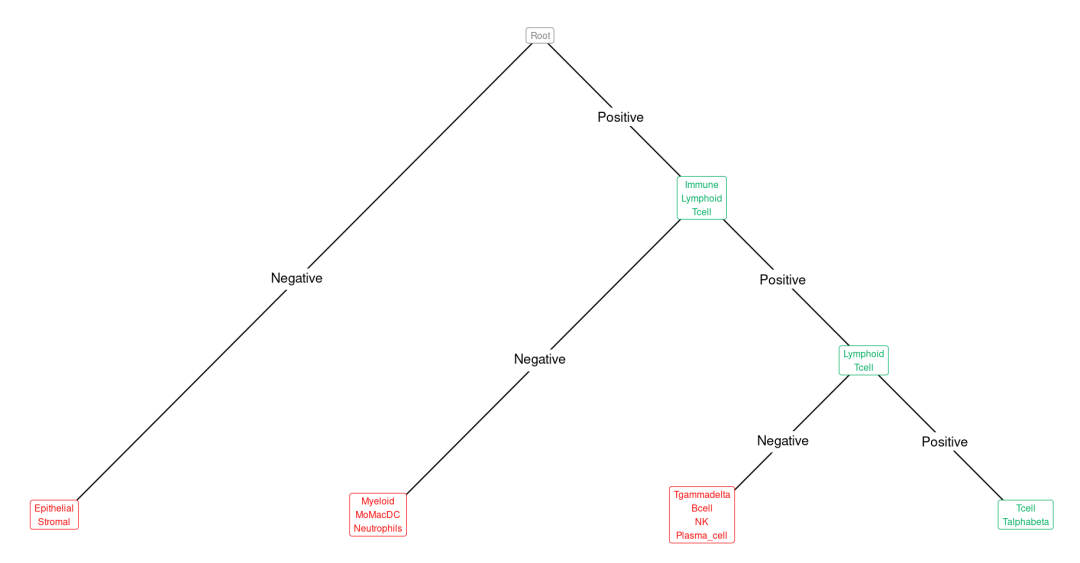
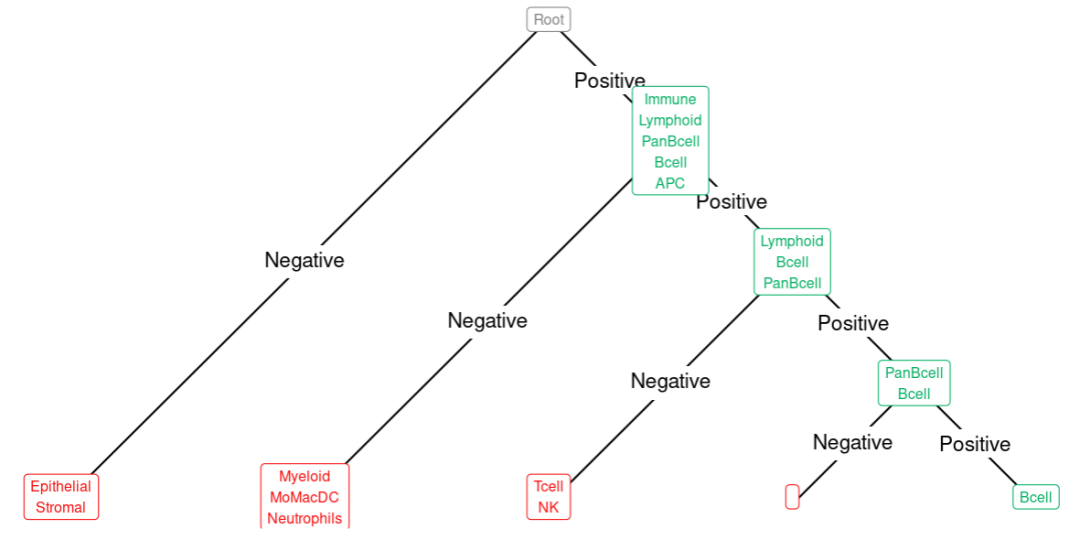
scGate::plot_tree(scGate_models_DB$human$generic$Tcell.alphabeta)
一个 "门控模型"(GM),由一组定义相关细胞群的标记基因组成。基因门控模型可以是简单的单一标记基因,也可以是阳性和阴性标记基因的组合。更复杂的 GM 可以分级方式构建,类似于流式细胞仪中使用的门控策略。
scGate as a multi-class classifier
scGate还可以用作细胞类型分类器,对数据集中的多个细胞类型进行注释。要用基于标记的细胞类型定义注释数据集,只需向scGate提供模型列表,例如:
models.list <- scGate_models_DB$human$generic[c("Bcell","MoMacDC","CD8T","CD4T","Erythrocyte")]
obj <- scGate(obj, model = models.list)
根据我们这个pbmc数据集修改代码:
models.list <- scGate_models_DB$human$generic[c("Bcell","Monocyte","CD4T","CD8T","NK","panDC")]
pbmc <- scGate(pbmc, model = models.list)
Warning: The following genes were not found and will be
imputed to exp=0:
* COL1A1,RAMP2,SFTPB,SLPI,WFDC2,PIGR,SLC34A2,SFTA2,AGR3,ELF3,KRT19,COL1A2,COL3A1,DCN,LUM,THY1,MSR1,TM4SF1,VWF,EPAS1,SPARCL1,CLDN18,APOE,APOC1,KIT,CPA3,TPSAB1,TPSB2,MS4A2,CLEC9A,XCR1,RAB7B,CCL19,CCL22,TRAC,TRBC1,TRBC2,TRDC,TRGC1,TRGC2,TRDV1,TRDV2,HBB,HBA2Warning: pseudoinverse used at -2.2368Warning: neighborhood radius 0.30103Warning: reciprocal condition number 1.9598e-16
### Detected a total of 345 pure 'Bcell' cells (13.08% of total)
### Detected a total of 652 pure 'Monocyte' cells (24.72% of total)
### Detected a total of 1027 pure 'CD4T' cells (38.93% of total)
### Detected a total of 438 pure 'CD8T' cells (16.60% of total)
### Detected a total of 142 pure 'NK' cells (5.38% of total)
### Detected a total of 12 pure 'panDC' cells (0.45% of total)
> table(pbmc@meta.data$scGate_multi)
Bcell CD4T CD8T Monocyte Multi NK
343 1025 434 652 6 138
> DimPlot(pbmc, group.by = "scGate_multi")
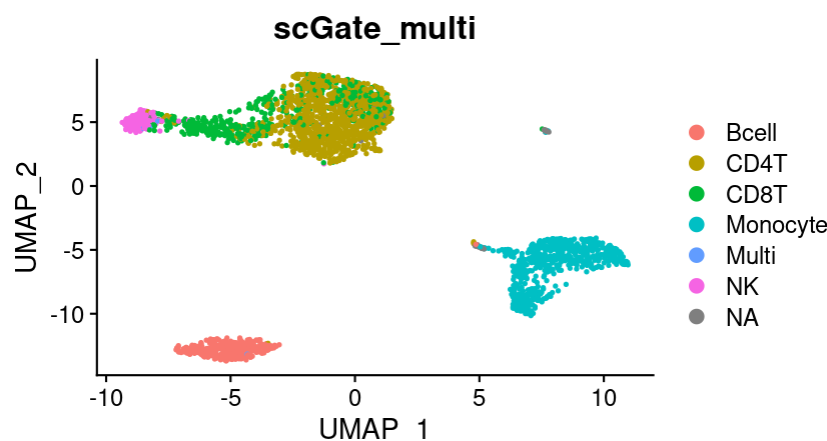
大致分类和我们前面手动选择markers注释一致
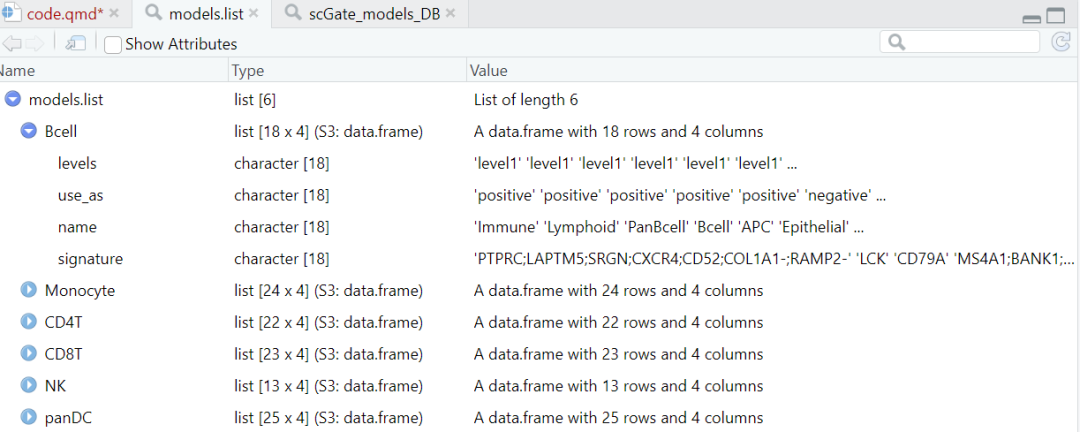
> models.list$Bcell
levels use_as name signature
1 level1 positive Immune PTPRC;LAPTM5;SRGN;CXCR4;CD52;COL1A1-;RAMP2-
2 level1 positive Lymphoid LCK
3 level1 positive PanBcell CD79A
4 level1 positive Bcell MS4A1;BANK1;PAX5;CD19
5 level1 positive APC HLA-DRA;HLA-DMB;HLA-DMA;HLA-DQA1;HLA-DQB1;HLA-DRB1;HLA-DPA1;HLA-DPB1
6 level1 negative Epithelial SFTPB;SLPI;WFDC2;PIGR;SLC34A2;SFTA2;AGR3;ELF3;KRT18;KRT19;KRT8
7 level1 negative Stromal COL1A1;COL1A2;COL3A1;DCN;LUM;ACTA2;THY1;KRT18-;KRT19-;KRT8-
8 level2 positive Lymphoid LCK
9 level2 positive Bcell MS4A1;BANK1;PAX5;CD19
10 level2 positive PanBcell CD79A
11 level2 negative Myeloid SPI1;CD79A-;CD19-
12 level2 negative MoMacDC LYZ;CSF1R;MSR1;MAFB;CD300E;ITGAX;CD68
13 level2 negative Neutrophils CSF3R;FCGR3B;ANXA2-
14 level3 positive PanBcell CD79A
15 level3 positive Bcell MS4A1;BANK1;PAX5;CD19
16 level3 negative Tcell CD3D;CD3E;CD3G;CD2
17 level3 negative NK KLRD1;NKG7;NCR1;FCGR3A;CD3D-;CD3E-;CD3G-;CD8A-;CD8B-
18 level4 positive Bcell MS4A1;BANK1;PAX5;CD19
一个 "门控模型"(GM),由一组定义相关细胞群的标记基因组成。基因门控模型可以是简单的单一标记基因,也可以是阳性和阴性标记基因的组合。更复杂的 GM 可以分级方式构建,类似于流式细胞仪中使用的门控策略。
我们也可以参考这个格式自定义一些model,利用这个框架,可以避免手动注释带来的误差和耗时
You may manually edit the available models (eg in Excel) or create new models for your cell type of interest. You can then load your custom model into R using:
my_scGate_model <- load_scGate_model("path_to_my.model")
Other single-cell modalities

singleR
https://github.com/dviraran/SingleR

> BiocManager::install("singleR")
'getOption("repos")' replaces Bioconductor standard repositories, see 'help("repositories", package =
"BiocManager")' for details.
Replacement repositories:
CRAN: https://mirrors.tuna.tsinghua.edu.cn/CRAN/
Bioconductor version 3.16 (BiocManager 1.30.21.1), R 4.2.2 (2022-10-31)
Installing package(s) 'singleR'
Warning: package ‘singleR’ is not available for Bioconductor version '3.16'

https://bioconductor.org/packages/3.16/bioc/html/SingleR.html
https://bioconductor.org/packages/3.16/bioc/src/contrib/SingleR_2.0.0.tar.gz
> install.packages("SingleR_2.0.0.tar.gz",repos = NULL)
Installing package into ‘/home/data/t120455/R/x86_64-pc-linux-gnu-library/4.2’
(as ‘lib’ is unspecified)
ERROR: dependencies ‘BiocSingular’, ‘beachmat’ are not available for package ‘SingleR’
* removing ‘/home/data/t120455/R/x86_64-pc-linux-gnu-library/4.2/SingleR’
Warning in install.packages :
installation of package ‘SingleR_2.0.0.tar.gz’ had non-zero exit status
安装依赖后
install.packages("SingleR_2.0.0.tar.gz",repos = NULL)
SingleR - Single-cell Recognition
单细胞RNA-seq(scRNA-seq)的最新进展使表征疾病模型中基因表达变化的粒度达到了前所未有的水平。已经开发了多种单细胞分析方法来检测基因表达变化并通过基因表达的相似性对细胞进行聚类。然而,按细胞类型对聚类的分类在很大程度上依赖于已知的标记基因,并且聚类的注释是手动进行的。这种策略具有主观性,并限制了密切相关的细胞亚群的充分分化。 在这里,我们提出了SingleR,一种新的无偏细胞类型识别scRNA-seq的计算方法。SingleR利用纯细胞类型的参考转录组数据集来独立推断每个单细胞的起源细胞。SingleR的注释与Seurat(一种为scRNA-seq设计的处理和分析包)相结合,为scRNA-seq数据的研究提供了强大的工具。我们开发了一个R包来生成带注释的scRNA-seq对象,然后可以使用SingleR web工具对数据进行可视化和进一步分析——http://comphealth.ucsf.edu/SingleR.
使用参考:
https://github.com/dviraran/SingleR#usage https://bioconductor.org/packages/3.16/bioc/manuals/SingleR/man/SingleR.pdf 基于SingleR和scmap两种算法的单细胞自动化注释示例演练
获取celldex参考数据集
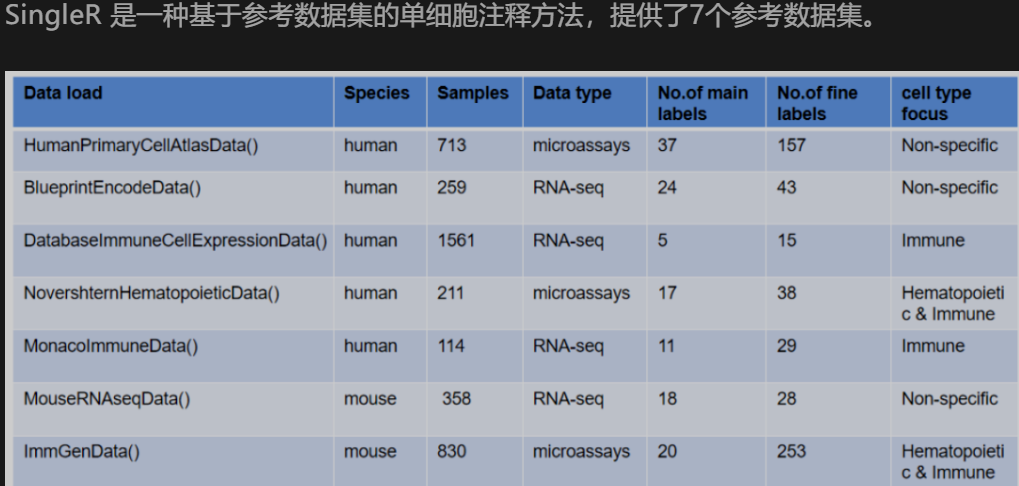
这些参考数据集集成到一个R包中,即celldex
# install.packages("SingleR_2.0.0.tar.gz",repos = NULL)
rm(list=ls())
load("pbmc.Rdata")
library(Seurat)
library(tidyverse)
library(patchwork)
library(SingleR)
pbmc
BiocManager::install("celldex")
第一次使用会提示创建一个cache文件夹,保存到本地
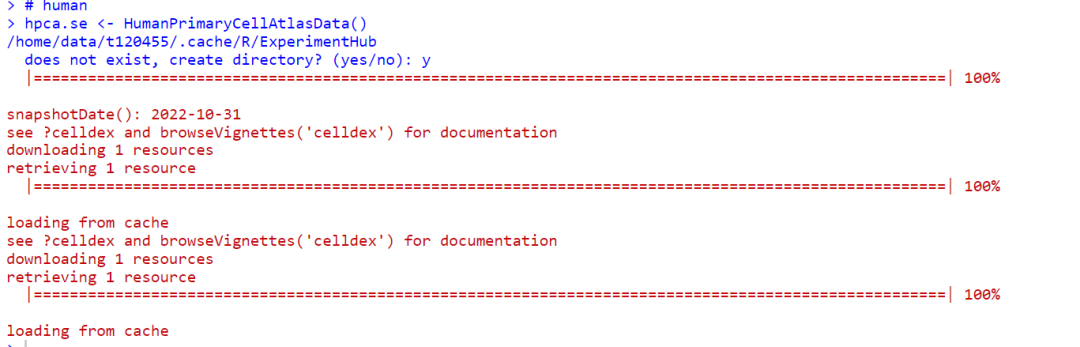
library(celldex)
# human
hpca.se <- HumanPrimaryCellAtlasData()
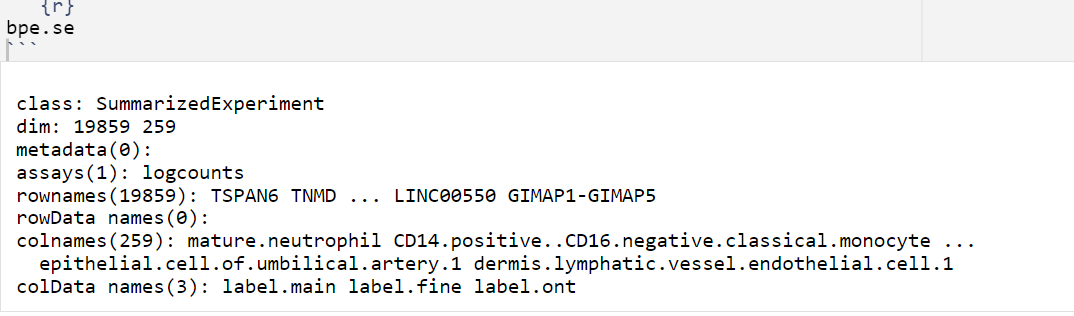
bpe.se <- BlueprintEncodeData()
DICE <- DatabaseImmuneCellExpressionData()
NHD <- NovershternHematopoieticData()
MID <- MonacoImmuneData()
# mouse
MRD <- MouseRNAseqData()
IGD <- ImmGenData()
加载单细胞数据
# 将Seurat对象,转换为 SingleCell 对象,一种 SingleR 可接受的输入文件
test <- as.SingleCellExperiment(pbmc)
细胞注释
我们这里前面也进行过了这样的步骤
基于单个参考集的注释:
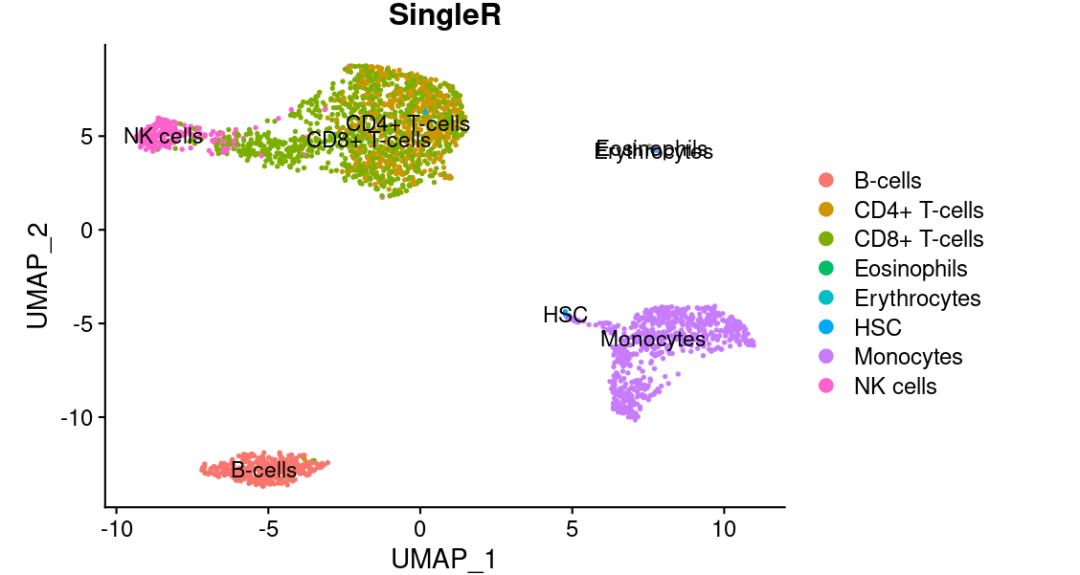
# 基于单个参考集的注释
Anno <- SingleR(test = test,
ref = bpe.se,
labels = bpe.se$label.main
)
pbmc@meta.data$SingleR <- Anno$labels
DimPlot(pbmc, group.by = "SingleR", reduction = "umap", label = TRUE)
img
基于多个参考集的注释:
# 基于多个参考集的注释
Anno2 <- SingleR(test = test,
ref = list(HP = hpca.se , BP = bpe.se),
labels = list(hpca.se$label.main , bpe.se$label.main))
pbmc@meta.data$SingleR2 <- Anno2$labels
DimPlot(pbmc, group.by = "SingleR2", reduction = "umap", label = TRUE)
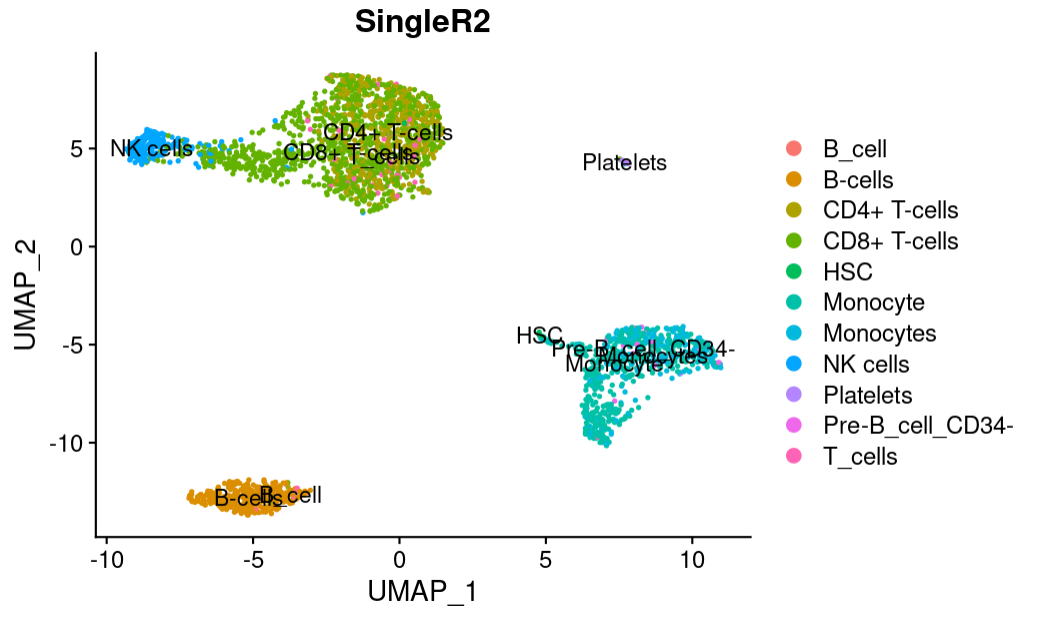
可以发现不同注释数据集中冗余的细胞类型
多个参考数据集的注释,注释结果中会有冲突,需要专业的知识的判断取舍;多个数据集的细胞命名有些许的差异,可以手动更改后再进行展示。
我感觉这个比scGate方便的点在于可以自行注释可能的细胞类型,不需要自己先手动地选择细胞类型来注释,当然别人给的不一定是适合的
SingleR的另一个优势是也可以自定义一个参考数据集,因为参考数据集的质量或者参考数据集的适用性也是影响注释好坏的一个因素。如果能够找到更好的参考数据集可以自定义,运用SingleR软件进行注释。
自定义参考集:
SingleR如何使用自定义的参考集
scmap
https://github.com/hemberg-lab/scmap
scmap-一种无监督投影单细胞RNA-seq数据的工具 单细胞RNA-seq(scRNA-seq)被广泛用于研究复杂组织的组成,因为该技术允许研究人员使用转录组的无监督聚类来定义细胞类型。然而,由于实验方法和计算分析的差异,直接比较两个不同实验中鉴定的细胞往往具有挑战性。在这里,我们介绍了scmap,这是一种将scRNA-seq实验中的细胞投射到不同实验中鉴定的细胞类型上的方法。
也是biocmanager安装
使用参考:
https://bioconductor.org/packages/3.16/bioc/manuals/scmap/man/scmap.pdf 基于SingleR和scmap两种算法的单细胞自动化注释示例演练
rm(list=ls())
load("pbmc.Rdata")
library(scmap)
library(celldex)
library(scater)
library(SingleCellExperiment)
# 待查询数据集,转化为 SingleCellExperiment 格式
# 前面已经进行(看看seurat对象的data和counts就知道了)
# 这里将省略标准化过程
pbmcAnno <- as.SingleCellExperiment(pbmc)
https://bioconductor.org/packages/3.16/bioc/src/contrib/scater_1.26.1.tar.gz
install.packages("scater_1.26.1.tar.gz",repos=NULL)
定义参考基因数据集文件
Scmap软件接受“SingleCellExperiment”对象文件。 在制定参考基因数据集文件时,默认的基因名称位于“feature_symbol”列,细胞类型位于“cell_type1”列。 Scmap要求参考数据集是进行归一化和对数转化的(LogNormalize)。
# 先下载 SingleR 的参考数据集文件
refData <- celldex::DatabaseImmuneCellExpressionData()
参考前面singleR celldex参考数据集
> refData <- celldex::DatabaseImmuneCellExpressionData()
snapshotDate(): 2022-10-31
see ?celldex and browseVignettes('celldex') for documentation
loading from cache
Error: failed to load resource
name: EH3488
title: DICE RNA-seq logcounts
reason: error reading from connection
怀疑是上次下载终端导致的,清除缓存文件:
DatabaseImmuneCellExpressionData这个数据集下载很慢,晚上感觉更慢,这里还是用前面的 HumanPrimaryCellAtlasData进行案例测试
# 先下载 SingleR 的参考数据集文件
# refData <- celldex::DatabaseImmuneCellExpressionData()
refData <- HumanPrimaryCellAtlasData()
refData
class: SummarizedExperiment
dim: 19363 713
metadata(0):
assays(1): logcounts
rownames(19363): A1BG A1BG-AS1 ... ZZEF1 ZZZ3
rowData names(0):
colnames(713): GSM112490 GSM112491 ... GSM92233
GSM92234
colData names(3): label.main label.fine label.ont
> colData(refData)
DataFrame with 713 rows and 3 columns
label.main label.fine label.ont
<character> <character> <character>
GSM112490 DC DC:monocyte-derived:.. CL:0000840
GSM112491 DC DC:monocyte-derived:.. CL:0000840
GSM112540 DC DC:monocyte-derived:.. CL:0000840
GSM112541 DC DC:monocyte-derived:.. CL:0000451
GSM112661 DC DC:monocyte-derived:.. CL:0000451
... ... ... ...
GSM556665 Monocyte Monocyte:S._typhimur.. CL:0000576
GSM92231 Neurons Neurons:Schwann_cell CL:0002573
GSM92232 Neurons Neurons:Schwann_cell CL:0002573
GSM92233 Neurons Neurons:Schwann_cell CL:0002573
GSM92234 Neurons Neurons:Schwann_cell CL:0002573
> rowData(refData)
DataFrame with 19363 rows and 0 columns
创建SingleCellExperiment对象:
# 定义 cell-type1 列
colData(refData)$cell_type1 <- colData(refData)$label.main
# 定义 feature_symbol 列
rowData(refData)$feature_symbol <- rownames(refData)
# 创建 scmap 参考数据集文件
refSCE <- SingleCellExperiment(
assays = list(
logcounts=Matrix::Matrix(assays(refData)$logcounts)
),
colData=colData(refData),
rowData=rowData(refData)
)
构建细胞索引
这一步是加快注释的步骤。构建索引使用的是高可变基因(含有更多的分类信息,去除混杂的影响因素)。对于筛选的基因的数量,可以通过参数“n_features”来指定,默认是500个基因。
refSCE <- scmap::selectFeatures(refSCE, suppress_plot = FALSE)
Error in lm.fit(x, y, offset = offset, singular.ok = singular.ok, ...) :
0 (non-NA) cases
In addition: Warning message:
In linearModel(object, n_features) :
Your object does not contain counts() slot. Dropouts were calculated using logcounts() slot...
出现问题
Error in lm.fit(x, y, offset = offset, singular.ok = singular.ok, ...) :
0 (non-NA) cases
参考: error in selectFeatures(https://github.com/hemberg-lab/scmap/issues/29)
类似的包还有MACA,只不过是基于python的scanpy框架的
https://github.com/ImXman/MACA
总的来说可以使用多种方法、多个参考cellmarkers数据集来划分细胞亚型,并可视化gplots::balloonplot()列联表,并且最好有较强的专业知识
本文参与 腾讯云自媒体同步曝光计划,分享自微信公众号。
原始发表:2023-09-19 12:30,如有侵权请联系 cloudcommunity@tencent.com 删除
评论
登录后参与评论
推荐阅读
目录

