单细胞转录和染色质可及性分析重新定义成人肾脏的细胞异质性
单细胞转录和染色质可及性分析重新定义成人肾脏的细胞异质性
生信技能树jimmy
发布于 2023-11-06 17:51:36
发布于 2023-11-06 17:51:36
文章概述
文章标题:《Single cell transcriptional and chromatin accessibility profiling redefine cellular heterogeneity in the adult human kidney》
发表日期和杂志:2021年发表在Nature Communications上
在线阅读链接:https://doi.org/10.1038/s41467-021-22368-w
实验设计
进行了单核ATAC(SnATAC-seq)和RNA(snRNA-seq)测序,以生成成对的、特定细胞类型的染色质可及性和成人肾脏的转录图谱。对5例健康成人肾脏标本进行了SNRNA-SEQ和SnATAC-SEQ检查.
单细胞转录组数据情况
数据链接是:https://www.ncbi.nlm.nih.gov/geo/query/acc.cgi?acc=GSE151302 Sequencing data are deposited in GEO under accession number GSE151302. Previously published snRNA-seq data for three adult kidneys are available in GEO (GSE131882).
可以看到单细胞RNA测序数据一共是5个数据集
GSM4572192 Healthy1_snRNAseq
GSM4572193 Healthy2_snRNAseq
GSM4572194 Healthy3_snRNAseq
GSM4572195 Healthy4_snRNAseq
GSM4572196 Healthy5_snRNAseq
提供了filtered_feature_bc_matrix.h5格式的单细胞数据h5格式文件,直接下载之后使用Read10X_h5函数读取
数据下载导入,并创建Seurat对象:
###### step1:导入数据 ######
library(data.table)
dir='GSE151302_RAW/'
samples=list.files( dir )
samples
library(data.table)
sceList = lapply(samples,function(pro){
# pro=samples[1]
print(pro)
sce=CreateSeuratObject( Read10X_h5(file.path(dir,pro)),
project = strsplit(pro,'_')[[1]][2],
min.cells = 5,
min.features = 300 )
return(sce)
})
names(sceList)
samples
library(stringr)
sce.all=merge(x=sceList[[1]],
y=sceList[ -1 ],
add.cell.ids = str_split(samples,'_',simplify = T)[,2])
as.data.frame(sce.all@assays$RNA@counts[1:10, 1:2])
head(sce.all@meta.data, 10)
table(sce.all$orig.ident)
循环读取小Tips:在循环读取之前,可以先使用pro=samples[1]读取进一个数据集测试一下代码,可以的话就进行后续的循环读取。
后面就是标准分析啦,对读取进来的数据进行质控、harmony整合以及细分亚群定义等。
第一层次降维聚类分群
文章中综合数据snRNA-seq基于谱系特异性标记的表达鉴定了肾皮质内的所有主要细胞类型:
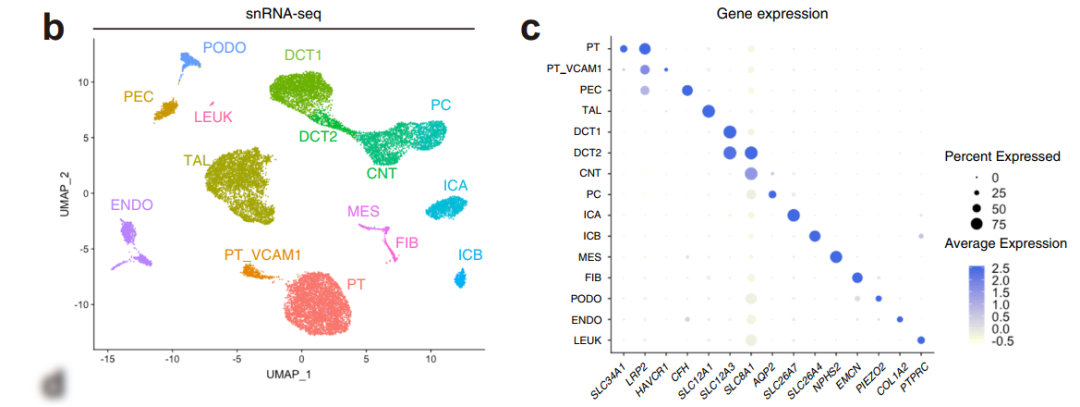
- proximal tubule (PT)
- parietal epithelial cells (PEC)
- thick ascending limb (TAL)
- distal tubule (DCT1,DCT2)
- connecting tubule (CNT)
- collecting duct (PC,ICA,ICB)
- endothelial cells (ENDO)
- glomerular cell types (MES,PODO)
- fibroblasts (FIB)
- a small population of leukocytes (LEUK)
通过BioinfoArk提供的中国区chatGPT查询到各个细分亚群的解释:
- 近曲小管 (PT):这是肾小管的一部分,负责从原尿中重吸收水分和电解质。常见的近曲小管标记基因有SLC3A1和SLC22A6。
- 壁细胞 (PEC):这些细胞位于肾小球的Bowman囊壁上,参与肾小球的结构维持和修复。常见的壁细胞标记基因有CLDN1。
- 粗升支 (TAL):这是肾单位的一部分,负责从尿液中重吸收水分和电解质。常见的粗升支标记基因有SLC12A1。
- 远曲小管 (DCT1,DCT2):这是肾小管的一部分,负责从尿液中重吸收水分和电解质。常见的远曲小管标记基因有SLC12A3。
- 连接小管 (CNT):这是肾小管的一部分,负责从尿液中重吸收水分和电解质。常见的连接小管标记基因有CLDN8。
- 收集管 (PC,ICA,ICB):这是肾小管的末端部分,负责从尿液中重吸收水分和电解质,以及调节尿液的酸碱度。常见的收集管标记基因有AQP2。
- 内皮细胞 (ENDO):内皮细胞构成血管和淋巴管的内层。内皮细胞的标记基因有CD31(PECAM1)。
- 肾小球细胞类型 (MES,PODO):肾小球是肾脏的过滤单位,由肾小球系膜细胞(MES)和足细胞(PODO)等多种细胞类型构成。常见的肾小球细胞标记基因有WT1(肾小球系膜细胞)和NPHS1(足细胞)。
- 成纤维细胞 (FIB):成纤维细胞是结缔组织的主要细胞类型,负责产生细胞外基质。成纤维细胞的标记基因有ACTA2(肌动蛋白)。
- 少量的白细胞 (LEUK):白细胞是免疫系统的重要组成部分,负责对抗感染和疾病。白细胞的标记基因有CD45(PTPRC)。
其它加分项
使用97%的置信度阈值对snATAC-seq数据集进行筛选,用于细胞类型分配以去除异型双元。
通过标记转移获得的SNATAC-SEQ细胞类型预测与非监督聚类的精选注释的比较
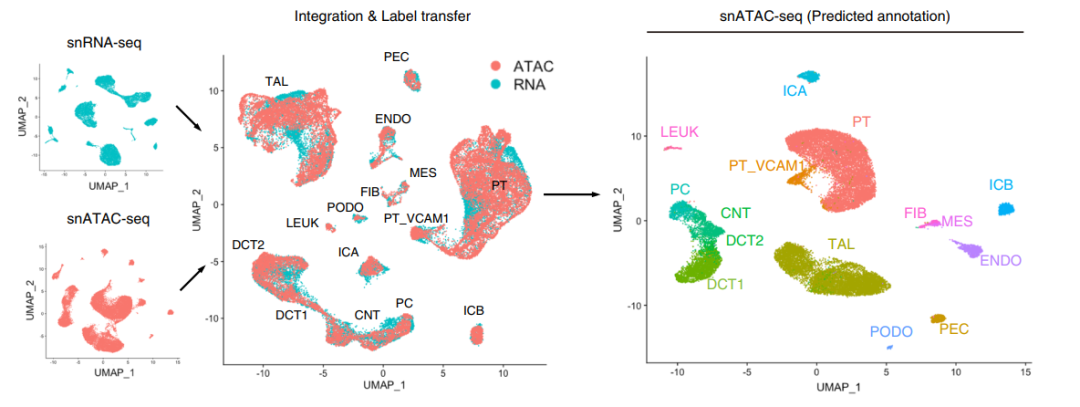
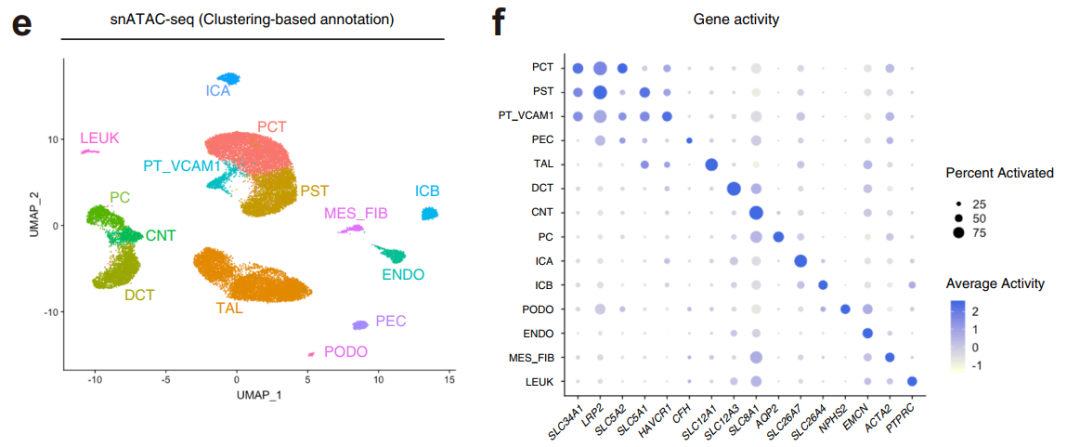
文章同时进行了SnRNA-seq和SnATAC-seq测序,以描述成人肾脏的转录和染色质可及性情况。
分析表明,SNRNA-SEQ和SnATAC-SEQ是确定细胞特性的可比方法,而特定细胞类型的染色质可及性提供了进一步阐明细胞异质性的额外信息。多模式单细胞图谱(“多组学”)极大地提高了检测独特细胞类型和状态的能力。
本文参与 腾讯云自媒体同步曝光计划,分享自微信公众号。
原始发表:2023-11-03,如有侵权请联系 cloudcommunity@tencent.com 删除
评论
登录后参与评论
推荐阅读
目录

